Side Menu
MTOR
基本情報
- 遺伝子名
-
mechanistic target of rapamycin kinase
- 慣用名
-
FRAP, FRAP1, FRAP2, RAFT1, RAPT1, SKS
- 遺伝子分類
-
がん遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
腫瘍形成・増殖
- シグナル伝達経路
-
PI3K/Akt/mTOR
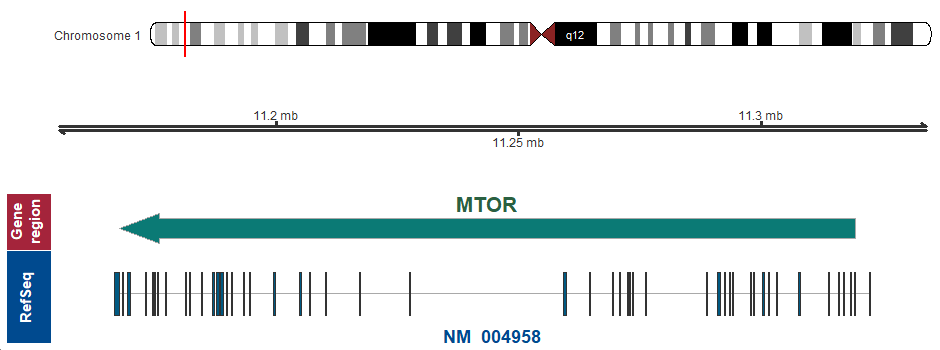
- 染色体上の位置 (GRCh37/hg19)
-
1p36.22 (chr1:11167542..11319466, complement)
- アミノ酸配列の長さ
-
2,549
遺伝子マップ
解説
MTOR (mTOR)はセリン/スレオニンキナーゼであり、代謝、血管新生、細胞増殖および生存を含むさまざまな細胞機能の制御に働くPI3K/Akt/mTORシグナルの主要な構成分子である。mTORは複数のタンパクと複合体を構成して存在しており、生理活性物質ラパマイシンに対して感受性を示すI型複合体 (mTORC1)および非感受性のII型複合体 (mTORC2)の2種類が知られている。複合体を構成するタンパクのうちmTOR, mLST8 (GβL )およびDEPTORは両複合体に共通して含まれている。一方で、RAPTORはmTORC1においてのみ含まれ、Rictor, Sin1およびPROTOR1/2はmTORC2においてのみ含まれる構成タンパクである。mTORC1はPI3K/Akt/mTORシグナルにおいてAKTの下流に位置し、その活性化には低分子量GTP結合蛋白質Rhebの活性化が必要である。AKTの活性化によりRheb抑制因子TSC1/TSC2複合体のうちTSC2の抑制的リン酸化部位である939番目のセリンおよび1462番目のチロシンがリン酸化されることで抑制機能が不活化され、Rhebが機能できる状態となりmTORC1の活性化が誘導される。活性化したmTOR1は下流のp70S6Kおよび4EBP1の活性調節を通して翻訳およびタンパク合成を促進する。また、転写制御因子であるHIF1αの活性化により血管新生に関与するVEGFやグルコースの取り込みに関与するGLUT1の発現を誘導する。そのため、mTORC1の過剰な活性化は細胞増殖や腫瘍の進展を強く亢進させる。このmTORC1を標的とした抗癌薬としてラパマイシンの誘導体 が開発されており、エベロリムスおよびテムシロリムスが承認され臨床で使用されている。一方mTORC2はAKTの473番目のセリン残基をリン酸化することでPDK1と協調してAKTの完全活性化に関与している。
シグナル伝達経路

遺伝子変異総量(Tumor Mutation Burden)の分布TMB

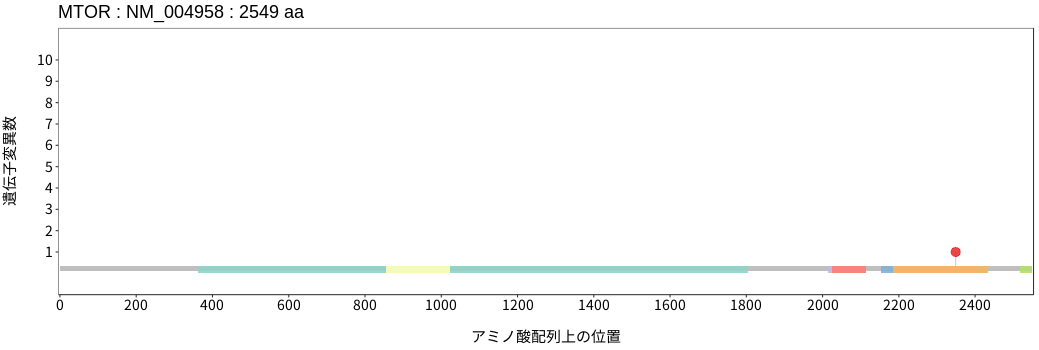
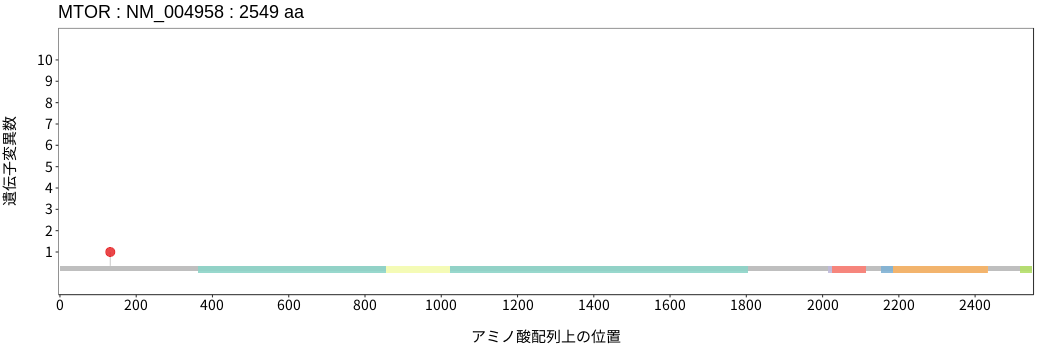
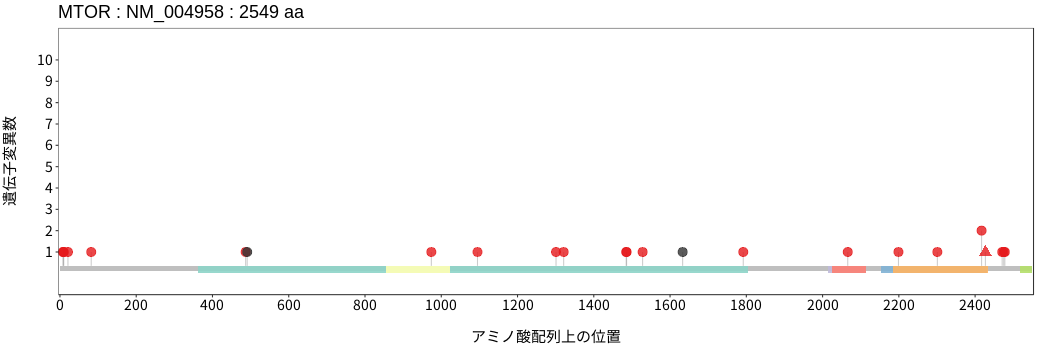
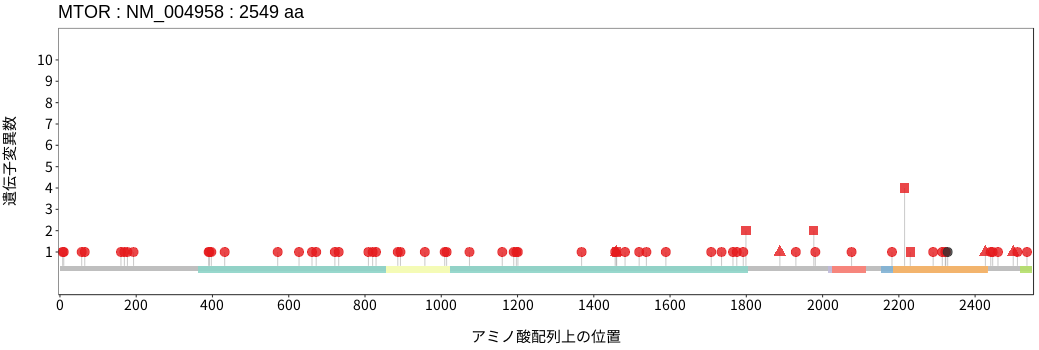
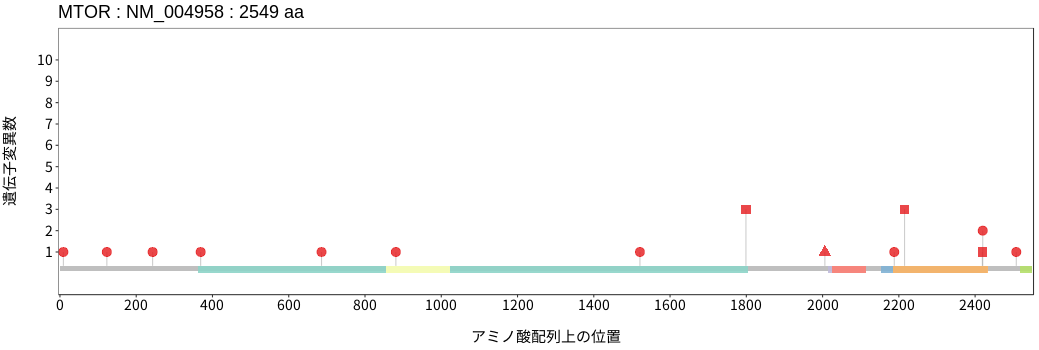
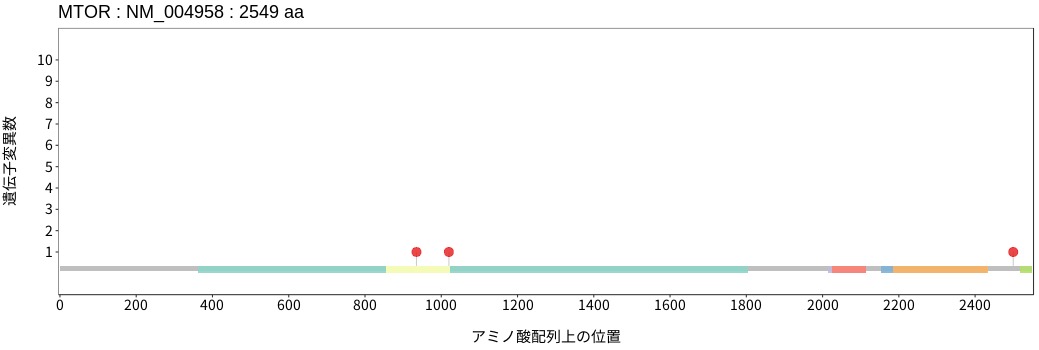
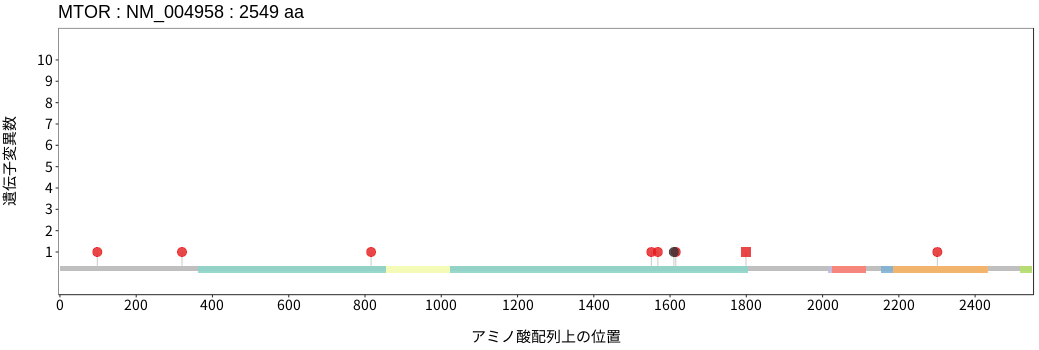
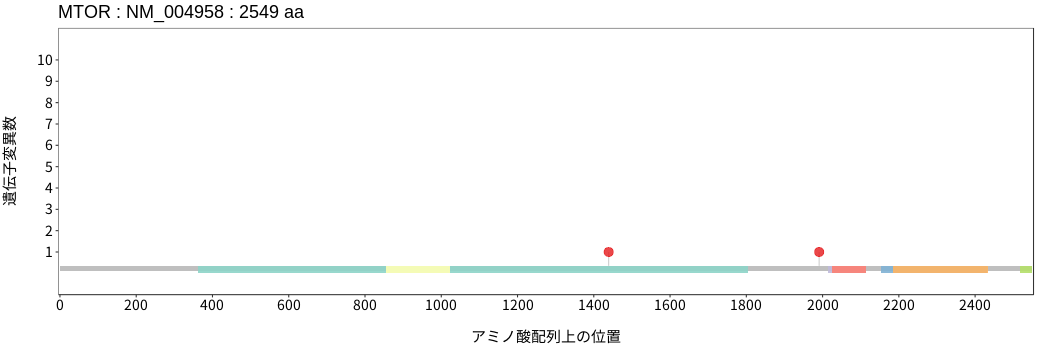
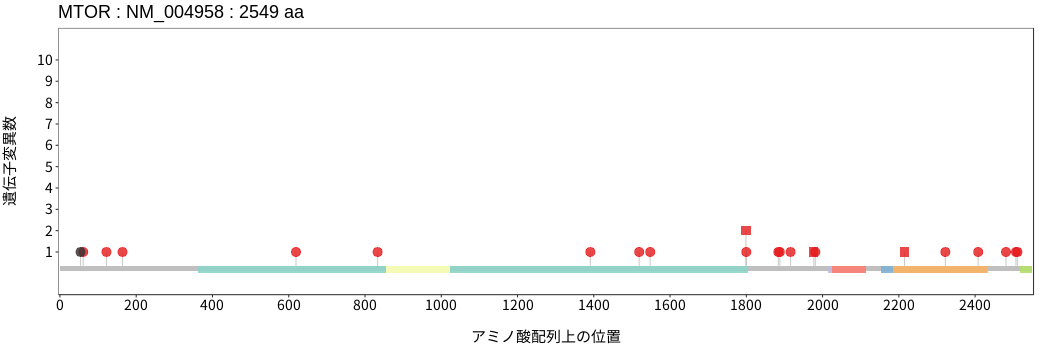
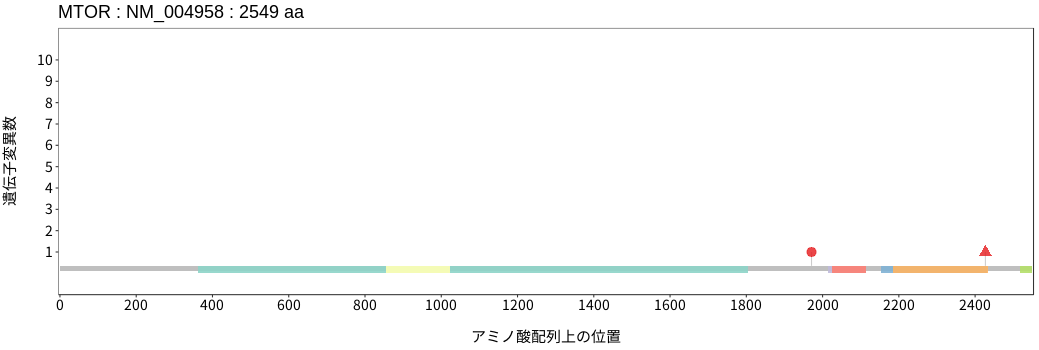
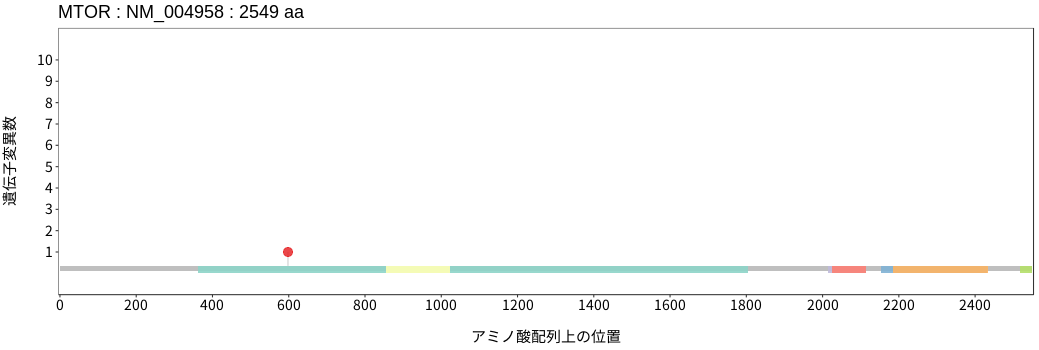
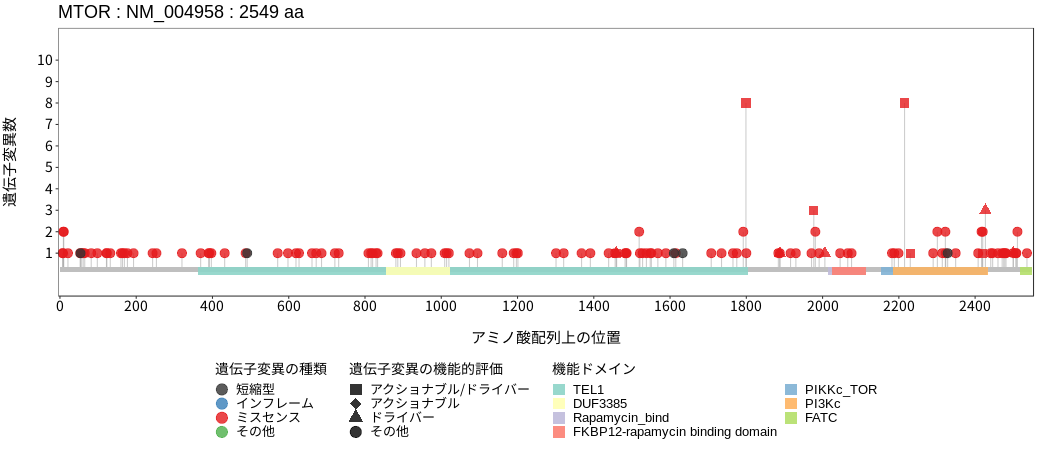
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr1:11217299 | A | G | 30 / 58 | p.L1460P | c.4379T>C | COSV63868845 | Tier1 | 1 / 176 |
| chr1:11217303 | C | G | 30 / 58 | p.A1459P | c.4375G>C | COSV63869424 | Tier2 | 1 / 176 |
| chr1:11190804 | C | T | 39 / 58 | p.E1799K | c.5395G>A | COSV63869964 | Tier2 | 8 / 176 |
| chr1:11189845 | G | T | 40 / 58 | p.F1888L | c.5664C>A | COSV63869154 | Tier2 | 1 / 176 |
| chr1:11188078 | C | A | 43 / 58 | p.V2006F | c.6016G>T | COSV63868121 | Tier2 | 1 / 176 |
| chr1:11188164 | G | A | 43 / 58 | p.T1977I | c.5930C>T | COSV63868784 | Tier1 | 1 / 176 |
| chr1:11188164 | G | T | 43 / 58 | p.T1977K | c.5930C>A | COSV63869673 | Tier1 | 2 / 176 |
| chr1:11184573 | G | A | 47 / 58 | p.S2215F | c.6644C>T | COSV63868278 | Tier1 | 4 / 176 |
| chr1:11184573 | G | T | 47 / 58 | p.S2215Y | c.6644C>A | COSV63868313 | Tier2 | 4 / 176 |
| chr1:11182158 | A | C | 48 / 58 | p.L2230V | c.6688T>G | COSV63875963 | Tier1 | 1 / 176 |
| chr1:11174395 | A | C | 53 / 58 | p.L2427R | c.7280T>G | COSV63869220 | Tier2 | 3 / 176 |
| chr1:11174420 | C | T | 53 / 58 | p.E2419K | c.7255G>A | COSV63869451 | Tier1 | 1 / 176 |
| chr1:11169377 | T | A | 56 / 58 | p.I2500F | c.7498A>T | COSV63869574 | Tier2 | 1 / 176 |