Side Menu
PIK3CA
基本情報
- 遺伝子名
-
phosphatidylinositol-4,5-bisphosphate 3-kinase catalytic subunit alpha
- 慣用名
-
CLAPO, CLOVE, CWS5, MCM, MCMTC, PI3K-alpha, p110-alpha
- 遺伝子分類
-
がん遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
腫瘍形成・増殖
- シグナル伝達経路
-
PI3K/Akt/mTOR
- 染色体上の位置 (GRCh37/hg19)
-
3q26.32 (chr3:178916614..178952152)
- アミノ酸配列の長さ
-
1,068
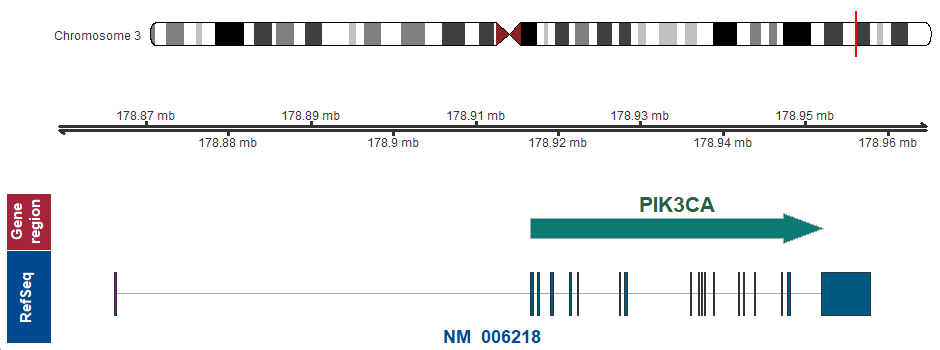
遺伝子マップ
解説
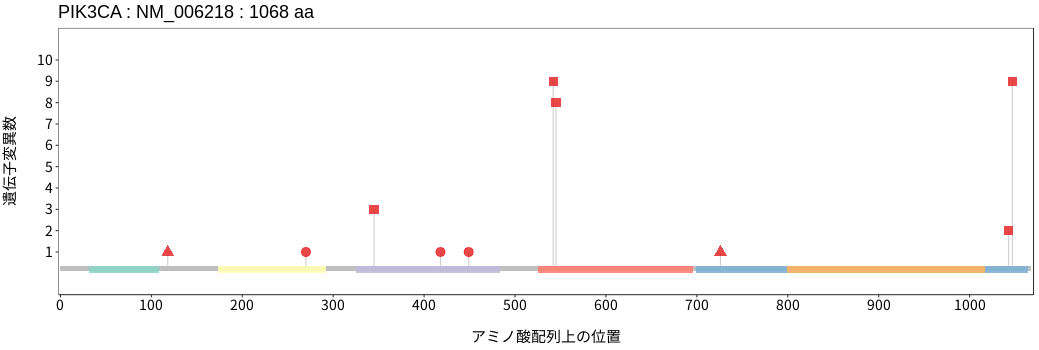
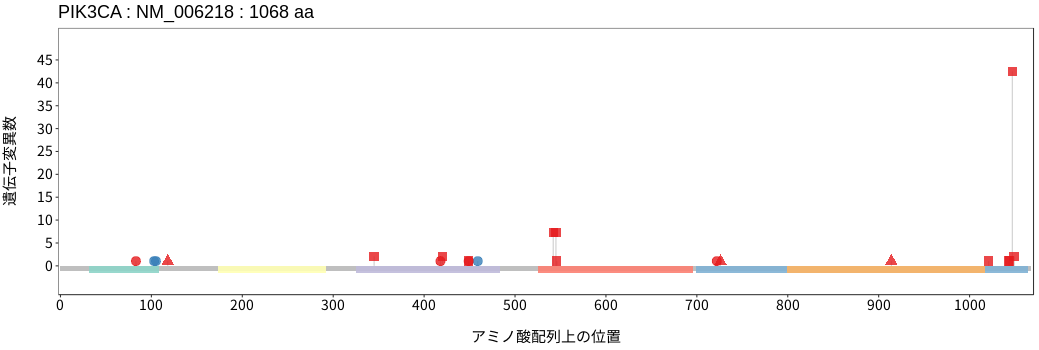
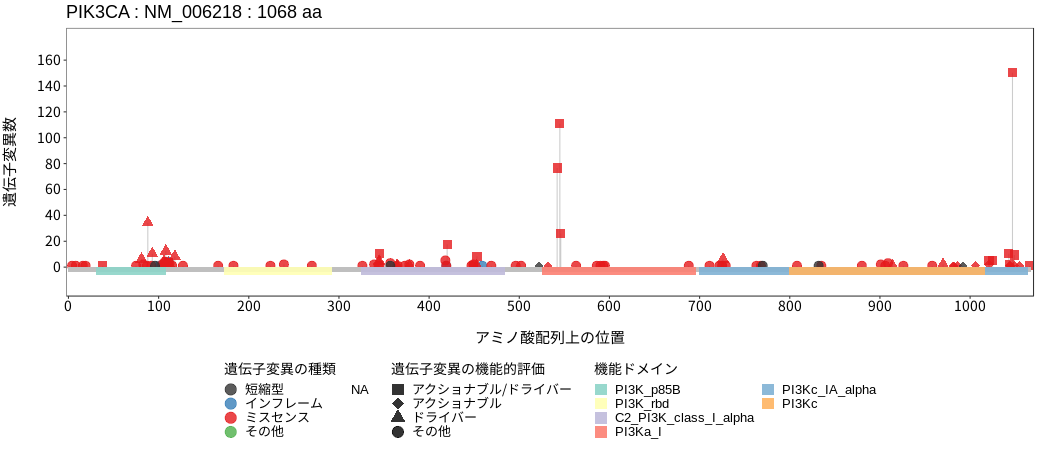
PIK3CAはホスファチジルイノシトール-3キナーゼ (PI3K)の触媒サブユニットp110α (PI3Kα)である。PI3Kは受容体チロシンキナーゼのシグナルを受け活性化したRASにより細胞膜上に動員され、細胞膜に存在するリン脂質であるホスファチジルイノシトール4,5-二リン酸 [PI(4,5)P2]をリン酸化し、ホスファチジルイノシトール3,4,5-三リン酸[PI(3,4,5)P3]を産生する。続いて、PI(3,4,5)P3は細胞膜上にAKTおよびPDK1を動員し、その下流分子を逐次活性化させることでPI3K/Akt/mTORシグナル経路を活性化状態にし、代謝、血管新生、そして細胞増殖を亢進させる。腫瘍におけるPIK3CAの遺伝子変化は多くのがん種において認められており、ホットスポットである542および545番目のアミノ酸 (グルタミン酸)、そして1047番目アミノ酸 (ヒスチジン)におけるバリアントは、PIK3CAのキナーゼ活性を亢進させるドライバーバリアントとして知られている。
シグナル伝達経路

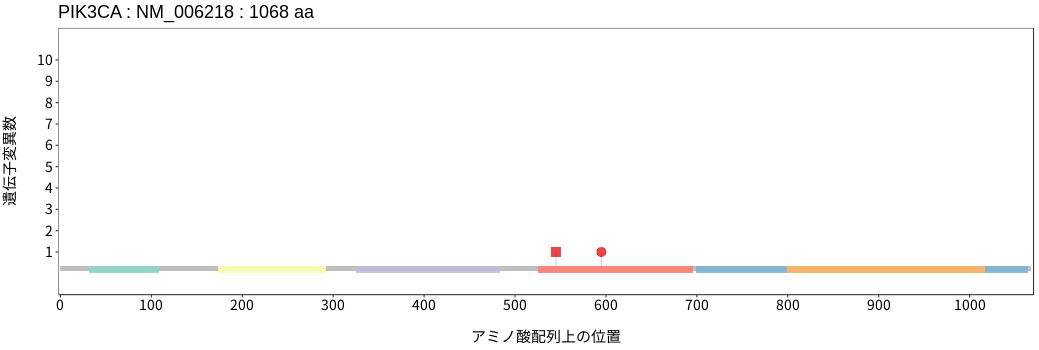
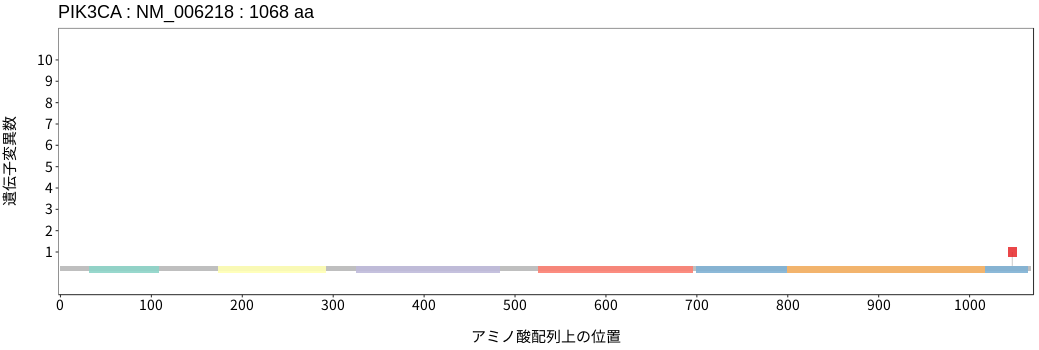
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

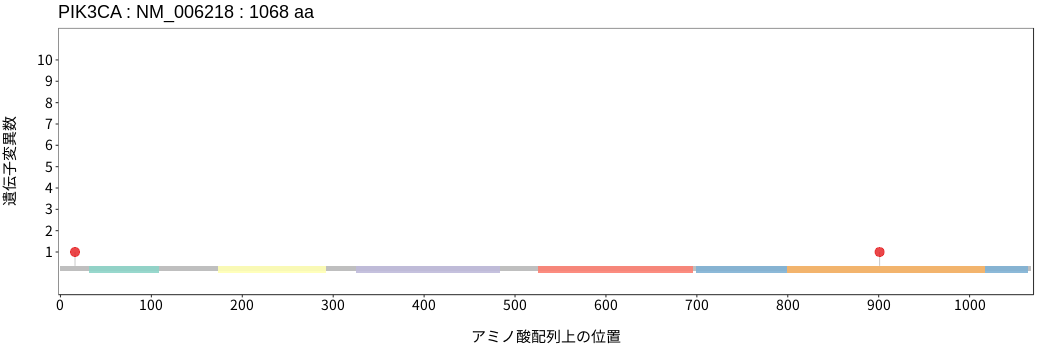
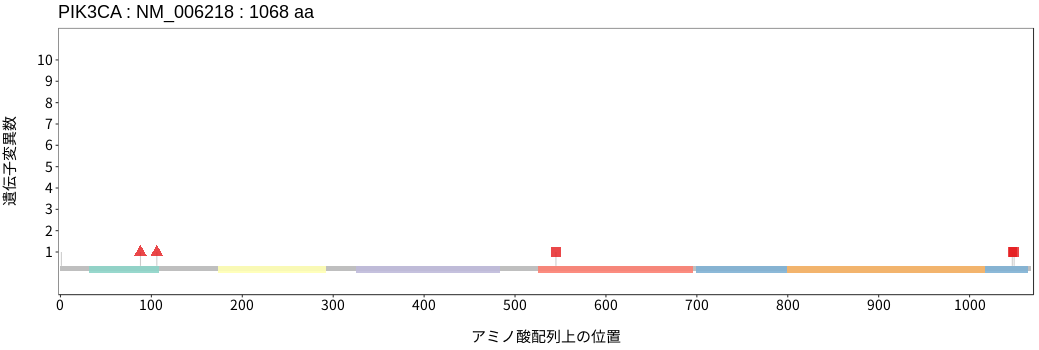
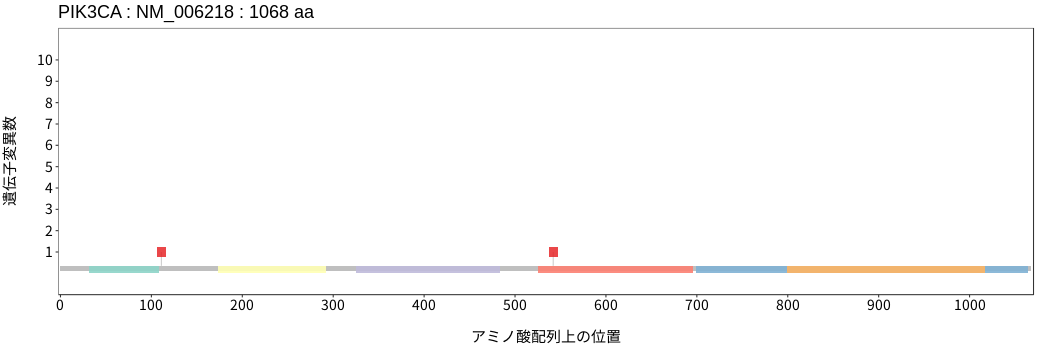
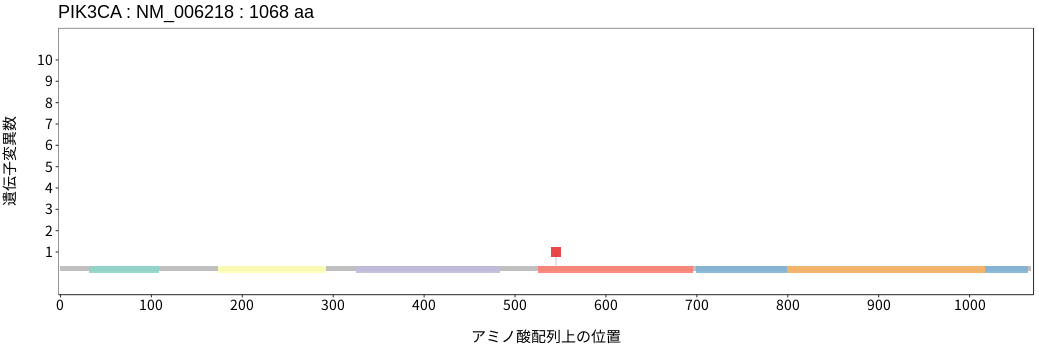
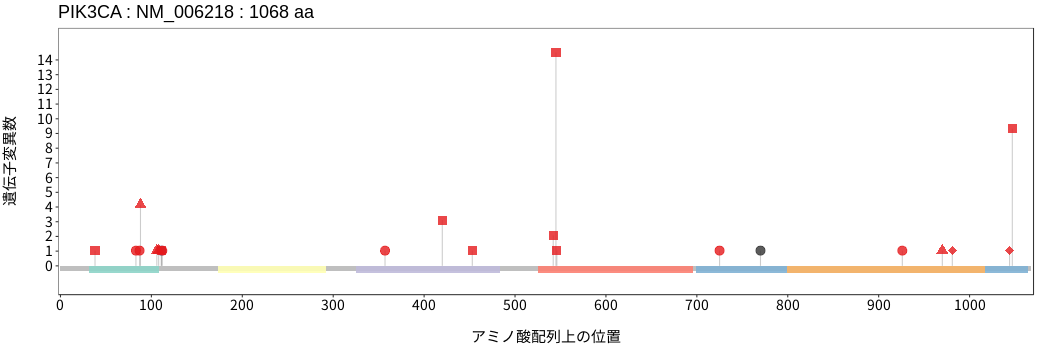
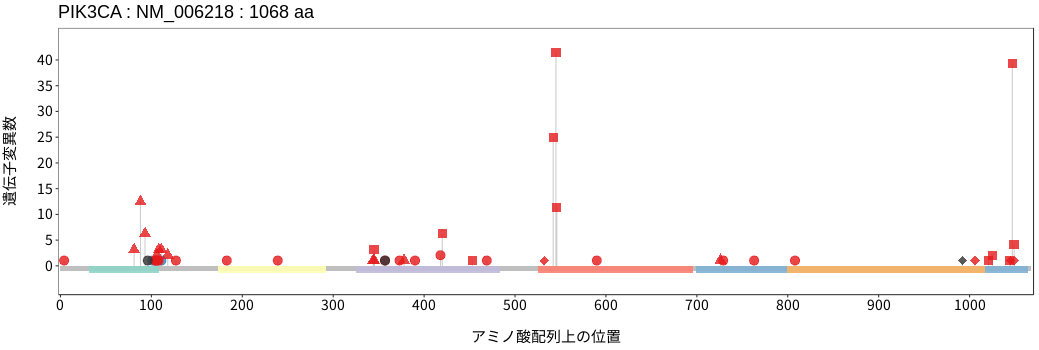
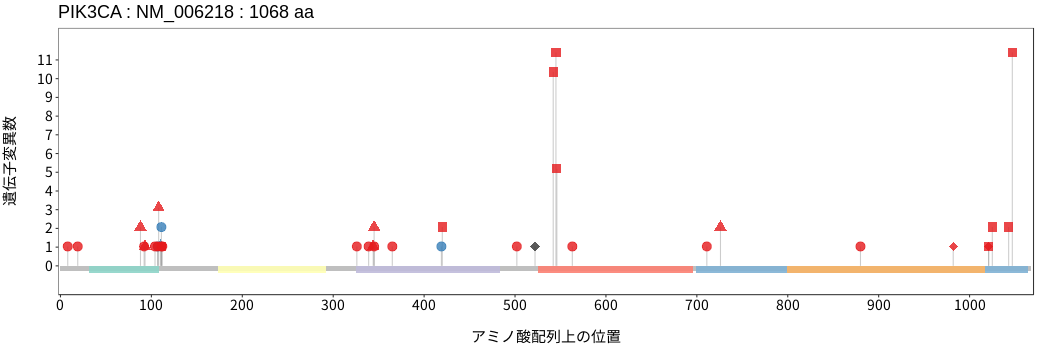
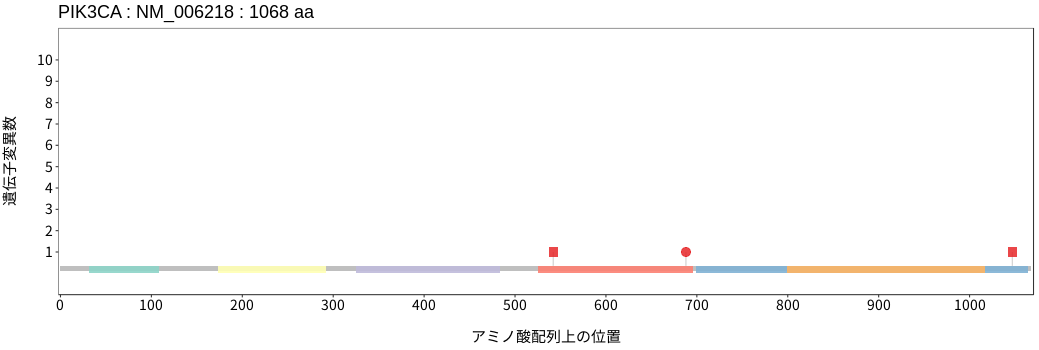
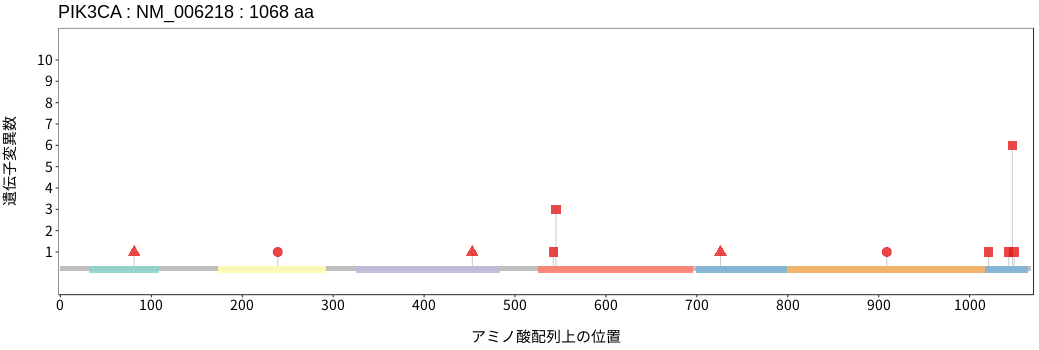
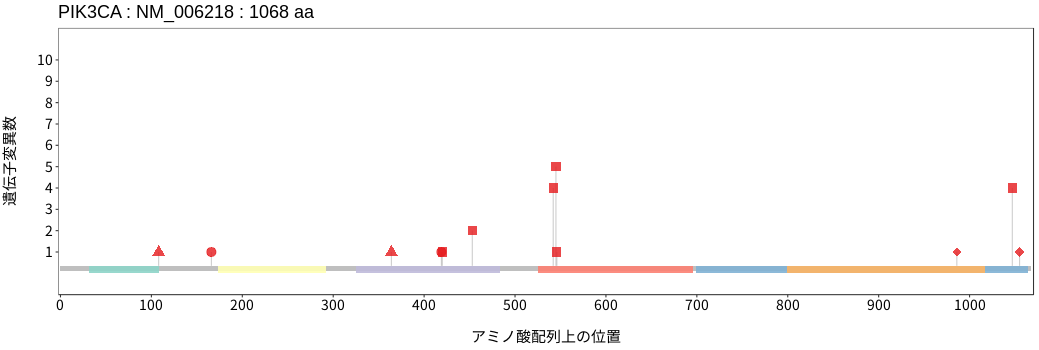
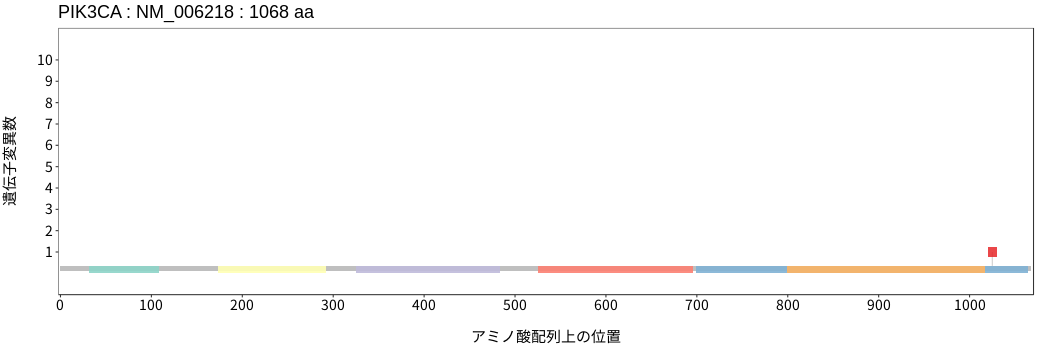
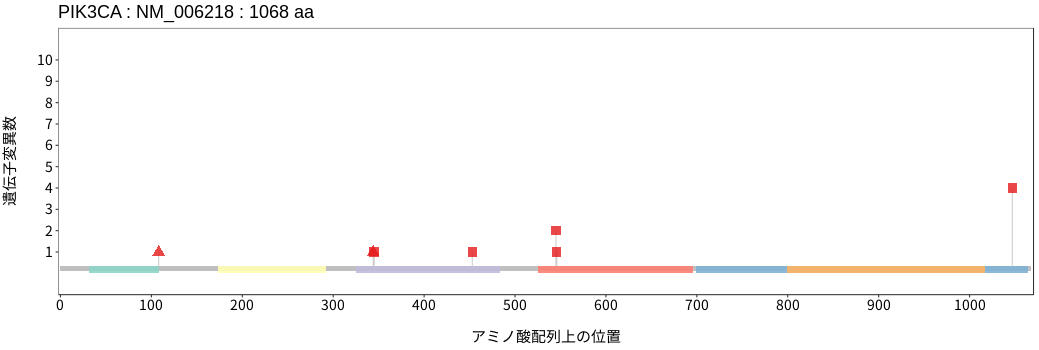
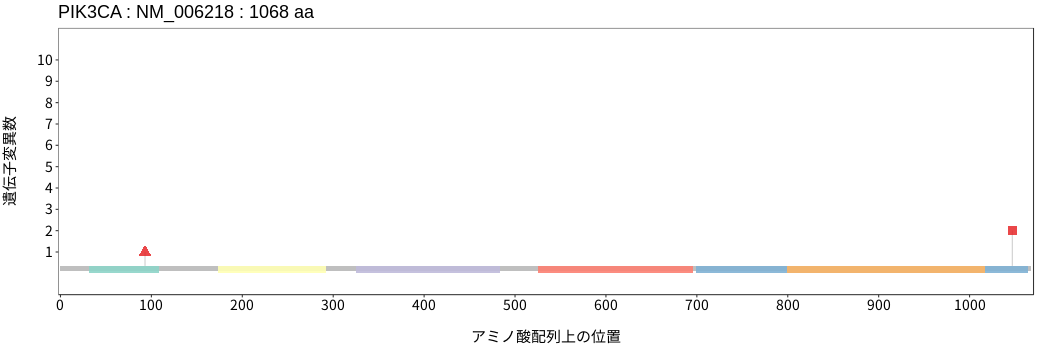
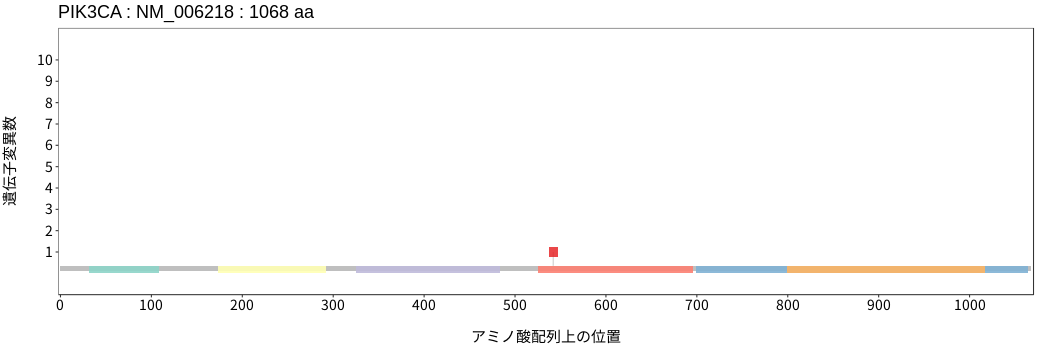
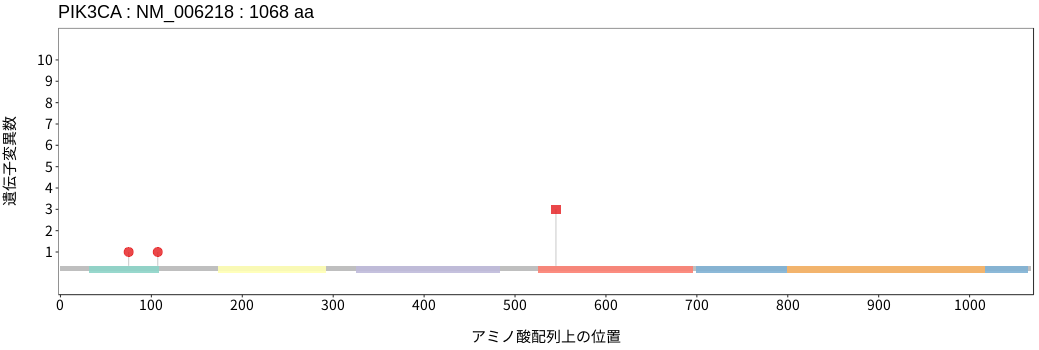
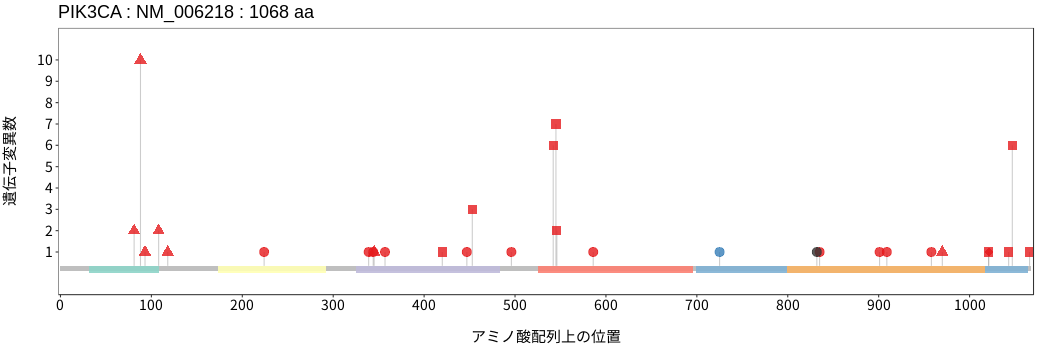
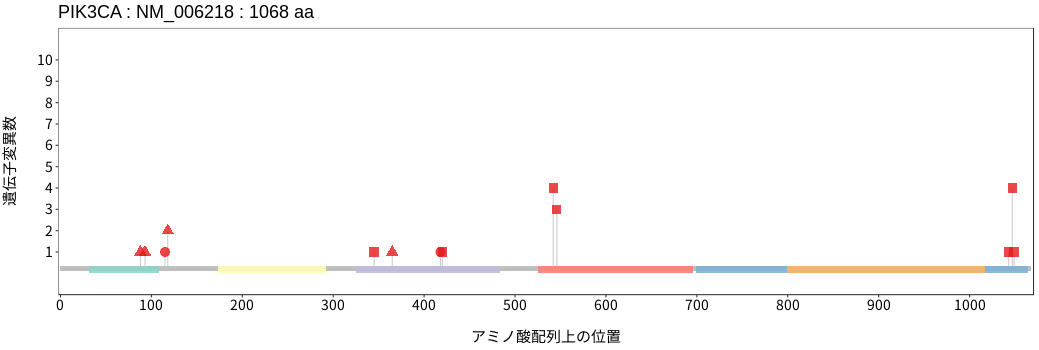
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr3:178916726 | G | A | 2 / 21 | p.R38H | c.113G>A | COSV55879949 | Tier1 | 1 / 560 |
| chr3:178916854 | G | A | 2 / 21 | p.E81K | c.241G>A | COSV55873676 | Tier2 | 6 / 560 |
| chr3:178916876 | G | A | 2 / 21 | p.R88Q | c.263G>A | COSV55874568 | Tier2 | 33 / 560 |
| chr3:178916890 | C | T | 2 / 21 | p.R93W | c.277C>T | COSV55874903 | Tier2 | 7 / 560 |
| chr3:178916891 | G | A | 2 / 21 | p.R93Q | c.278G>A | COSV55877982 | Tier2 | 3 / 560 |
| chr3:178916929 | G | C | 2 / 21 | p.G106R | c.316G>C | COSV55878215 | Tier2 | 1 / 560 |
| chr3:178916930 | G | T | 2 / 21 | p.G106V | c.317G>T | COSV55874291 | Tier2 | 2 / 560 |
| chr3:178916930 | G | A | 2 / 21 | p.G106D | c.317G>A | COSV55926774 | Tier2 | 1 / 560 |
| chr3:178916936 | G | A | 2 / 21 | p.R108H | c.323G>A | COSV55873442 | Tier2 | 12 / 560 |
| chr3:178916937 | TGAA | T | 2 / 21 | p.E110del | c.328_330delGAA | COSV55874554 | Tier2 | 1 / 560 |
| chr3:178916944 | A | G | 2 / 21 | p.K111E | c.331A>G | COSV55878486 | Tier1 | 3 / 560 |
| chr3:178916946 | G | T | 2 / 21 | p.K111N | c.333G>T | COSV55876538 | Tier2 | 3 / 560 |
| chr3:178917478 | G | A | 3 / 21 | p.G118D | c.353G>A | COSV55877290 | Tier2 | 8 / 560 |
| chr3:178921548 | G | A | 5 / 21 | p.V344M | c.1030G>A | COSV55881590 | Tier2 | 3 / 560 |
| chr3:178921549 | T | G | 5 / 21 | p.V344G | c.1031T>G | COSV55876054 | Tier2 | 1 / 560 |
| chr3:178921551 | A | C | 5 / 21 | p.N345H | c.1033A>C | COSV55957136 | Tier2 | 1 / 560 |
| chr3:178921552 | A | C | 5 / 21 | p.N345T | c.1034A>C | COSV55902438 | Tier2 | 2 / 560 |
| chr3:178921552 | A | T | 5 / 21 | p.N345I | c.1034A>T | COSV55902706 | Tier2 | 1 / 560 |
| chr3:178921553 | T | A | 5 / 21 | p.N345K | c.1035T>A | COSV55873276 | Tier1 | 10 / 560 |
| chr3:178922321 | G | A | 6 / 21 | p.G364R | c.1090G>A | COSV55903618 | Tier2 | 1 / 560 |
| chr3:178922324 | G | A | 6 / 21 | p.E365K | c.1093G>A | COSV55881636 | Tier2 | 1 / 560 |
| chr3:178922363 | T | C | 6 / 21 | p.C378R | c.1132T>C | COSV55882697 | Tier2 | 1 / 560 |
| chr3:178927980 | T | C | 8 / 21 | p.C420R | c.1258T>C | COSV55874020 | Tier1 | 17 / 560 |
| chr3:178928067 | C | A | 8 / 21 | p.P449T | c.1345C>A | COSV55932192 | Tier1 | 1 / 560 |
| chr3:178928079 | G | A | 8 / 21 | p.E453K | c.1357G>A | COSV55874585 | Tier1 | 8 / 560 |
| chr3:178928079 | G | C | 8 / 21 | p.E453Q | c.1357G>C | COSV55873816 | Tier2 | 1 / 560 |
| chr3:178936082 | G | A | 10 / 21 | p.E542K | c.1624G>A | COSV55873227 | Tier1 | 66 / 560 |
| chr3:178936082 | G | C | 10 / 21 | p.E542Q | c.1624G>C | COSV55894248 | Tier2 | 4 / 560 |
| chr3:178936083 | A | T | 10 / 21 | p.E542V | c.1625A>T | COSV55881194 | Tier1 | 2 / 560 |
| chr3:178936083 | A | C | 10 / 21 | p.E542A | c.1625A>C | COSV55893680 | Tier2 | 2 / 560 |
| chr3:178936091 | G | A | 10 / 21 | p.E545K | c.1633G>A | COSV55873239 | Tier1 | 88 / 560 |
| chr3:178936091 | G | C | 10 / 21 | p.E545Q | c.1633G>C | COSV55878227 | Tier1 | 1 / 560 |
| chr3:178936092 | A | C | 10 / 21 | p.E545A | c.1634A>C | COSV55873209 | Tier1 | 8 / 560 |
| chr3:178936092 | A | G | 10 / 21 | p.E545G | c.1634A>G | COSV55873220 | Tier1 | 8 / 560 |
| chr3:178936093 | G | C | 10 / 21 | p.E545D | c.1635G>C | COSV55875881 | Tier2 | 2 / 560 |
| chr3:178936094 | C | A | 10 / 21 | p.Q546K | c.1636C>A | COSV55873527 | Tier1 | 13 / 560 |
| chr3:178936094 | C | G | 10 / 21 | p.Q546E | c.1636C>G | COSV55882350 | Tier1 | 2 / 560 |
| chr3:178936095 | A | G | 10 / 21 | p.Q546R | c.1637A>G | COSV55876869 | Tier1 | 7 / 560 |
| chr3:178936095 | A | C | 10 / 21 | p.Q546P | c.1637A>C | COSV55875400 | Tier1 | 2 / 560 |
| chr3:178936096 | G | T | 10 / 21 | p.Q546H | c.1638G>T | COSV55883555 | Tier2 | 1 / 560 |
| chr3:178938934 | G | A | 14 / 21 | p.E726K | c.2176G>A | COSV55875460 | Tier2 | 6 / 560 |
| chr3:178947865 | G | C | 19 / 21 | p.G914R | c.2740G>C | COSV55944375 | Tier2 | 1 / 560 |
| chr3:178948136 | G | A | 20 / 21 | p.E970K | c.2908G>A | COSV55875690 | Tier2 | 2 / 560 |
| chr3:178952007 | A | G | 21 / 21 | p.Y1021C | c.3062A>G | COSV55882161 | Tier2 | 5 / 560 |
| chr3:178952018 | A | G | 21 / 21 | p.T1025A | c.3073A>G | COSV55873252 | Tier1 | 5 / 560 |
| chr3:178952072 | A | G | 21 / 21 | p.M1043V | c.3127A>G | COSV55879858 | Tier1 | 3 / 560 |
| chr3:178952073 | T | C | 21 / 21 | p.M1043T | c.3128T>C | COSV55900332 | Tier2 | 1 / 560 |
| chr3:178952074 | G | A | 21 / 21 | p.M1043I | c.3129G>A | COSV55873376 | Tier1 | 1 / 560 |
| chr3:178952074 | G | C | 21 / 21 | p.M1043I | c.3129G>C | COSV55898229 | Tier1 | 1 / 560 |
| chr3:178952074 | G | T | 21 / 21 | p.M1043I | c.3129G>T | COSV55878974 | Tier1 | 4 / 560 |
| chr3:178952077 | T | A | 21 / 21 | p.N1044K | c.3132T>A | COSV55897116 | Tier1 | 1 / 560 |
| chr3:178952077 | T | G | 21 / 21 | p.N1044K | c.3132T>G | COSV55875087 | Tier1 | 1 / 560 |
| chr3:178952084 | C | T | 21 / 21 | p.H1047Y | c.3139C>T | COSV55876499 | Tier1 | 4 / 560 |
| chr3:178952085 | A | G | 21 / 21 | p.H1047R | c.3140A>G | COSV55873195 | Tier1 | 120 / 560 |
| chr3:178952085 | A | T | 21 / 21 | p.H1047L | c.3140A>T | COSV55873401 | Tier1 | 21 / 560 |
| chr3:178952090 | G | C | 21 / 21 | p.G1049R | c.3145G>C | COSV55874453 | Tier1 | 7 / 560 |
| chr3:178952090 | G | A | 21 / 21 | p.G1049S | c.3145G>A | COSV55878564 | Tier2 | 2 / 560 |
| chr3:178952142 | C | T | 21 / 21 | p.A1066V | c.3197C>T | COSV55892714 | Tier2 | 1 / 560 |