Side Menu
SOX9
基本情報
- 遺伝子名
-
SRY-box transcription factor 9
- 慣用名
-
CMD1, CMPD1, SRA1, SRXX2, SRXY10
- 遺伝子分類
-
がん遺伝子 / がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
分化
- シグナル伝達経路
-
WNT
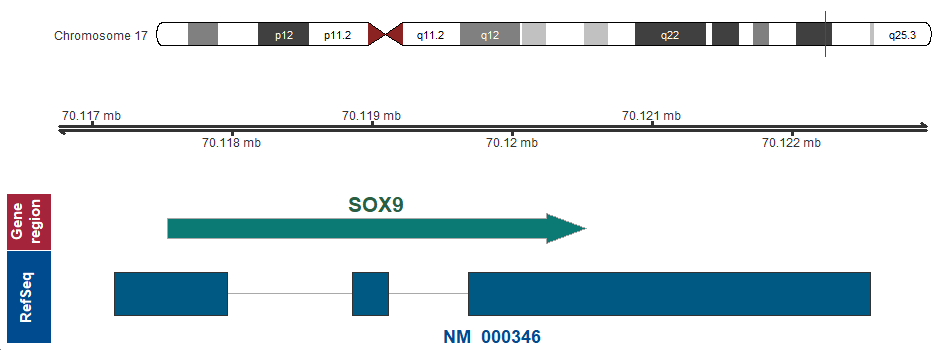
- 染色体上の位置 (GRCh37/hg19)
-
17q24.3 (chr17:70117533..70120528)
- アミノ酸配列の長さ
-
509
遺伝子マップ
解説
SOX9は臓器および骨格の発達に関与する転写制御因子であり、その発現はWNTシグナル経路により誘導される。一方で、WNTシグナル経路のシグナル伝達因子であるβ-カテニンを制御するβ-カテニン分解複合体の構成因子GSK3βに作用し、β-カテニンのリン酸化と分解を促進することでWNTシグナル経路の活性を抑制している。
シグナル伝達経路

遺伝子変異総量(Tumor Mutation Burden)の分布TMB

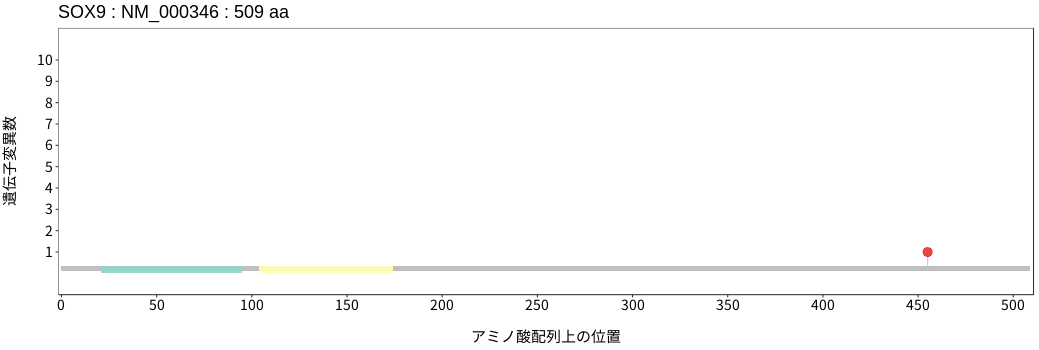
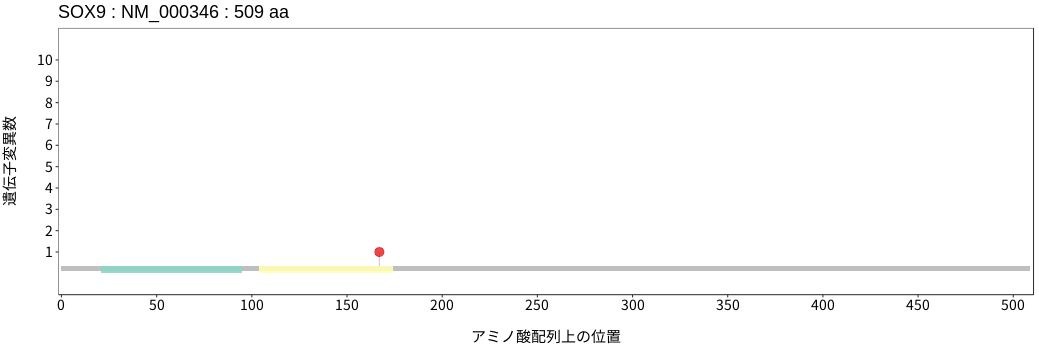
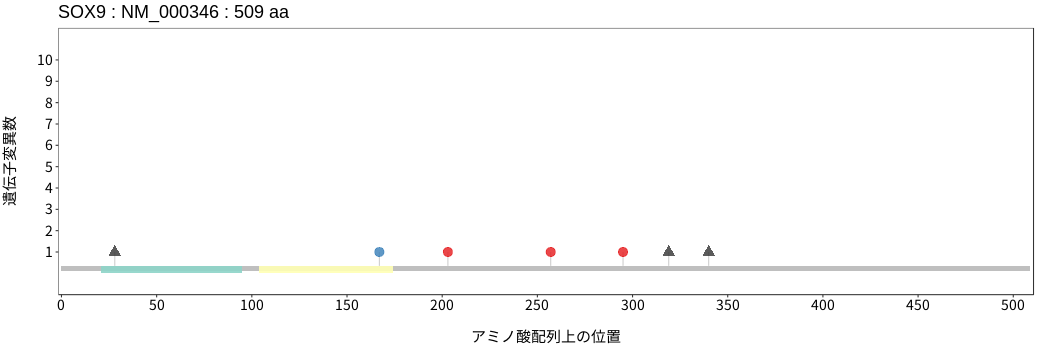
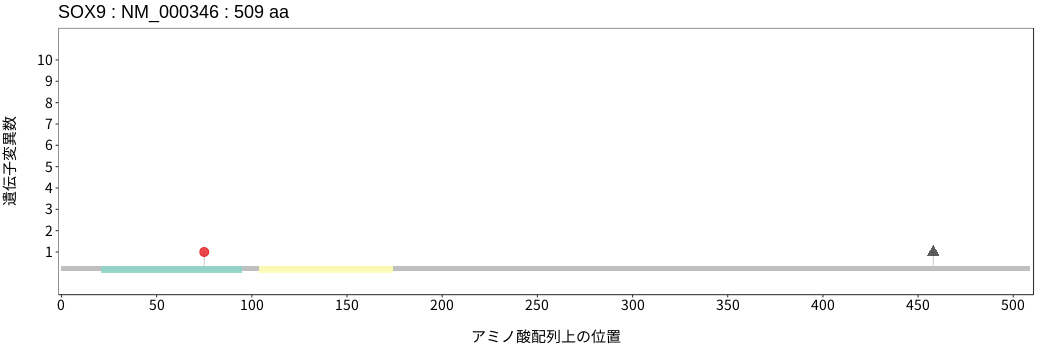
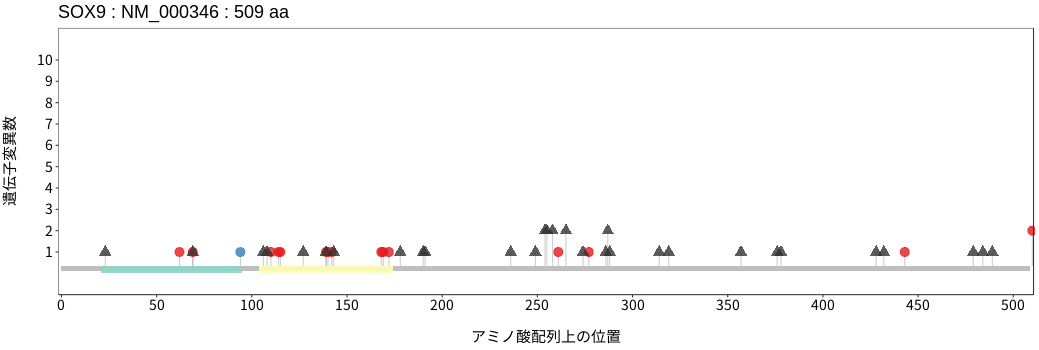
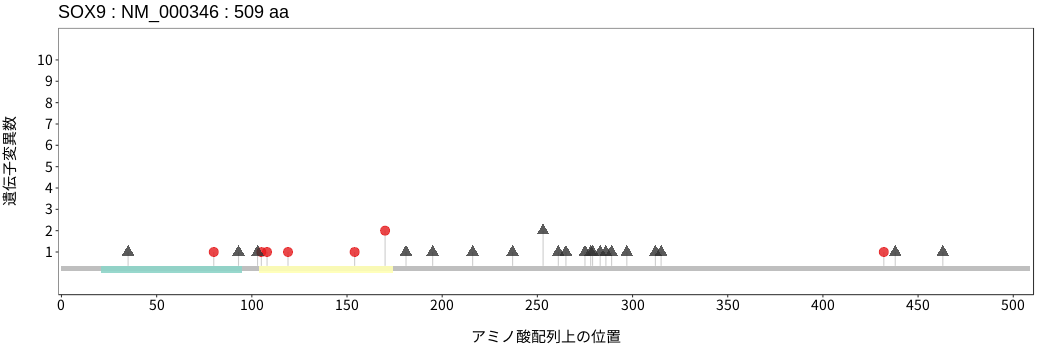
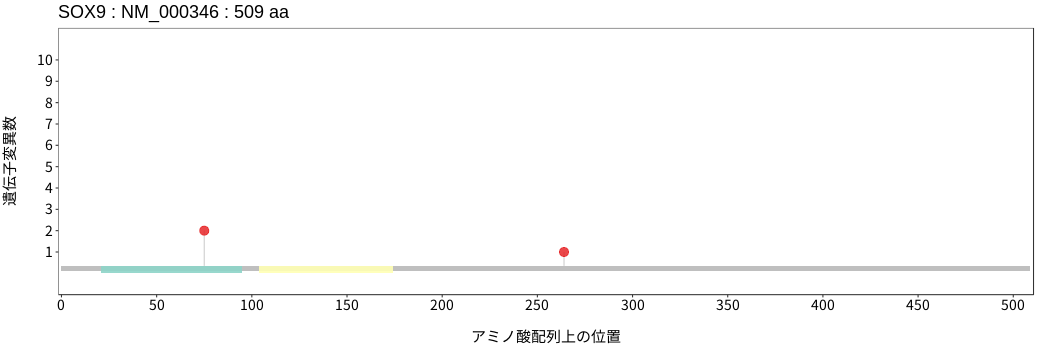
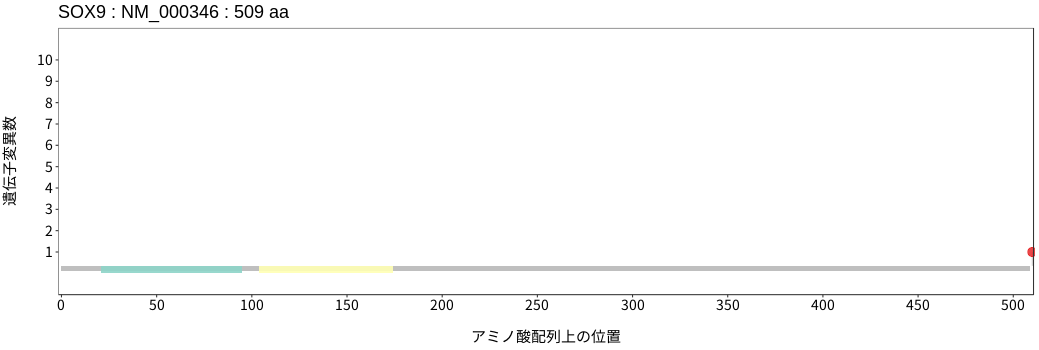
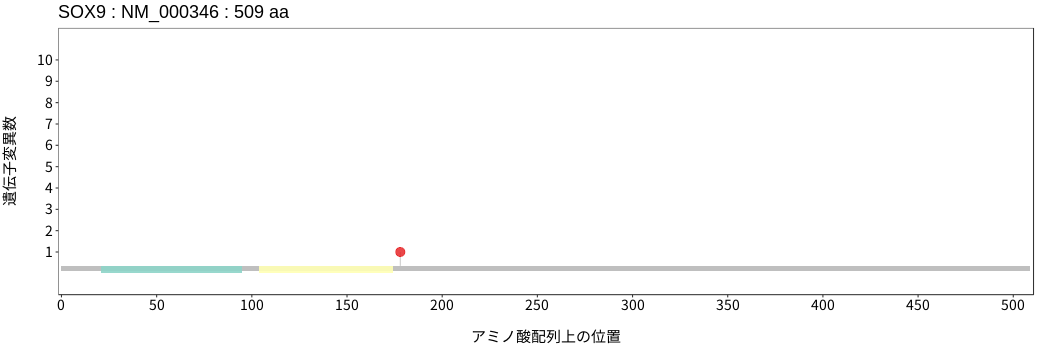
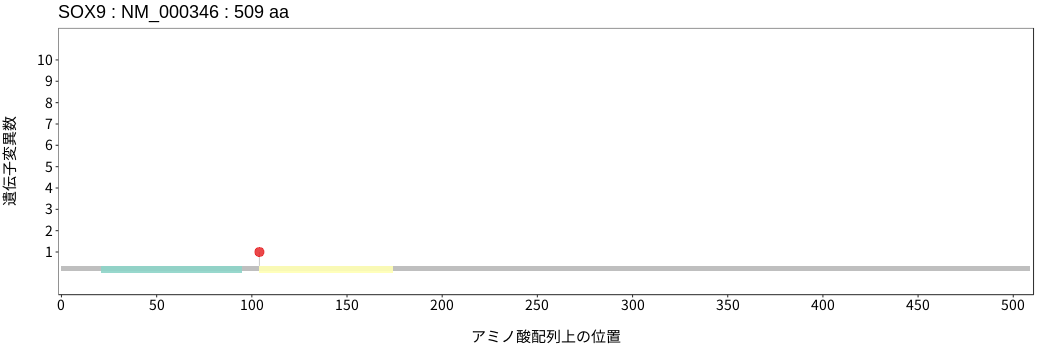
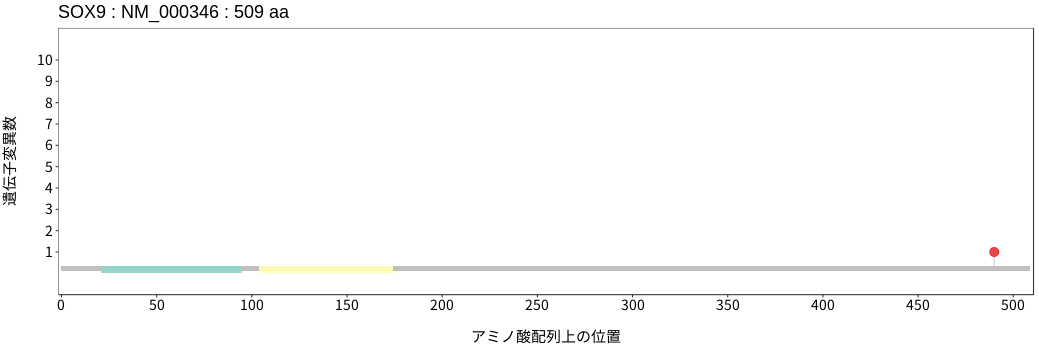
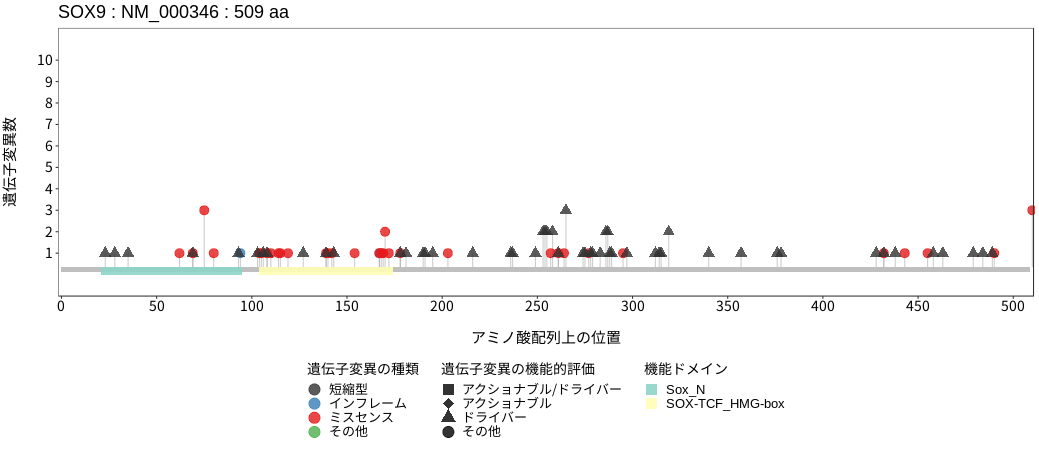
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr17:70117599 | AGC | A | 1 / 3 | p.S23fs | c.68_69delGC | Tier2 | 1 / 117 | |
| chr17:70117614 | G | T | 1 / 3 | p.E28* | c.82G>T | COSV55422908 | Tier2 | 1 / 117 |
| chr17:70117635 | T | TG | 1 / 3 | p.C35fs | c.104dupG | Tier2 | 1 / 117 | |
| chr17:70117738 | T | TA | 1 / 3 | p.F69fs | c.206_207insA | Tier2 | 1 / 117 | |
| chr17:70117807 | C | CGG | 1 / 3 | p.V93fs | c.276_277dupGG | Tier2 | 1 / 117 | |
| chr17:70117835 | C | CAA | 1 / 3 | p.P103fs | c.304_305dupAA | Tier2 | 1 / 117 | |
| chr17:70117848 | A | T | 1 / 3 | p.K106* | c.316A>T | COSV55430122 | Tier2 | 1 / 117 |
| chr17:70117853 | GCCCATGAACGCCTTCAT | G | 1 / 3 | p.P108fs | c.322_338delCCCATGAACGCCTTCAT | Tier2 | 1 / 117 | |
| chr17:70117911 | T | TC | 1 / 3 | p.Y127fs | c.379_380insC | Tier2 | 1 / 117 | |
| chr17:70117944 | A | ACG | 1 / 3 | p.L139fs | c.414_415dupGC | Tier2 | 1 / 117 | |
| chr17:70117960 | G | A | 1 / 3 | p.W143* | c.428G>A | COSV55423111 | Tier2 | 1 / 117 |
| chr17:70118959 | G | GC | 2 / 3 | p.R178fs | c.532dupC | Tier2 | 1 / 117 | |
| chr17:70118970 | C | A | 2 / 3 | p.S181* | c.542C>A | Tier2 | 1 / 117 | |
| chr17:70118996 | G | T | 2 / 3 | p.E190* | c.568G>T | COSV55430562 | Tier2 | 1 / 117 |
| chr17:70118999 | G | T | 2 / 3 | p.E191* | c.571G>T | Tier2 | 1 / 117 | |
| chr17:70119011 | C | T | 2 / 3 | p.Q195* | c.583C>T | Tier2 | 1 / 117 | |
| chr17:70119072 | CCTCCG | C | 2 / 3 | p.S216fs | c.645_649delCTCCG | Tier2 | 1 / 117 | |
| chr17:70119696 | GCCCACCGACCCCA | G | 3 / 3 | p.T236fs | c.705_717delGACCCCACCCACC | Tier2 | 1 / 117 | |
| chr17:70119699 | CACCGACCCCACCCACCACCCCCAAA | C | 3 / 3 | p.P237fs | c.708_732delCCCACCCACCACCCCCAAAACCGAC | Tier2 | 1 / 117 | |
| chr17:70119740 | G | GGC | 3 / 3 | p.K249fs | c.743_744dupGC | COSV55424964 | Tier2 | 1 / 117 |
| chr17:70119746 | G | GCTGAC | 3 / 3 | p.K253fs | c.753_757dupCCTGA | Tier2 | 1 / 117 | |
| chr17:70119752 | C | CTG | 3 / 3 | p.K253fs | c.755_756dupTG | Tier2 | 1 / 117 | |
| chr17:70119758 | C | T | 3 / 3 | p.R254* | c.760C>T | COSV55424668 | Tier2 | 2 / 117 |
| chr17:70119758 | CGA | C | 3 / 3 | p.E255fs | c.764_765delAG | COSV55424287 | Tier2 | 1 / 117 |
| chr17:70119761 | G | T | 3 / 3 | p.E255* | c.763G>T | Tier2 | 1 / 117 | |
| chr17:70119765 | G | GGC | 3 / 3 | p.P258fs | c.770_771dupGC | COSV55424143 | Tier2 | 1 / 117 |
| chr17:70119769 | C | CG | 3 / 3 | p.P258fs | c.771_772insG | Tier2 | 1 / 117 | |
| chr17:70119779 | G | T | 3 / 3 | p.E261* | c.781G>T | COSV55427494 | Tier2 | 1 / 117 |
| chr17:70119791 | C | T | 3 / 3 | p.Q265* | c.793C>T | COSV55423080 | Tier2 | 3 / 117 |
| chr17:70119814 | C | CGT | 3 / 3 | p.D274fs | c.818_819dupTG | COSV55422681 | Tier2 | 1 / 117 |
| chr17:70119820 | CA | C | 3 / 3 | p.I275fs | c.823delA | Tier2 | 1 / 117 | |
| chr17:70119829 | G | GC | 3 / 3 | p.L278fs | c.832dupC | Tier2 | 1 / 117 | |
| chr17:70119831 | T | TCCGC | 3 / 3 | p.S279fs | c.833_834insCCGC | Tier2 | 1 / 117 | |
| chr17:70119842 | G | GTC | 3 / 3 | p.I283fs | c.845_846dupTC | Tier2 | 1 / 117 | |
| chr17:70119851 | A | AACATCGAG | 3 / 3 | p.F289fs | c.856_863dupATCGAGAC | Tier2 | 1 / 117 | |
| chr17:70119853 | CAT | C | 3 / 3 | p.I286fs | c.856_857delAT | Tier2 | 1 / 117 | |
| chr17:70119855 | TC | T | 3 / 3 | p.I286fs | c.858delC | Tier2 | 1 / 117 | |
| chr17:70119855 | T | TC | 3 / 3 | p.E287fs | c.858dupC | Tier2 | 1 / 117 | |
| chr17:70119856 | C | CGA | 3 / 3 | p.T288fs | c.861_862dupGA | Tier2 | 1 / 117 | |
| chr17:70119857 | G | T | 3 / 3 | p.E287* | c.859G>T | COSV55422733 | Tier2 | 1 / 117 |
| chr17:70119889 | C | A | 3 / 3 | p.Y297* | c.891C>A | COSV55430013 | Tier2 | 1 / 117 |
| chr17:70119928 | C | CG | 3 / 3 | p.Q312fs | c.932dupG | Tier2 | 1 / 117 | |
| chr17:70119935 | G | GTC | 3 / 3 | p.T314fs | c.938_939dupTC | Tier2 | 1 / 117 | |
| chr17:70119941 | T | TA | 3 / 3 | p.Y315fs | c.944dupA | Tier2 | 1 / 117 | |
| chr17:70119952 | C | CT | 3 / 3 | p.Y319fs | c.955dupT | Tier2 | 1 / 117 | |
| chr17:70119955 | C | A | 3 / 3 | p.Y319* | c.957C>A | Tier2 | 1 / 117 | |
| chr17:70120016 | C | T | 3 / 3 | p.Q340* | c.1018C>T | COSV55426963 | Tier2 | 1 / 117 |
| chr17:70120067 | C | T | 3 / 3 | p.Q357* | c.1069C>T | COSV55429202(SNP) | Tier2 | 1 / 117 |
| chr17:70120124 | C | T | 3 / 3 | p.Q376* | c.1126C>T | Tier2 | 1 / 117 | |
| chr17:70120130 | C | T | 3 / 3 | p.Q378* | c.1132C>T | Tier2 | 1 / 117 | |
| chr17:70120282 | C | G | 3 / 3 | p.Y428* | c.1284C>G | Tier2 | 1 / 117 | |
| chr17:70120294 | C | A | 3 / 3 | p.Y432* | c.1296C>A | Tier2 | 1 / 117 | |
| chr17:70120311 | C | G | 3 / 3 | p.S438* | c.1313C>G | Tier2 | 1 / 117 | |
| chr17:70120370 | C | T | 3 / 3 | p.Q458* | c.1372C>T | COSV55428140 | Tier2 | 1 / 117 |
| chr17:70120381 | CCT | C | 3 / 3 | p.Y463fs | c.1386_1387delCT | Tier2 | 1 / 117 | |
| chr17:70120430 | ACC | A | 3 / 3 | p.P479fs | c.1436_1437delCC | Tier2 | 1 / 117 | |
| chr17:70120446 | CCT | C | 3 / 3 | p.S484fs | c.1451_1452delCT | COSV55423201 | Tier2 | 1 / 117 |
| chr17:70120460 | TC | T | 3 / 3 | p.I489fs | c.1464delC | Tier2 | 1 / 117 |