Side Menu
B2M
基本情報
- 遺伝子名
-
beta-2-microglobulin
- 慣用名
-
IMD43
- 遺伝子分類
-
がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
免疫
- シグナル伝達経路
-
Immune
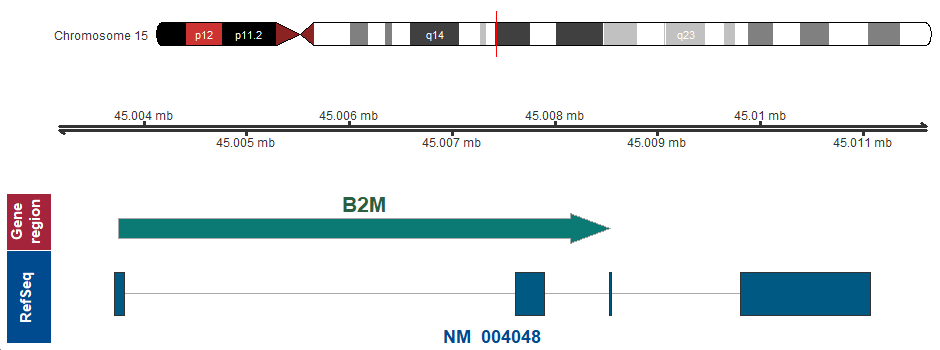
- 染色体上の位置 (GRCh37/hg19)
-
15q21.1 (chr15:45003745..45008540)
- アミノ酸配列の長さ
-
119
遺伝子マップ
解説
B2Mは、ほぼすべての有核細胞の表面にある主要組織適合遺伝子複合体(MHC)クラスI重鎖に関連して発見された血清タンパク質をコードする。この遺伝子は、大部分がβプリーツシート構造であり、いくつかの病状でアミロイド原線維を形成する。この遺伝子のバリアントは、異化亢進による低タンパク血症と関連している。
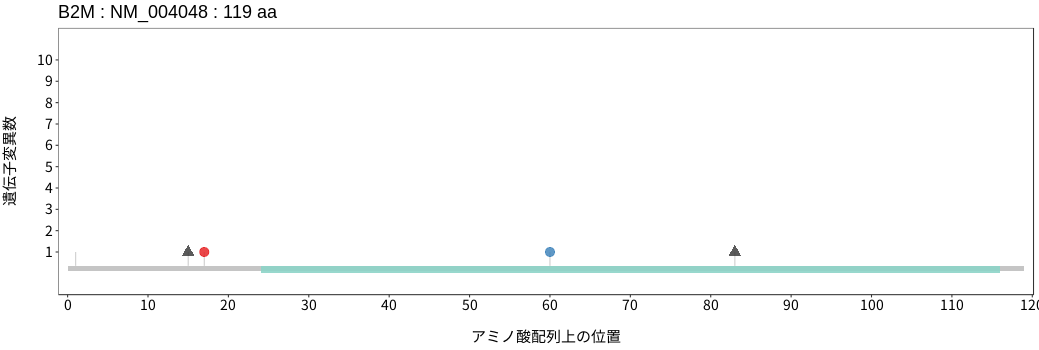
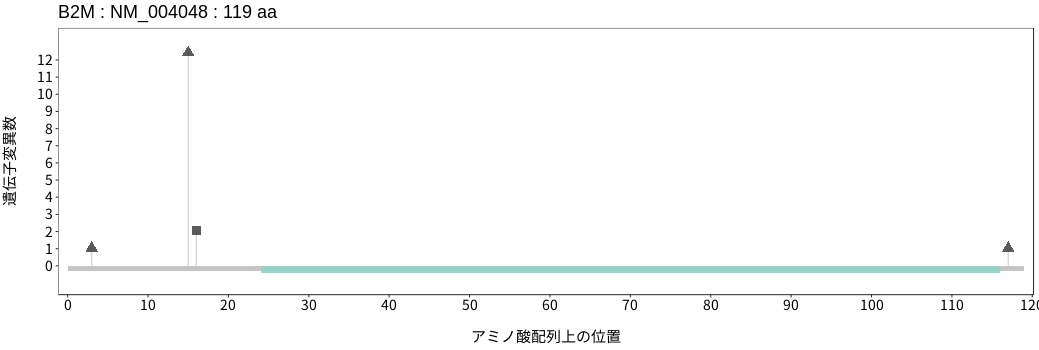
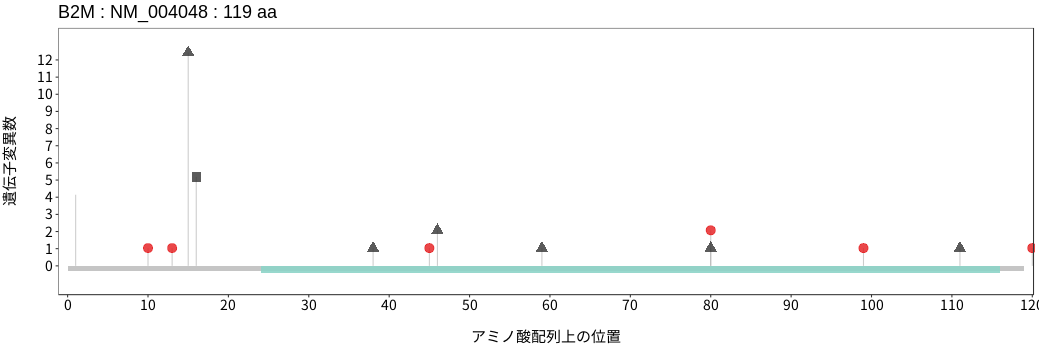
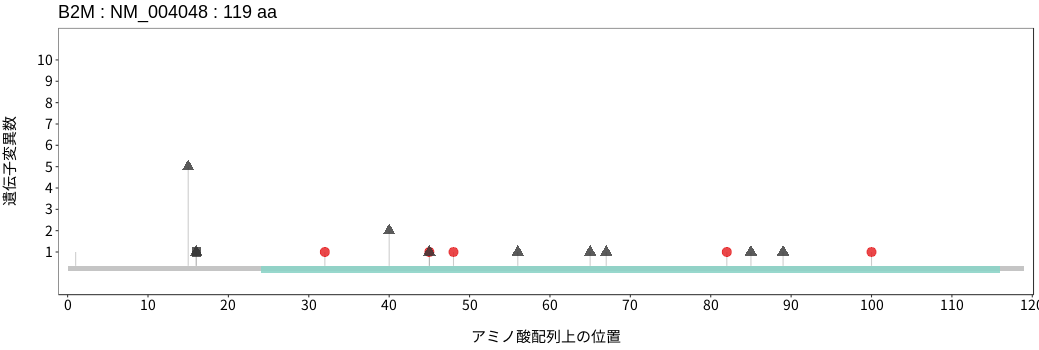
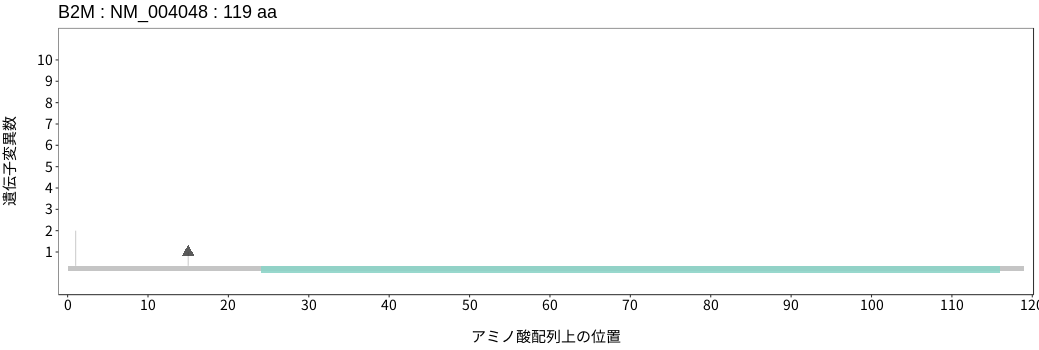
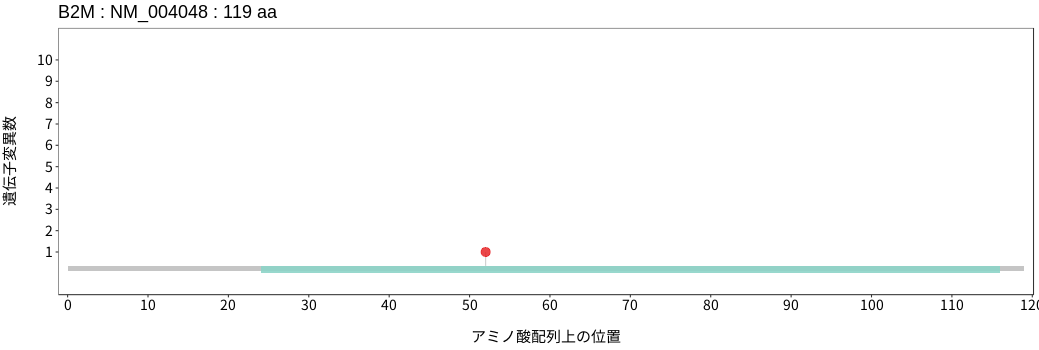
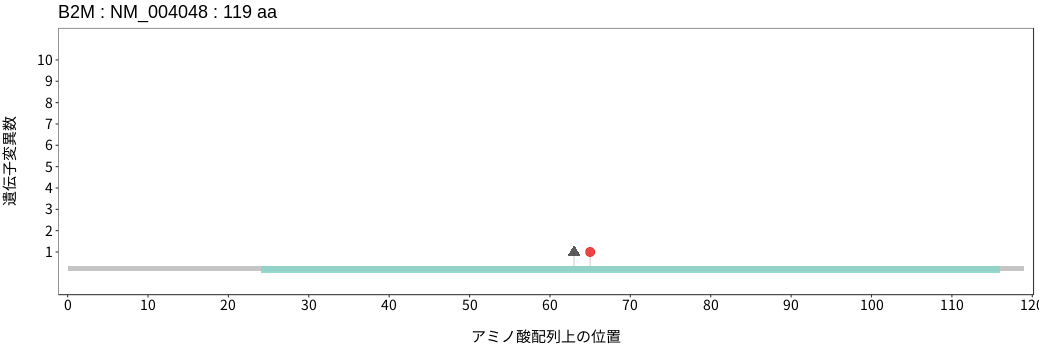
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

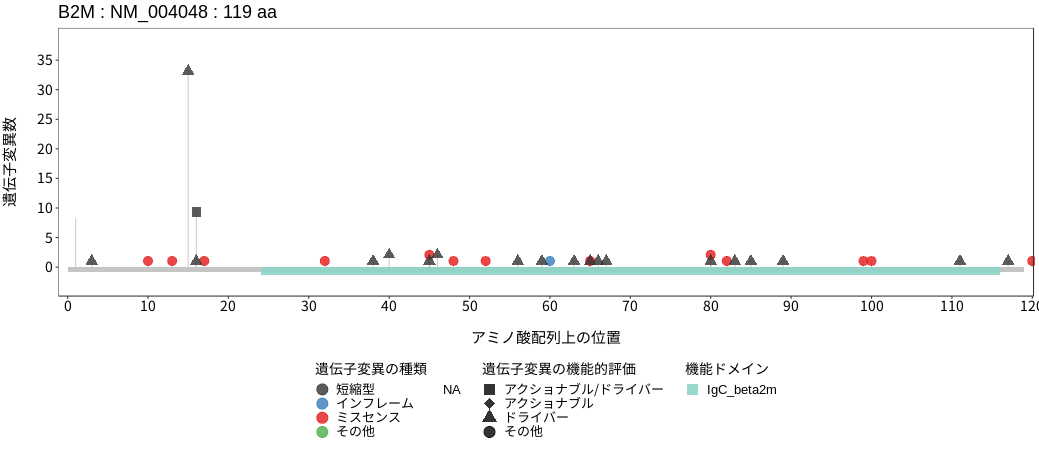
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr15:45003745 | A | G | 1 / 4 | p.M1? | c.1A>G | COSV62562870 | Tier2 | 4 / 92 |
| chr15:45003745 | A | T | 1 / 4 | p.M1? | c.1A>T | COSV62562840 | Tier2 | 2 / 92 |
| chr15:45003746 | T | C | 1 / 4 | p.M1? | c.2T>C | COSV62563174 | Tier2 | 2 / 92 |
| chr15:45003750 | TCG | T | 1 / 4 | p.R3fs | c.8_9delGC | Tier2 | 1 / 92 | |
| chr15:45003780 | ACT | A | 1 / 4 | p.L15fs | c.43_44delCT | COSV62562847 | Tier2 | 32 / 92 |
| chr15:45003780 | A | ACT | 1 / 4 | p.S16fs | c.43_44dupCT | Tier2 | 1 / 92 | |
| chr15:45003785 | CTCTT | C | 1 / 4 | p.S16fs | c.45_48delTTCT | COSV62562863|COSV62562993 | Tier2 | 9 / 92 |
| chr15:45007665 | G | T | 2 / 4 | p.G38* | c.112G>T | Tier2 | 1 / 92 | |
| chr15:45007672 | C | A | 2 / 4 | p.S40* | c.119C>A | Tier2 | 2 / 92 | |
| chr15:45007688 | CTA | C | 2 / 4 | p.Y46fs | c.137_138delAT | COSV62563472 | Tier2 | 2 / 92 |
| chr15:45007688 | C | A | 2 / 4 | p.C45* | c.135C>A | COSV62563284 | Tier2 | 1 / 92 |
| chr15:45007719 | G | T | 2 / 4 | p.E56* | c.166G>T | COSV62563605 | Tier2 | 1 / 92 |
| chr15:45007729 | T | G | 2 / 4 | p.L59* | c.176T>G | Tier2 | 1 / 92 | |
| chr15:45007740 | G | T | 2 / 4 | p.G63* | c.187G>T | COSV62564125 | Tier2 | 1 / 92 |
| chr15:45007740 | GGA | G | 2 / 4 | p.R65fs | c.194_195delGA | COSV62563423 | Tier2 | 1 / 92 |
| chr15:45007747 | GA | G | 2 / 4 | p.I66fs | c.196delA | Tier2 | 1 / 92 | |
| chr15:45007752 | G | T | 2 / 4 | p.E67* | c.199G>T | COSV62564724 | Tier2 | 1 / 92 |
| chr15:45007793 | G | A | 2 / 4 | p.W80* | c.240G>A | Tier2 | 1 / 92 | |
| chr15:45007799 | CTA | C | 2 / 4 | p.Y83fs | c.248_249delAT | COSV62563005 | Tier2 | 1 / 92 |
| chr15:45007801 | ATC | A | 2 / 4 | p.L85fs | c.252_253delCT | COSV62567214 | Tier2 | 1 / 92 |
| chr15:45007818 | G | T | 2 / 4 | p.E89* | c.265G>T | COSV62563917 | Tier2 | 1 / 92 |
| chr15:45007883 | CAA | C | 2 / 4 | p.K111fs | c.331_332delAA | Tier2 | 1 / 92 | |
| chr15:45008529 | C | T | 3 / 4 | p.R117* | c.349C>T | COSV62562883 | Tier2 | 1 / 92 |