Side Menu
BRAF
基本情報
- 遺伝子名
-
B-Raf proto-oncogene, serine/threonine kinase
- 慣用名
-
B-RAF1, B-raf, BRAF1, NS7, RAFB1
- 遺伝子分類
-
がん遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
腫瘍形成・増殖
- シグナル伝達経路
-
MAPK
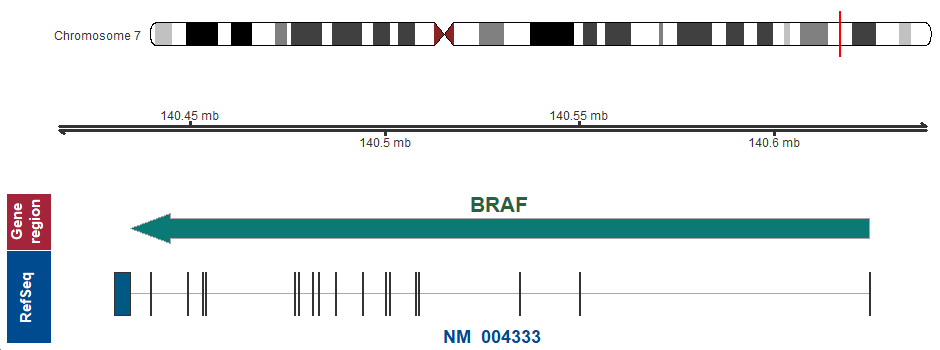
- 染色体上の位置 (GRCh37/hg19)
-
7q34 (chr7:140434397..140624503, complement)
- アミノ酸配列の長さ
-
766
遺伝子マップ
解説
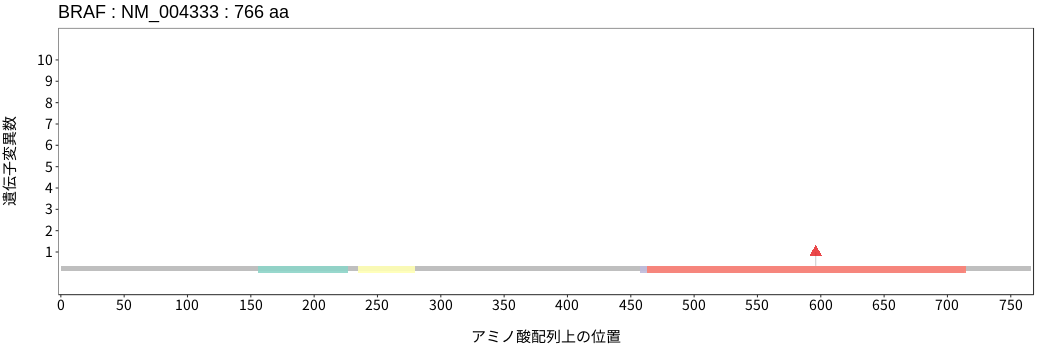
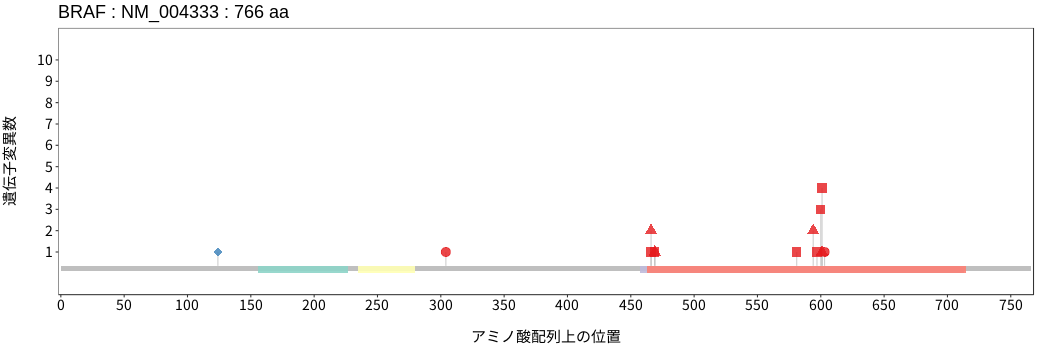
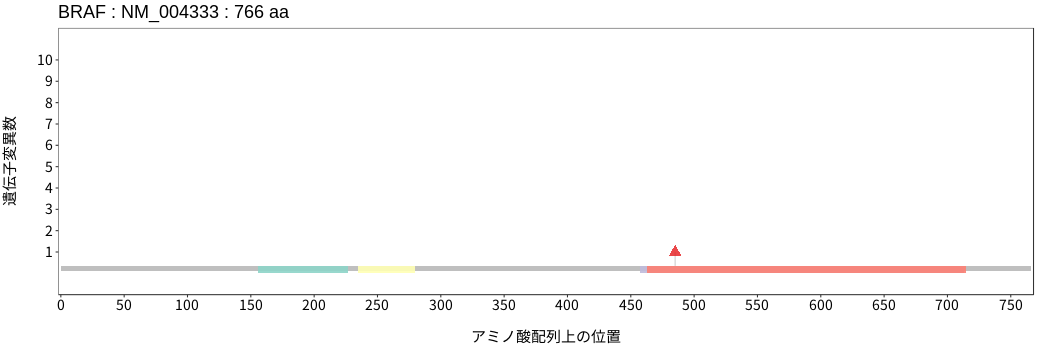
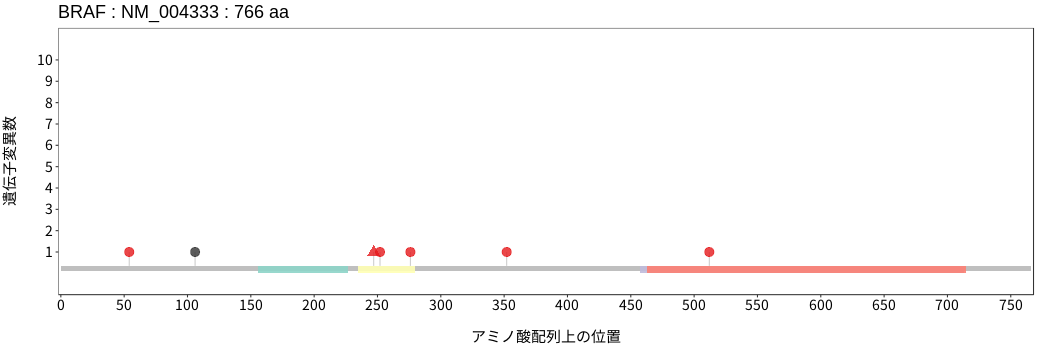
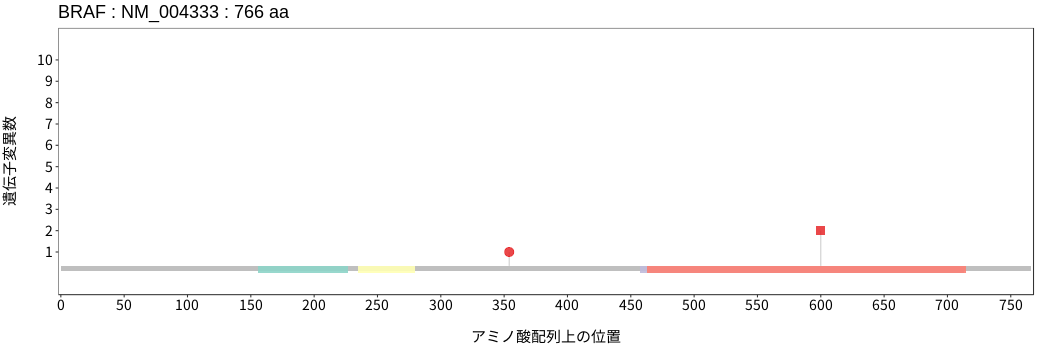
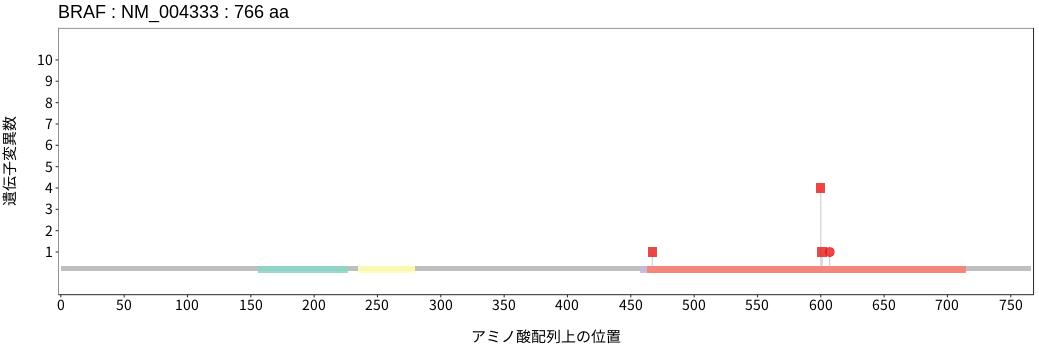
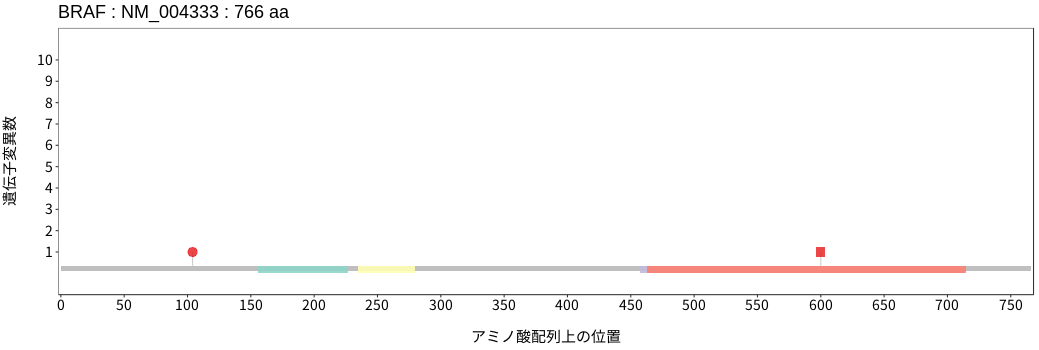
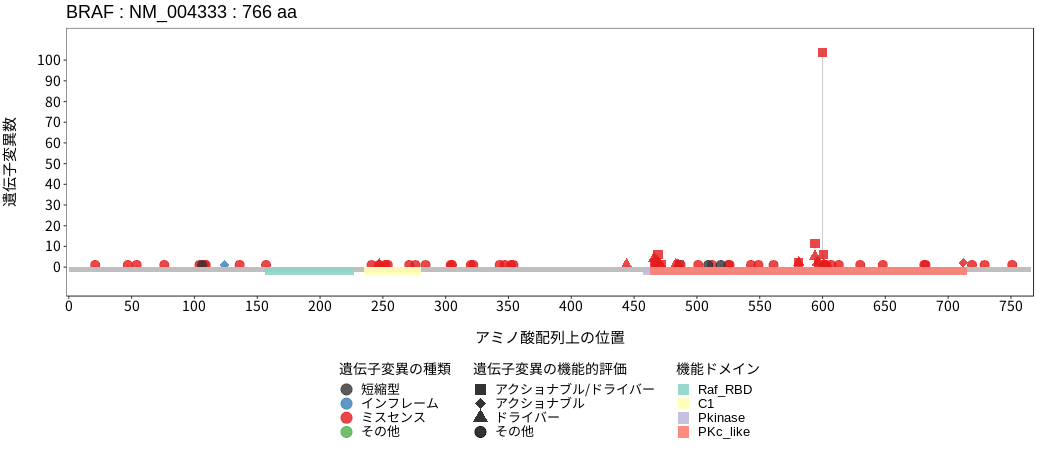
BRAFはRAFファミリーに属するセリン/スレオニンキナーゼである。細胞増殖を含む細胞機能の調節に関与するMAPKシグナルにおいてRASの下流に位置し、RASからのシグナルを下流のMEKをリン酸化することで伝達する。体細胞変異は悪性黒色腫および甲状腺癌において高い頻度で認められる。BRAFの変異はBRAF自体のキナーゼ活性への影響の程度に加えて、上流RASシグナルへの依存性およびBRAF/CRAFとの二量体形成の有無により3種類のクラスに分類される。Class I変異はホットスポットである600番目のバリン残基の変異であり、上流RASからのシグナル非依存的にかつ単量体でBRAFのキナーゼ活性を強く亢進させることができる。Class II変異は上流RASからのシグナル非依存的にBRAFのキナーゼ活性を亢進できるものの、その活性化にはBRAF/CRAFとの二量体形成を必要とする。Class III変異はClass I/II変異とは異なりBRAFのキナーゼ活性の亢進は誘導しないものの、BRAF/CRAFとの二量体を形成し上流RASからのシグナルを受け、MAPKシグナル伝達経路全体の活性を増幅する増幅器としての働きを示す変異である。
国内承認薬 (分子標的治療薬)
2022/06/10 更新
| 一般名 (販売名) | 外部サイトへのリンク | |
|---|---|---|
| ソラフェニブ(ネクサバール)※ | PMDA | KEGG DRUG |
| ベムラフェニブ(ゼルボラフ) | PMDA | KEGG DRUG |
| レゴラフェニブ(スチバーガ)※ | PMDA | KEGG DRUG |
| ダブラフェニブ(タフィンラー) | PMDA | KEGG DRUG |
| エンコラフェニブ(ビラフトビ) | PMDA | KEGG DRUG |
※JCGAに掲載されている460遺伝子の中に複数の標的遺伝子がある薬剤 (他の遺伝子においても当該薬剤が表記されています)
薬剤の標的とされる遺伝子において該当する薬剤名を列挙しています。
各薬剤の承認情報(適応がん種、効果・効能の情報)を反映したものではありません。
国内承認薬に関する情報の詳細は、独立行政法人 医薬品医療機器総合機構(PMDA)が公開している情報を必ず確認してください。
薬剤の標的とされる遺伝子において該当する薬剤名を列挙しています。
各薬剤の承認情報(適応がん種、効果・効能の情報)を反映したものではありません。
国内承認薬に関する情報の詳細は、独立行政法人 医薬品医療機器総合機構(PMDA)が公開している情報を必ず確認してください。
シグナル伝達経路

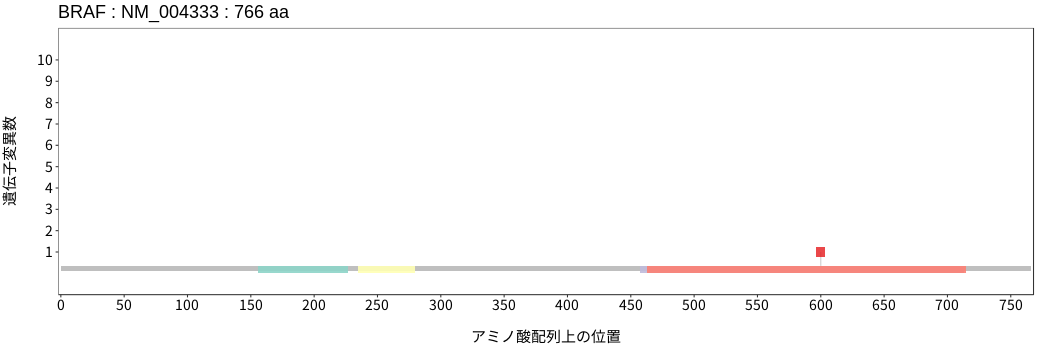
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

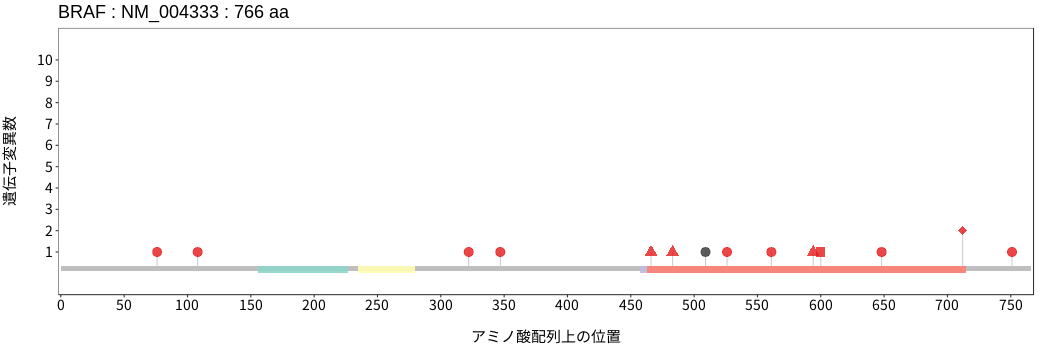
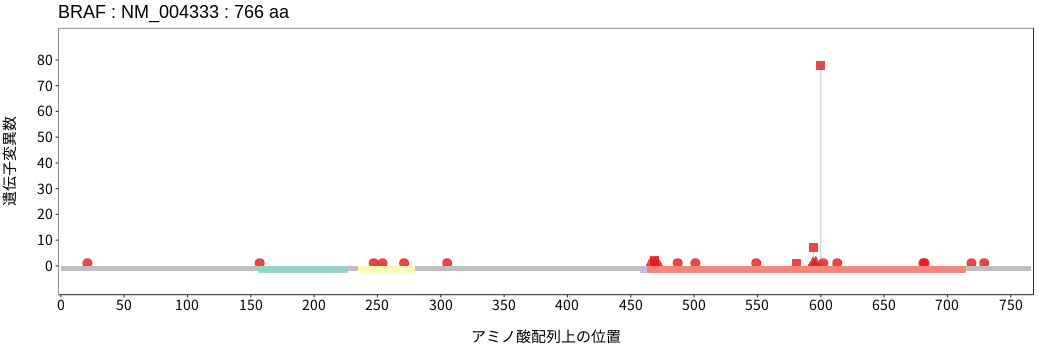
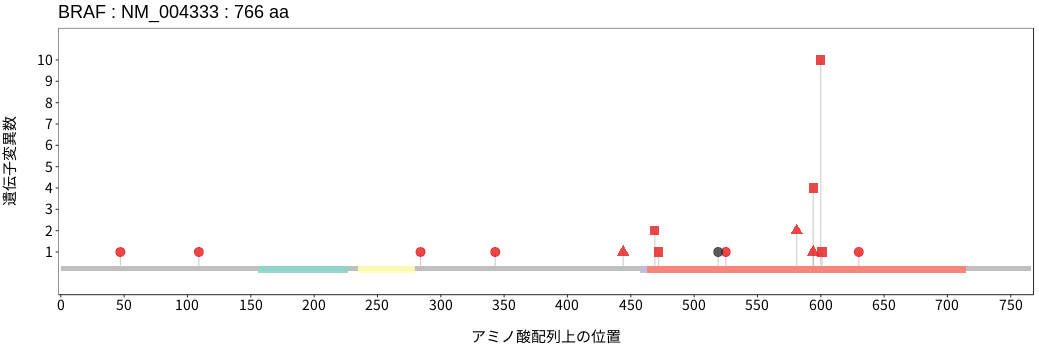
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr7:140501331 | A | C | 6 / 18 | p.F247L | c.741T>G | COSV56165267 | Tier2 | 1 / 200 |
| chr7:140481393 | T | C | 11 / 18 | p.Y472C | c.1415A>G | COSV56067953 | Tier1 | 1 / 200 |
| chr7:140481397 | C | A | 11 / 18 | p.V471F | c.1411G>T | COSV56229839 | Tier1 | 1 / 200 |
| chr7:140481402 | C | G | 11 / 18 | p.G469A | c.1406G>C | COSV56061424 | Tier1 | 4 / 200 |
| chr7:140481402 | C | T | 11 / 18 | p.G469E | c.1406G>A | COSV56065622 | Tier1 | 2 / 200 |
| chr7:140481403 | C | T | 11 / 18 | p.G469R | c.1405G>A | COSV56070557 | Tier1 | 1 / 200 |
| chr7:140481403 | C | G | 11 / 18 | p.G469R | c.1405G>C | COSV56082352 | Tier1 | 1 / 200 |
| chr7:140481408 | G | A | 11 / 18 | p.S467L | c.1400C>T | COSV56106484 | Tier1 | 1 / 200 |
| chr7:140481411 | C | T | 11 / 18 | p.G466E | c.1397G>A | COSV56059390 | Tier1 | 1 / 200 |
| chr7:140481411 | C | G | 11 / 18 | p.G466A | c.1397G>C | COSV56070593 | Tier1 | 1 / 200 |
| chr7:140481411 | C | A | 11 / 18 | p.G466V | c.1397G>T | COSV56057462 | Tier1 | 2 / 200 |
| chr7:140481412 | C | G | 11 / 18 | p.G466R | c.1396G>C | COSV56064038 | Tier2 | 1 / 200 |
| chr7:140481412 | C | T | 11 / 18 | p.G466R | c.1396G>A | COSV56099462 | Tier2 | 1 / 200 |
| chr7:140481478 | G | A | 11 / 18 | p.R444W | c.1330C>T | COSV56083156 | Tier2 | 1 / 200 |
| chr7:140477836 | GTAGGTGCTGTCACAT | G | 12 / 18 | p.N486_P490del | c.1457_1471delATGTGACAGCACCTA | COSV56100024 | Tier2 | 1 / 200 |
| chr7:140477853 | C | G | 12 / 18 | p.L485F | c.1455G>C | Tier2 | 1 / 200 | |
| chr7:140477861 | T | C | 12 / 18 | p.K483E | c.1447A>G | COSV56236813 | Tier2 | 1 / 200 |
| chr7:140453132 | T | A | 15 / 18 | p.K601N | c.1803A>T | COSV56058093 | Tier1 | 1 / 200 |
| chr7:140453134 | T | C | 15 / 18 | p.K601E | c.1801A>G | COSV56058494 | Tier1 | 6 / 200 |
| chr7:140453136 | A | T | 15 / 18 | p.V600E | c.1799T>A | COSV56056643 | Tier1 | 100 / 200 |
| chr7:140453145 | A | T | 15 / 18 | p.L597Q | c.1790T>A | COSV56072463 | Tier1 | 1 / 200 |
| chr7:140453148 | C | A | 15 / 18 | p.G596V | c.1787G>T | COSV99951808 | Tier2 | 2 / 200 |
| chr7:140453154 | T | C | 15 / 18 | p.D594G | c.1781A>G | COSV56065695 | Tier1 | 10 / 200 |
| chr7:140453154 | T | G | 15 / 18 | p.D594A | c.1781A>C | COSV56224199 | Tier1 | 1 / 200 |
| chr7:140453155 | C | T | 15 / 18 | p.D594N | c.1780G>A | COSV56061673 | Tier1 | 5 / 200 |
| chr7:140453193 | T | A | 15 / 18 | p.N581I | c.1742A>T | COSV56106422 | Tier1 | 2 / 200 |
| chr7:140453193 | T | C | 15 / 18 | p.N581S | c.1742A>G | COSV56058108 | Tier1 | 2 / 200 |