Side Menu
CDKN2A
基本情報
- 遺伝子名
-
cyclin dependent kinase inhibitor 2A
- 慣用名
-
ARF, CDKN2, CMM2, INK4, INK4A, MLM, MTS-1, MTS1, P14, P14ARF, P16, P16-INK4A, P16INK4, P16INK4A, P19, P19ARF, TP16
- 遺伝子分類
-
がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
細胞周期
- シグナル伝達経路
-
Cell cycle
- 染色体上の位置 (GRCh37/hg19)
-
9p21.3 (chr9:21968228..21974826, complement)
- アミノ酸配列の長さ
-
156
遺伝子マップ

解説
CDKN2Aの座位には2種類のタンパクp16INK4Aおよびp14ARFがコードされている。p16INK4Aはサイクリン依存性キナーゼ (CDK)阻害分子であり、CDK4/6のサイクリンDへの結合を阻害する。そのため、サイクリンD-CDK4/6複合体によるRBのリン酸化が抑制され、RBによる転写制御因子E2Fの阻害が引き起こされる。その結果、E2Fの制御下にあるG1期からS期への移行に関与する遺伝子群の発現が抑制され細胞周期の進行が停滞する。一方で、p14ARFはMDM2によるTP53の分解を阻害することで、TP53の細胞周期の停止機構やアポトーシスの誘導を促進かつ維持する働きを有する。上記の通り、CDKN2Aの座位より作られるp16INK4Aおよびp14ARFは共にがん抑制遺伝子としての機能を有している。腫瘍における遺伝子変化はがん種横断的に検出されており、欠失や短縮型の遺伝子変化がその大部分を占める。生殖細胞系列における遺伝子変化は、常染色体優性遺伝の疾患である膵臓がん・悪性黒色腫症候群 (家族性異型多発母斑黒色腫症候群, FAMMMPC)や家族性膵癌 (FPC)と関連することが知られている。CDKN2Aは同じくCDK4/6の抑制に働くCDK阻害分子であるCDKN2B (p15INK5B)と近接して存在する。
シグナル伝達経路

遺伝子変異総量(Tumor Mutation Burden)の分布TMB

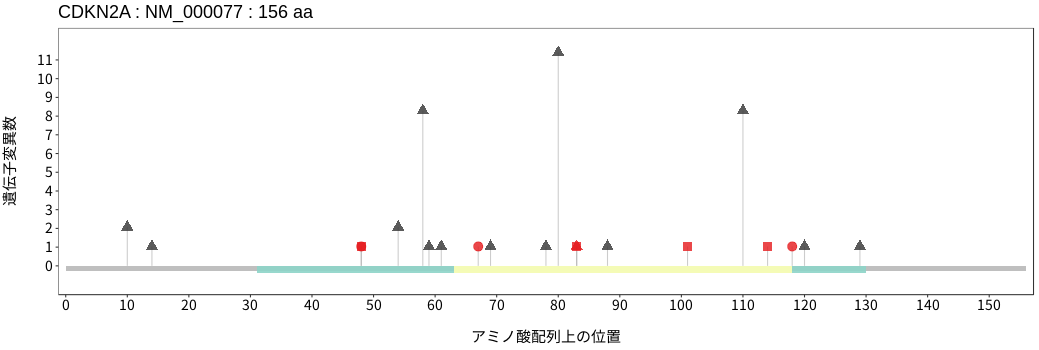
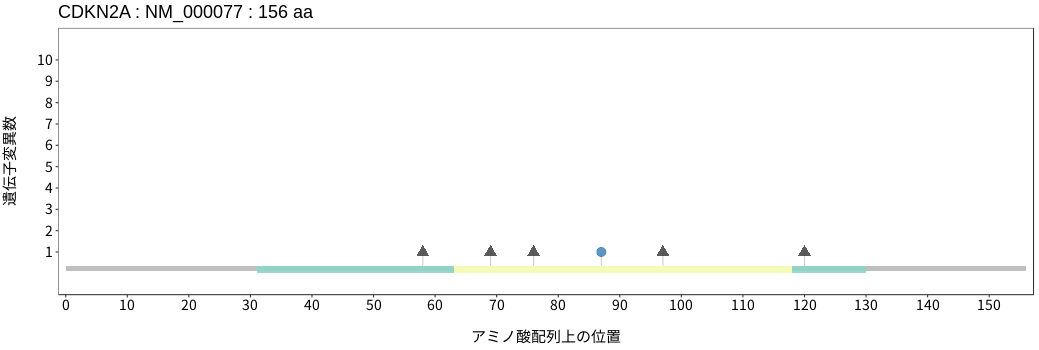
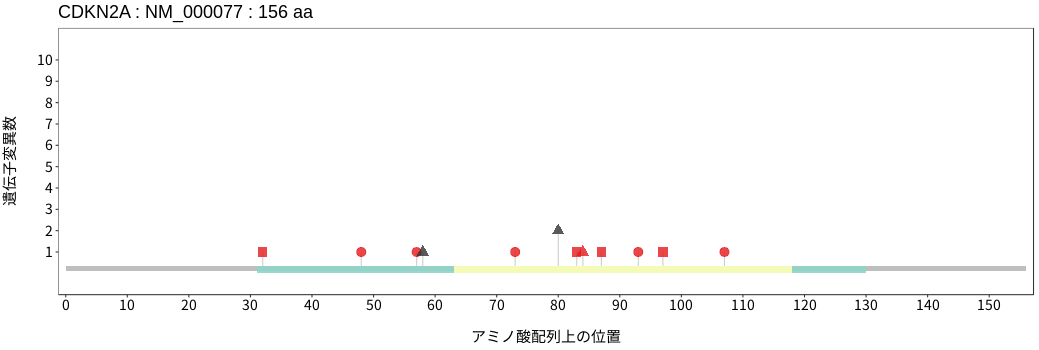
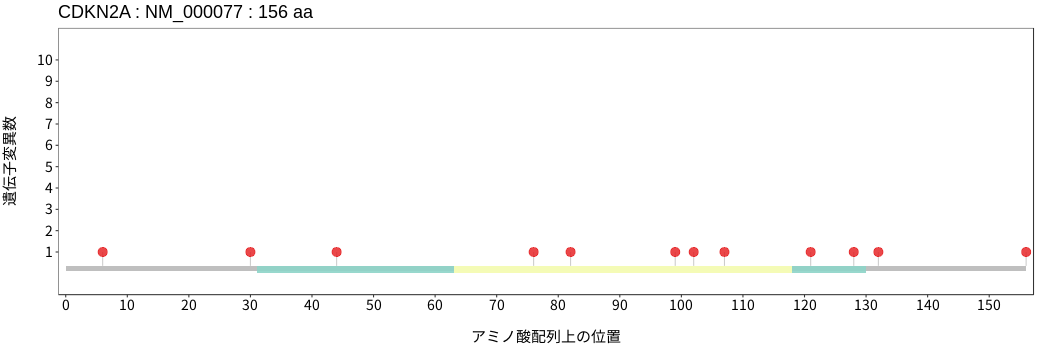
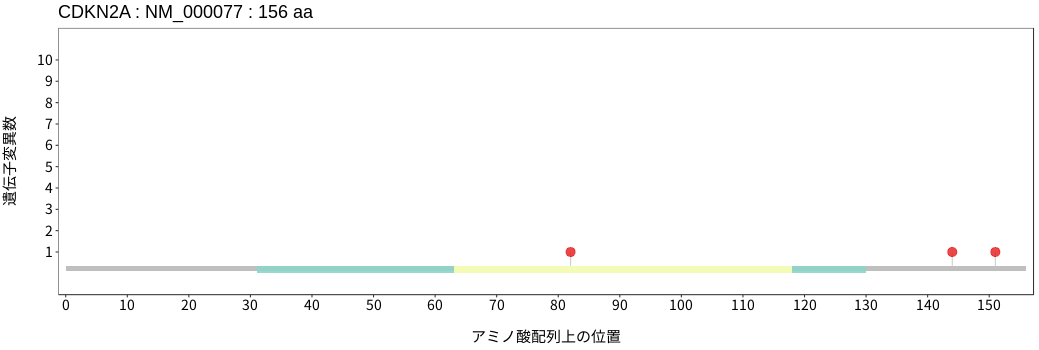
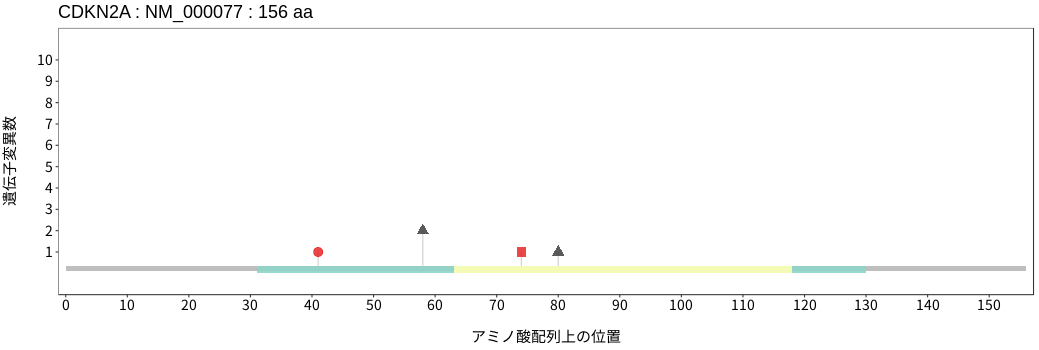
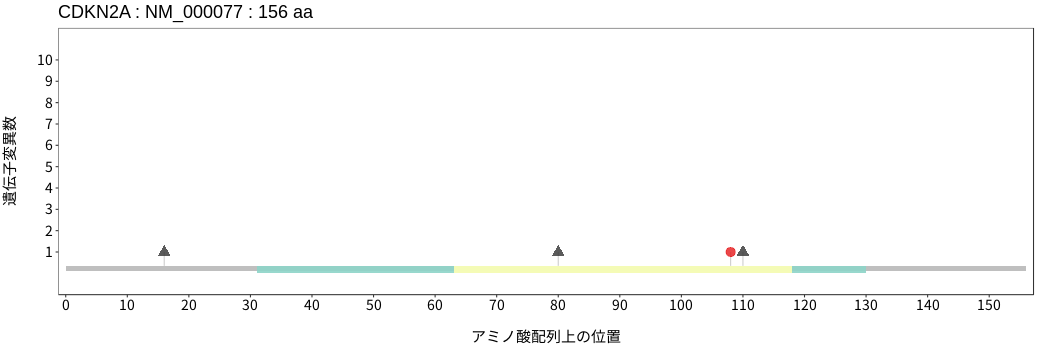
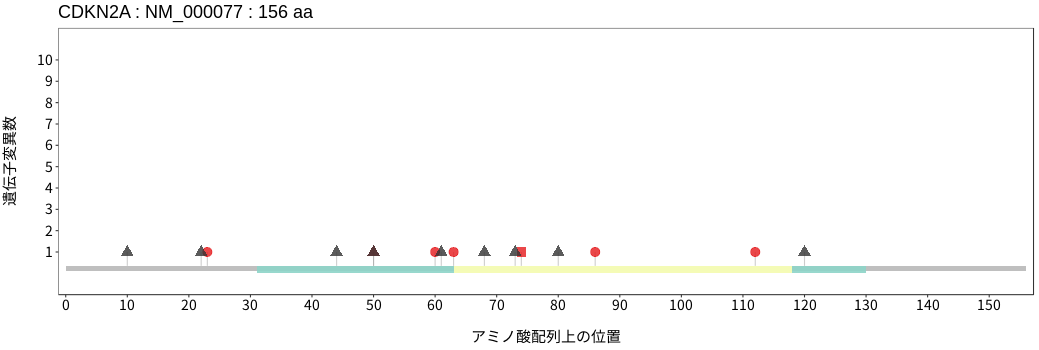
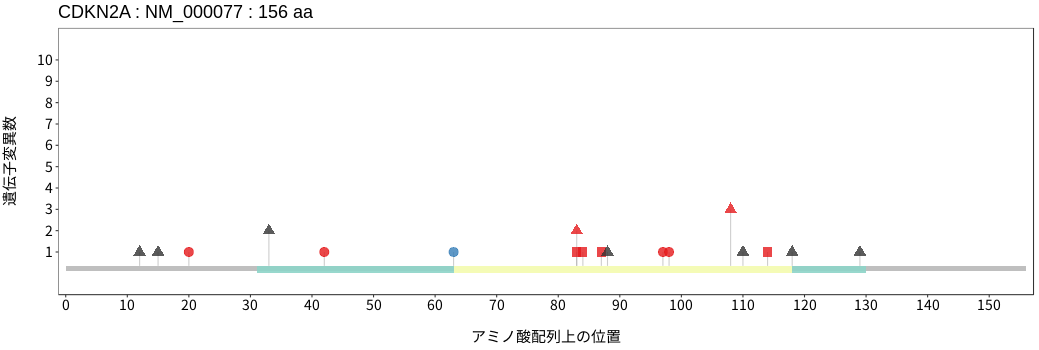
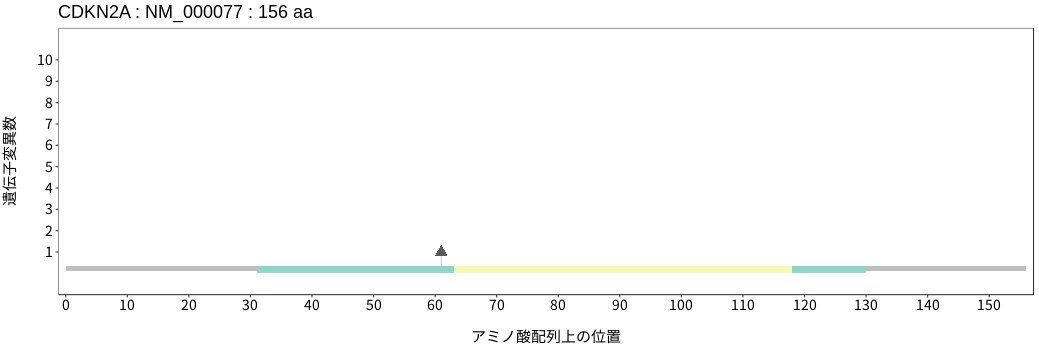
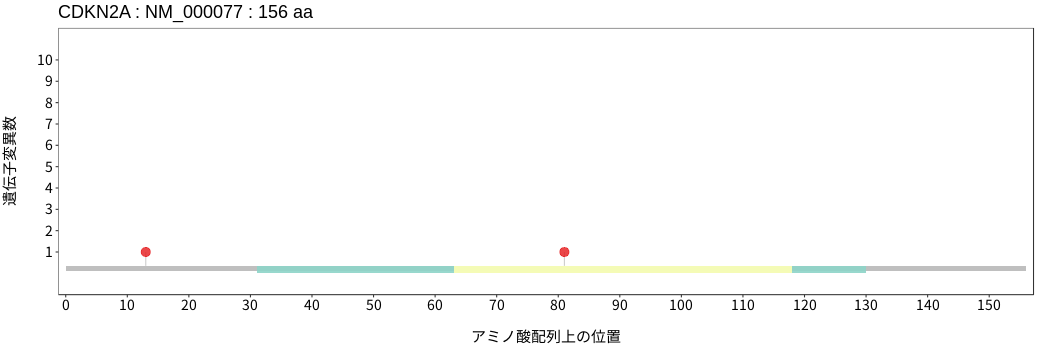
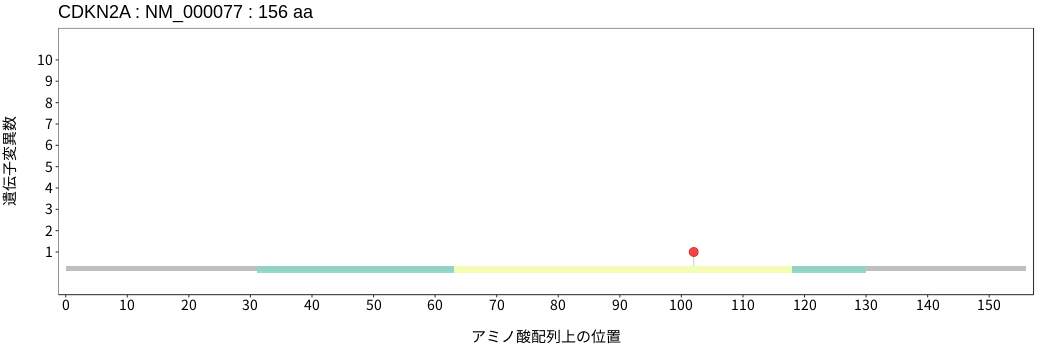
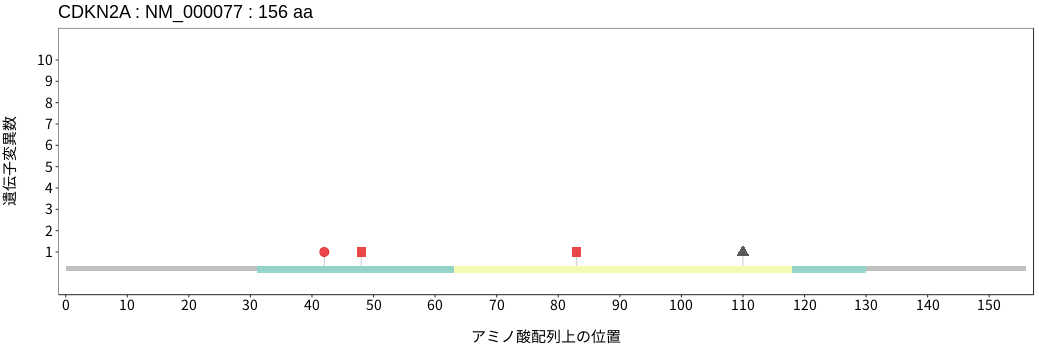
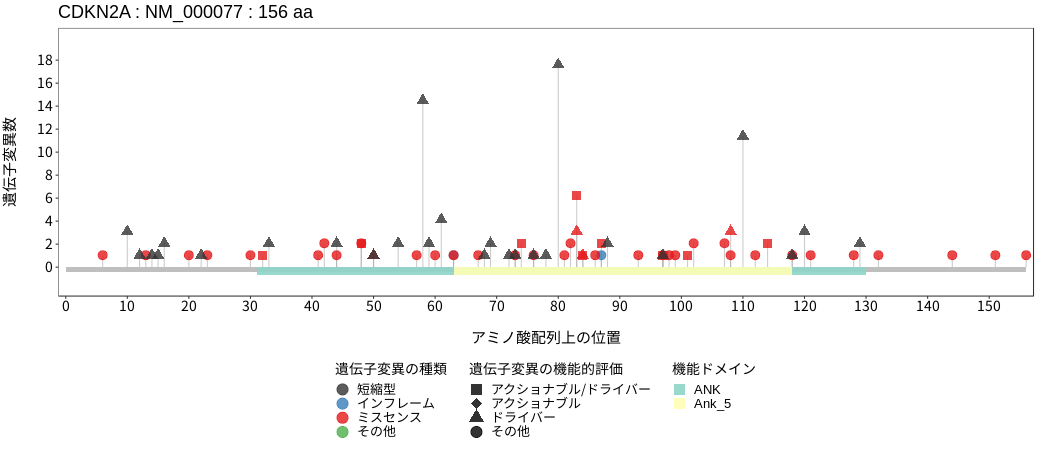
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr9:21974677 | C | A | 1 / 3 | p.Q50H | c.150G>T | COSV58692109 | Tier2 | 1 / 159 |
| chr9:21974679 | G | A | 1 / 3 | p.Q50* | c.148C>T | COSV58683786 | Tier1 | 1 / 159 |
| chr9:21974684 | G | A | 1 / 3 | p.P48L | c.143C>T | COSV58683273 | Tier1 | 2 / 159 |
| chr9:21974695 | G | C | 1 / 3 | p.Y44* | c.132C>G | COSV58696346 | Tier2 | 2 / 159 |
| chr9:21974730 | C | A | 1 / 3 | p.E33* | c.97G>T | COSV58682729 | Tier2 | 2 / 159 |
| chr9:21974732 | A | G | 1 / 3 | p.L32P | c.95T>C | COSV58685103 | Tier1 | 1 / 159 |
| chr9:21974735 | AGCGCCCGCACCTCCTCTACCCGACCCCGGGCC | A | 1 / 3 | p.R22fs | c.60_91delGGCCCGGGGTCGGGTAGAGGAGGTGCGGGCGC | Tier2 | 1 / 159 | |
| chr9:21974759 | CCCCGGGCCGCGGCCGTGGCCAG | C | 1 / 3 | p.L16fs | c.46_67delCTGGCCACGGCCGCGGCCCGGG | Tier2 | 1 / 159 | |
| chr9:21974776 | GGCCA | G | 1 / 3 | p.L16fs | c.47_50delTGGC | COSV58683946 | Tier2 | 1 / 159 |
| chr9:21974782 | C | T | 1 / 3 | p.W15* | c.45G>A | COSV58683123 | Tier2 | 1 / 159 |
| chr9:21974787 | C | CT | 1 / 3 | p.D14fs | c.39_40insA | Tier2 | 1 / 159 | |
| chr9:21974792 | G | T | 1 / 3 | p.S12* | c.35C>A | COSV58689693 | Tier2 | 1 / 159 |
| chr9:21974799 | C | A | 1 / 3 | p.E10* | c.28G>T | COSV58705987 | Tier2 | 3 / 159 |
| chr9:21970971 | G | T | 2 / 3 | p.Y129* | c.387C>A | COSV58684005 | Tier2 | 2 / 159 |
| chr9:21971000 | C | A | 2 / 3 | p.E120* | c.358G>T | COSV58683444 | Tier2 | 3 / 159 |
| chr9:21971005 | GC | G | 2 / 3 | p.A118fs | c.352delG | Tier2 | 1 / 159 | |
| chr9:21971017 | G | A | 2 / 3 | p.P114L | c.341C>T | COSV58683051 | Tier1 | 2 / 159 |
| chr9:21971028 | C | T | 2 / 3 | p.W110* | c.330G>A | COSV58682827 | Tier2 | 7 / 159 |
| chr9:21971029 | C | T | 2 / 3 | p.W110* | c.329G>A | COSV58682976 | Tier2 | 4 / 159 |
| chr9:21971036 | C | G | 2 / 3 | p.D108H | c.322G>C | COSV58690035 | Tier2 | 2 / 159 |
| chr9:21971036 | C | A | 2 / 3 | p.D108Y | c.322G>T | COSV58682998 | Tier2 | 1 / 159 |
| chr9:21971057 | C | A | 2 / 3 | p.G101W | c.301G>T | COSV58692144 | Tier1 | 1 / 159 |
| chr9:21971068 | A | C | 2 / 3 | p.L97R | c.290T>G | COSV58684740 | Tier1 | 1 / 159 |
| chr9:21971069 | G | GCACC | 2 / 3 | p.L97fs | c.285_288dupGGTG | Tier2 | 1 / 159 | |
| chr9:21971096 | C | A | 2 / 3 | p.E88* | c.262G>T | COSV58683071 | Tier2 | 2 / 159 |
| chr9:21971099 | G | A | 2 / 3 | p.R87W | c.259C>T | COSV58718452 | Tier1 | 2 / 159 |
| chr9:21971108 | C | A | 2 / 3 | p.D84Y | c.250G>T | COSV58683210 | Tier1 | 1 / 159 |
| chr9:21971108 | C | T | 2 / 3 | p.D84N | c.250G>A | COSV58683289 | Tier2 | 1 / 159 |
| chr9:21971110 | T | C | 2 / 3 | p.H83R | c.248A>G | COSV58689512 | Tier2 | 2 / 159 |
| chr9:21971111 | G | A | 2 / 3 | p.H83Y | c.247C>T | COSV58682852 | Tier1 | 6 / 159 |
| chr9:21971111 | G | C | 2 / 3 | p.H83D | c.247C>G | COSV58690009 | Tier2 | 1 / 159 |
| chr9:21971118 | TC | T | 2 / 3 | p.R80fs | c.239delG | COSV58729341 | Tier2 | 1 / 159 |
| chr9:21971120 | G | A | 2 / 3 | p.R80* | c.238C>T | COSV58682746 | Tier2 | 16 / 159 |
| chr9:21971123 | TGA | T | 2 / 3 | p.L78fs | c.233_234delTC | COSV58687173 | Tier2 | 1 / 159 |
| chr9:21971127 | AGTGG | A | 2 / 3 | p.A76fs | c.227_230delCCAC | Tier2 | 1 / 159 | |
| chr9:21971138 | C | T | 2 / 3 | p.D74N | c.220G>A | COSV58688841 | Tier1 | 2 / 159 |
| chr9:21971139 | GGC | G | 2 / 3 | p.A73fs | c.217_218delGC | Tier2 | 1 / 159 | |
| chr9:21971142 | G | T | 2 / 3 | p.C72* | c.216C>A | COSV58683179 | Tier2 | 1 / 159 |
| chr9:21971150 | GC | G | 2 / 3 | p.E69fs | c.207delG | Tier2 | 1 / 159 | |
| chr9:21971150 | GCT | G | 2 / 3 | p.E69fs | c.206_207delAG | Tier2 | 1 / 159 | |
| chr9:21971155 | GC | G | 2 / 3 | p.A68fs | c.202delG | Tier2 | 1 / 159 | |
| chr9:21971174 | GCTCCGCCACT | G | 2 / 3 | p.V59fs | c.174_183delAGTGGCGGAG | Tier2 | 1 / 159 | |
| chr9:21971177 | C | A | 2 / 3 | p.E61* | c.181G>T | COSV58683250 | Tier2 | 4 / 159 |
| chr9:21971179 | GCCACT | G | 2 / 3 | p.V59fs | c.174_178delAGTGG | COSV58693461 | Tier2 | 1 / 159 |
| chr9:21971186 | G | A | 2 / 3 | p.R58* | c.172C>T | COSV58682666 | Tier2 | 14 / 159 |
| chr9:21971197 | A | ATCAT | 2 / 3 | p.M54fs | c.157_160dupATGA | Tier2 | 1 / 159 | |
| chr9:21971198 | T | TCA | 2 / 3 | p.M54fs | c.158_159dupTG | Tier2 | 1 / 159 |