Side Menu
CTNNB1
基本情報
- 遺伝子名
-
catenin beta 1
- 慣用名
-
CTNNB, EVR7, MRD19, NEDSDV, armadillo
- 遺伝子分類
-
がん遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
分化
- シグナル伝達経路
-
WNT
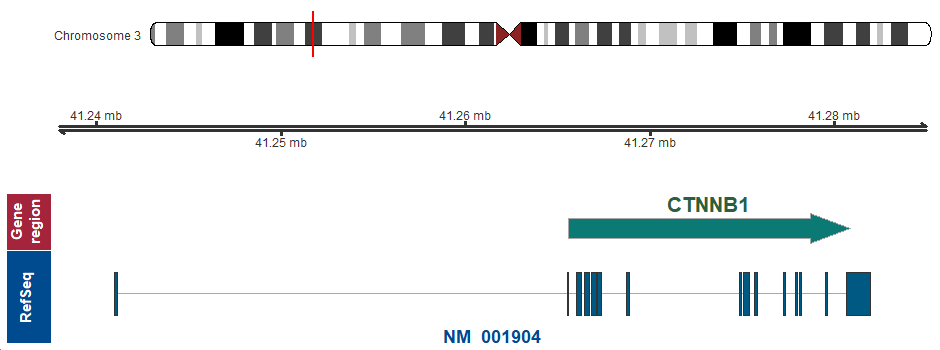
- 染色体上の位置 (GRCh37/hg19)
-
3p22.1 (chr3:41265560..41280833)
- アミノ酸配列の長さ
-
781
遺伝子マップ
解説
CTNNB1 (β-カテニン)はWNTシグナル伝達経路におけるメディエーターであり、TCF /LEFファミリーの転写制御因子と相互作用することで、標的遺伝子の転写の調節に働く。WNTリガンド非存在下では、APC、AXINそしてGSK3βなどの分子によって構成されるβ-カテニン分解複合体により細胞質に隔離され、ユビキチン化の後に分解される。WNTリガンド存在下では、β-カテニン分解複合体が解体され、β-カテニンは核に移行し機能する。βカテニン-TCF/LEF複合体により発現誘導される代表的な遺伝子としてサイクリンD1およびMYCが知られており、両分子とも細胞周期の進行を亢進させる。
シグナル伝達経路

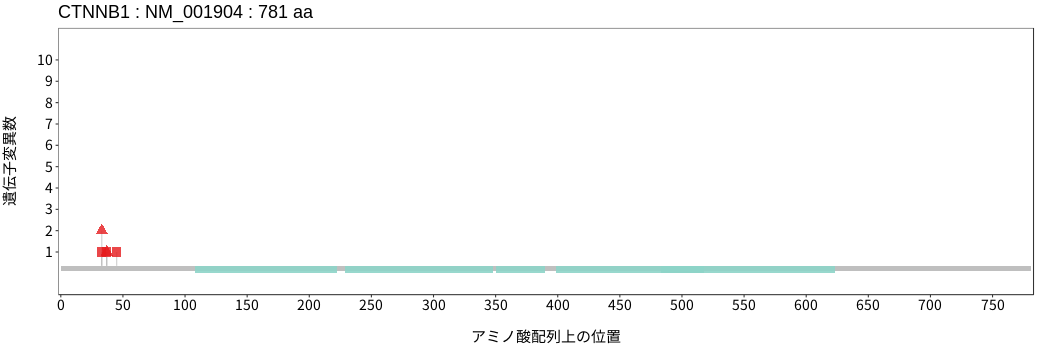
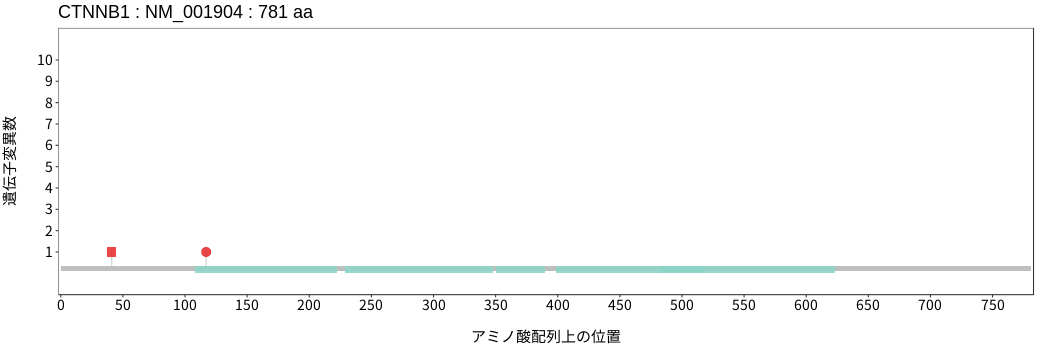
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

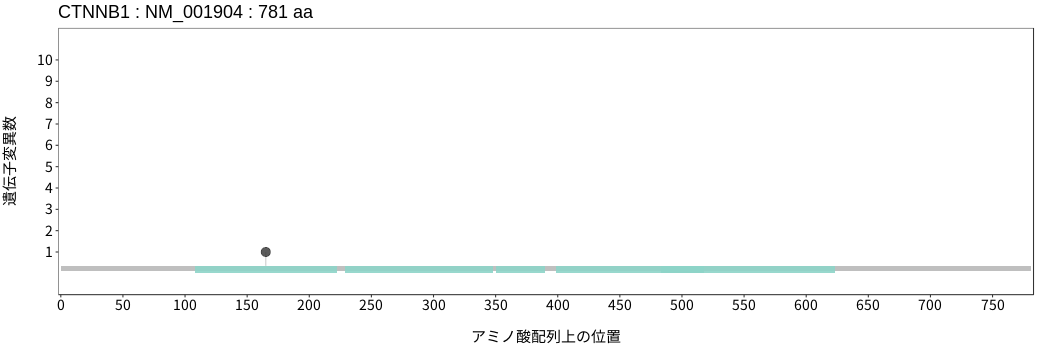
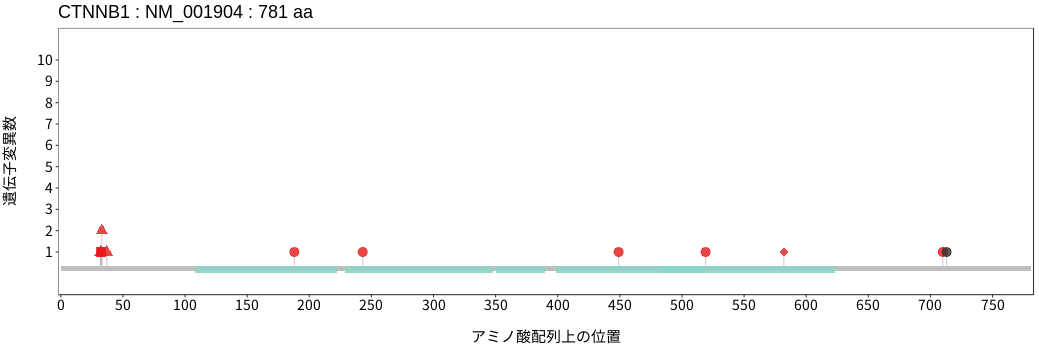
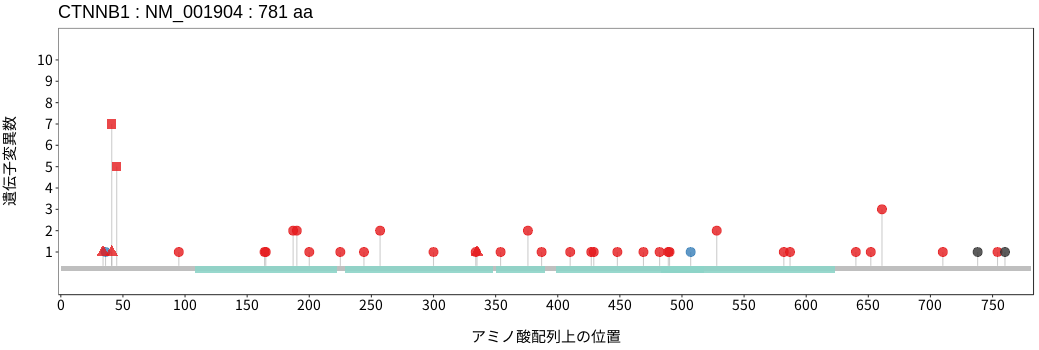
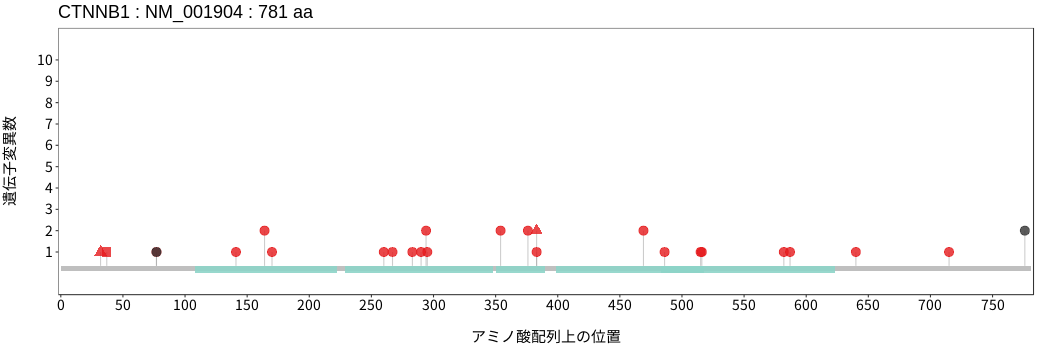
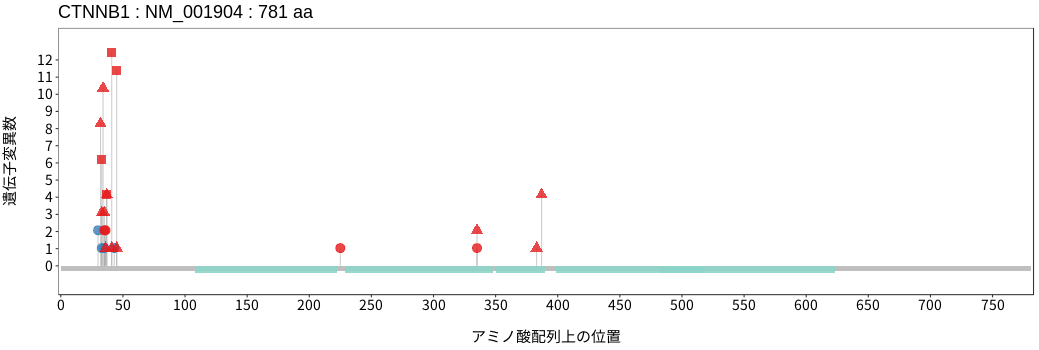
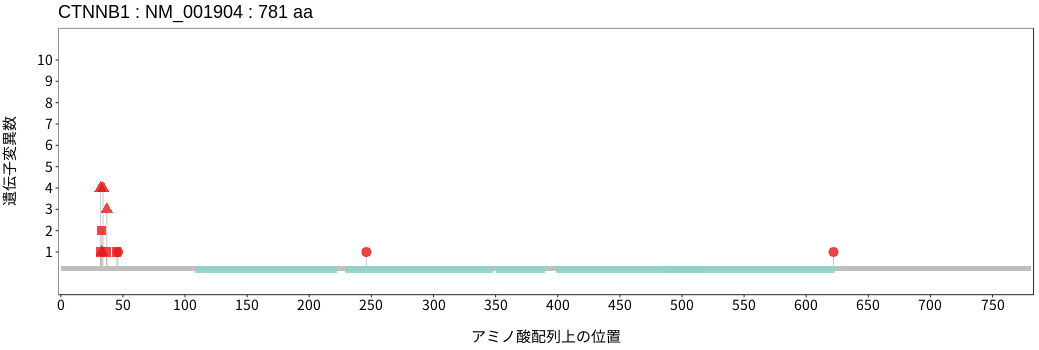
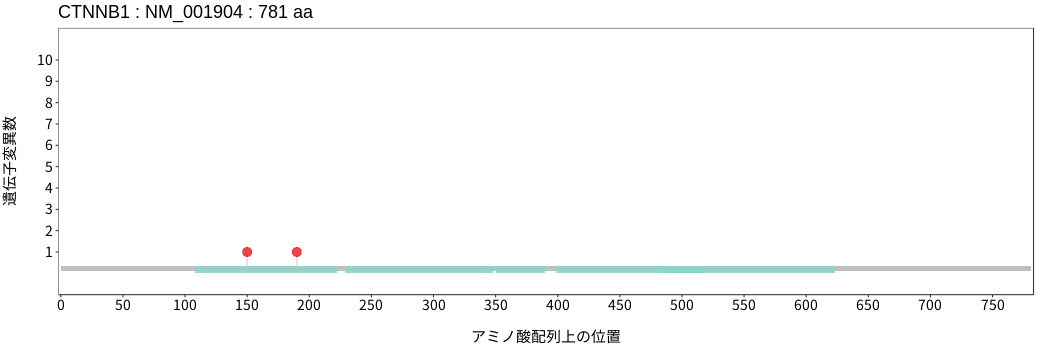
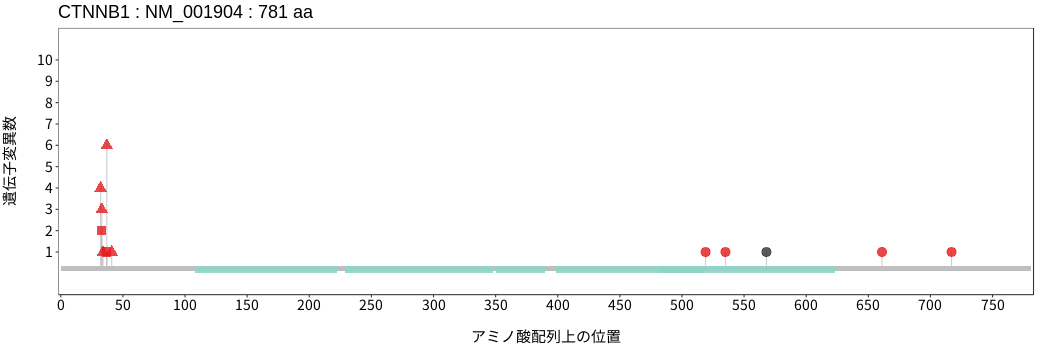
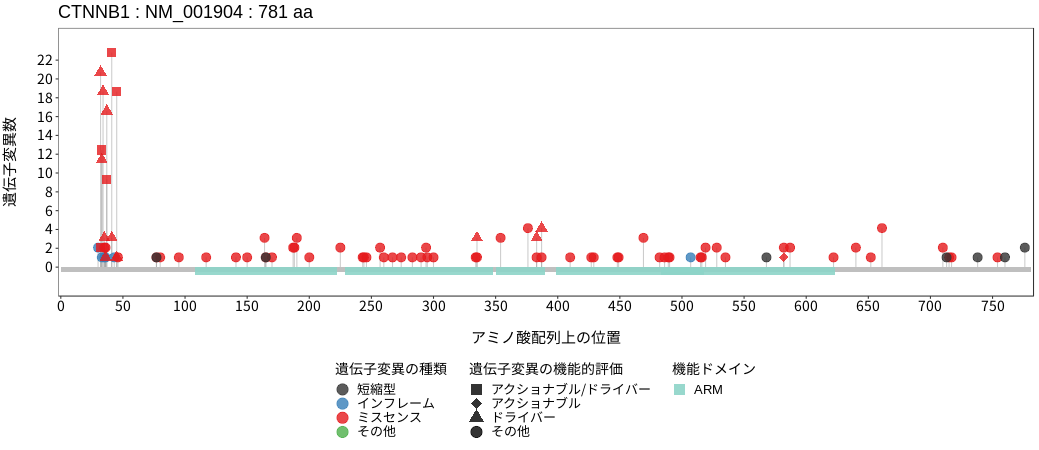
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr3:41266097 | G | A | 3 / 15 | p.D32N | c.94G>A | COSV62687956 | Tier2 | 2 / 249 |
| chr3:41266097 | G | C | 3 / 15 | p.D32H | c.94G>C | COSV62688665 | Tier2 | 2 / 249 |
| chr3:41266097 | G | T | 3 / 15 | p.D32Y | c.94G>T | COSV62688134 | Tier1 | 9 / 249 |
| chr3:41266098 | A | G | 3 / 15 | p.D32G | c.95A>G | COSV62688001 | Tier2 | 5 / 249 |
| chr3:41266098 | A | C | 3 / 15 | p.D32A | c.95A>C | COSV62688037 | Tier2 | 2 / 249 |
| chr3:41266098 | A | T | 3 / 15 | p.D32V | c.95A>T | COSV62688249 | Tier2 | 2 / 249 |
| chr3:41266100 | T | G | 3 / 15 | p.S33A | c.97T>G | COSV62689225 | Tier1 | 2 / 249 |
| chr3:41266100 | T | C | 3 / 15 | p.S33P | c.97T>C | COSV62688086 | Tier2 | 2 / 249 |
| chr3:41266101 | C | G | 3 / 15 | p.S33C | c.98C>G | COSV62687839 | Tier1 | 9 / 249 |
| chr3:41266101 | C | T | 3 / 15 | p.S33F | c.98C>T | COSV62688300 | Tier1 | 9 / 249 |
| chr3:41266101 | C | A | 3 / 15 | p.S33Y | c.98C>A | COSV62688644 | Tier1 | 1 / 249 |
| chr3:41266103 | G | A | 3 / 15 | p.G34R | c.100G>A | COSV62687911 | Tier2 | 6 / 249 |
| chr3:41266103 | G | C | 3 / 15 | p.G34R | c.100G>C | COSV62687931 | Tier2 | 2 / 249 |
| chr3:41266104 | G | T | 3 / 15 | p.G34V | c.101G>T | COSV62687976 | Tier1 | 5 / 249 |
| chr3:41266104 | G | A | 3 / 15 | p.G34E | c.101G>A | COSV62688267 | Tier2 | 5 / 249 |
| chr3:41266107 | T | G | 3 / 15 | p.I35S | c.104T>G | COSV62687967 | Tier2 | 3 / 249 |
| chr3:41266110 | A | C | 3 / 15 | p.H36P | c.107A>C | COSV62688989 | Tier2 | 1 / 249 |
| chr3:41266112 | T | C | 3 / 15 | p.S37P | c.109T>C | COSV62688537 | Tier2 | 4 / 249 |
| chr3:41266112 | T | G | 3 / 15 | p.S37A | c.109T>G | COSV62688488 | Tier2 | 1 / 249 |
| chr3:41266113 | C | T | 3 / 15 | p.S37F | c.110C>T | COSV62687856 | Tier1 | 11 / 249 |
| chr3:41266113 | C | A | 3 / 15 | p.S37Y | c.110C>A | COSV62689256 | Tier1 | 4 / 249 |
| chr3:41266113 | C | G | 3 / 15 | p.S37C | c.110C>G | COSV62688281 | Tier1 | 5 / 249 |
| chr3:41266124 | A | G | 3 / 15 | p.T41A | c.121A>G | COSV62687862 | Tier1 | 22 / 249 |
| chr3:41266125 | C | T | 3 / 15 | p.T41I | c.122C>T | COSV62688008 | Tier2 | 3 / 249 |
| chr3:41266136 | T | C | 3 / 15 | p.S45P | c.133T>C | COSV62687880 | Tier1 | 8 / 249 |
| chr3:41266137 | C | T | 3 / 15 | p.S45F | c.134C>T | COSV62687872 | Tier1 | 10 / 249 |
| chr3:41266137 | C | A | 3 / 15 | p.S45Y | c.134C>A | COSV62689938 | Tier1 | 1 / 249 |
| chr3:41268766 | A | T | 7 / 15 | p.K335I | c.1004A>T | COSV62688046 | Tier2 | 3 / 249 |
| chr3:41274897 | T | C | 8 / 15 | p.W383R | c.1147T>C | COSV62701352 | Tier2 | 2 / 249 |
| chr3:41274897 | T | A | 8 / 15 | p.W383R | c.1147T>A | COSV62690170 | Tier2 | 1 / 249 |
| chr3:41274911 | T | A | 8 / 15 | p.N387K | c.1161T>A | COSV62688055 | Tier2 | 2 / 249 |
| chr3:41274911 | T | G | 8 / 15 | p.N387K | c.1161T>G | COSV62689041 | Tier2 | 2 / 249 |