Side Menu
EGFR
基本情報
- 遺伝子名
-
epidermal growth factor receptor
- 慣用名
-
ERBB, ERBB1, HER1, NISBD2, PIG61, mENA
- 遺伝子分類
-
がん遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
腫瘍形成・増殖
- シグナル伝達経路
-
RTK
- 染色体上の位置 (GRCh37/hg19)
-
7p11.2 (chr7:55086971..55273310)
- アミノ酸配列の長さ
-
1,210
遺伝子マップ
解説
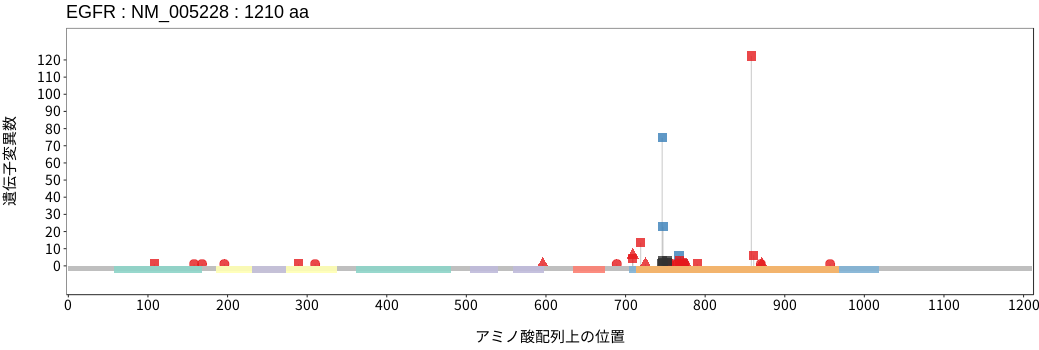
EGFRはERBBファミリーに属する受容体チロシンキナーゼである。EGF, TGFα, amphiregulin,そしてHeparin-binding EGF-like Growth Factor (HB-EGF)などのリガンドの結合により二量体化し、その二分子間での相互リン酸化(自己リン酸化)が細胞質内ドメインにおいて起きることで活性化する。活性化EGFRは、GRB2およびSOS1を細胞膜に動員し、RASの活性化を介して下流のMAPKおよびPI3K/Akt/mTORシグナル経路を活性化させることで細胞増殖、遊走、血管新生、代謝および分化の制御に関与する。体細胞バリアントは、肺腺癌および膠芽腫において高い頻度で認められる。肺腺癌において検出される遺伝子変化の大部分は細胞質内に位置するキナーゼドメインが含まれるエクソン18~21に存在する。特に、エクソン19領域のインフレームの欠失やインフレームの挿入欠失、そしてエクソン21のミスセンスバリアントL858Rの頻度が高く、これらバリアントはEGFRの恒常的な活性化を引き起こすドライバーバリアントであり、EGFR阻害剤に対し高い感受性を示すことが知られている。一方で、エクソン20領域のインフレームの挿入 (重複)およびミスセンスバリアントT790MはEGFR阻害剤への感受性の低下と関連することが知られている。膠芽腫においてはエクソン6~7の細胞外領域のバリアントや、染色体再構成により生じるエクソン2~7を欠失したEGFRvIII (EGFR variant III)と呼ばれる遺伝子変化が認められる。増幅は主に肺腺癌、膠芽腫そして頭頚部扁平上皮癌において認められる。生殖細胞系列バリアントは、家族性肺癌との関連性が少数例報告されている。
国内承認薬 (分子標的治療薬)
2022/06/10 更新
| 一般名 (販売名) | 外部サイトへのリンク | |
|---|---|---|
| ゲフィチニブ(イレッサ) | PMDA | KEGG DRUG |
| セツキシマブ(アービタックス) | PMDA | KEGG DRUG |
| エルロチニブ(タルセバ) | PMDA | KEGG DRUG |
| パニツムマブ(ベクティビックス) | PMDA | KEGG DRUG |
| ラパチニブ(タイケルブ)※ | PMDA | KEGG DRUG |
| バンデタニブ(カプレルサ)※ | PMDA | KEGG DRUG |
| アファチニブ(ジオトリフ)※ | PMDA | KEGG DRUG |
| オシメルチニブ(タグリッソ) | PMDA | KEGG DRUG |
| ネシツムマブ(ポートラーザ) | PMDA | KEGG DRUG |
| ダコミチニブ(ビジンプロ)※ | PMDA | KEGG DRUG |
| セツキシマブ サロタロカンナトリウム (アキャルックス) |
PMDA | KEGG DRUG |
※JCGAに掲載されている460遺伝子の中に複数の標的遺伝子がある薬剤 (他の遺伝子においても当該薬剤が表記されています)
薬剤の標的とされる遺伝子において該当する薬剤名を列挙しています。
各薬剤の承認情報(適応がん種、効果・効能の情報)を反映したものではありません。
国内承認薬に関する情報の詳細は、独立行政法人 医薬品医療機器総合機構(PMDA)が公開している情報を必ず確認してください。
薬剤の標的とされる遺伝子において該当する薬剤名を列挙しています。
各薬剤の承認情報(適応がん種、効果・効能の情報)を反映したものではありません。
国内承認薬に関する情報の詳細は、独立行政法人 医薬品医療機器総合機構(PMDA)が公開している情報を必ず確認してください。
シグナル伝達経路

遺伝子変異総量(Tumor Mutation Burden)の分布TMB

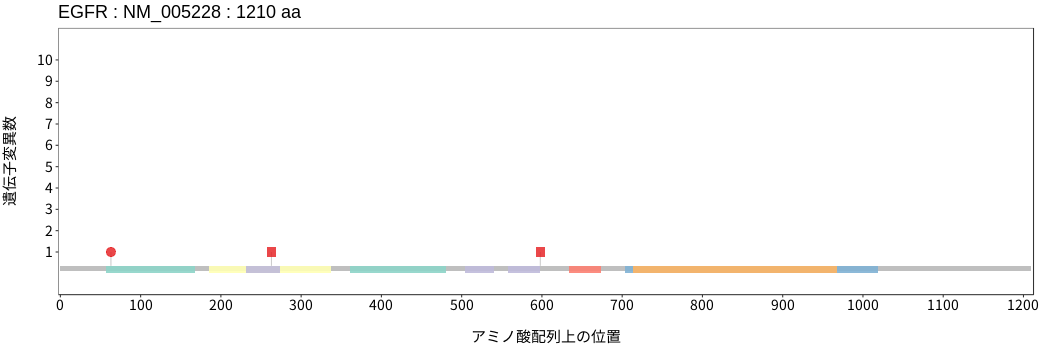
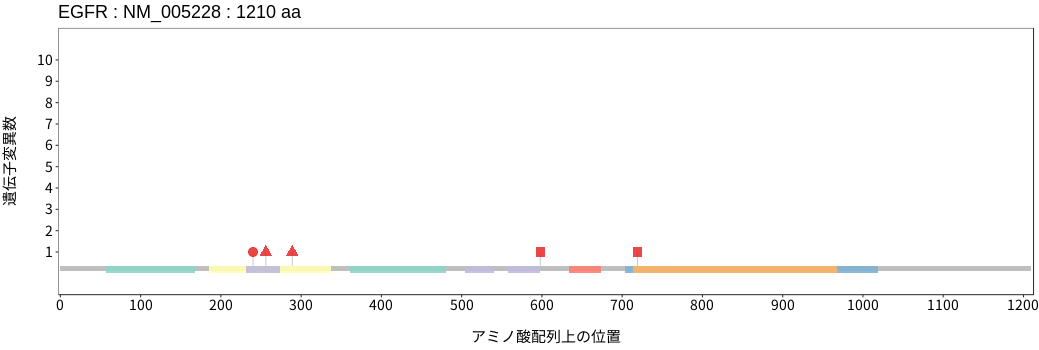
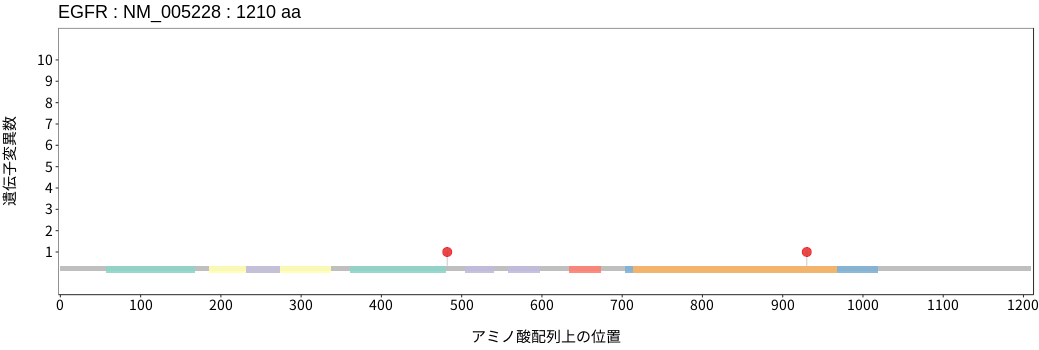
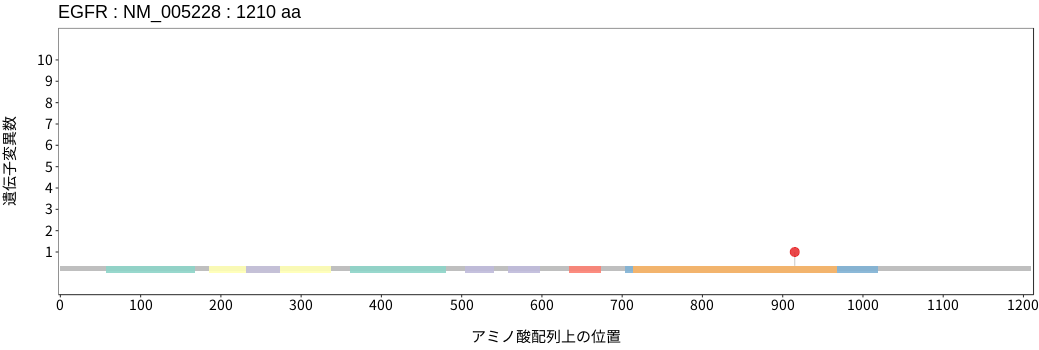
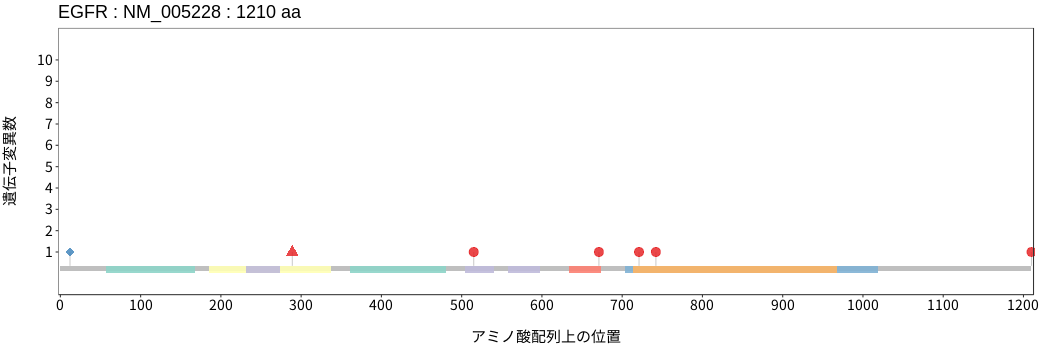
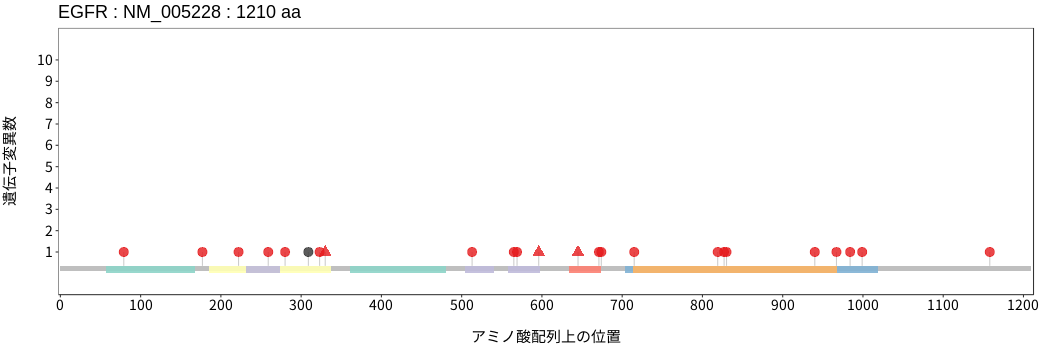
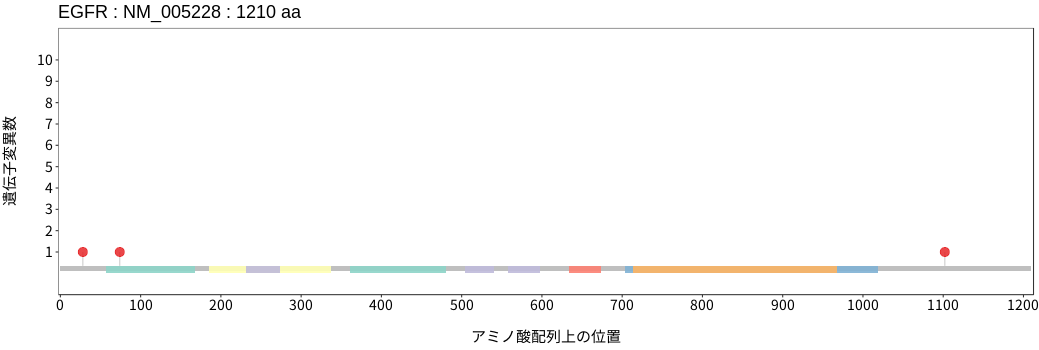
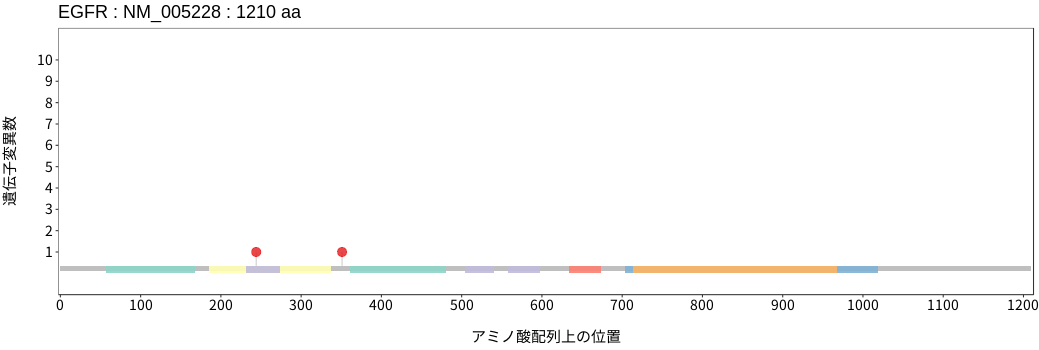
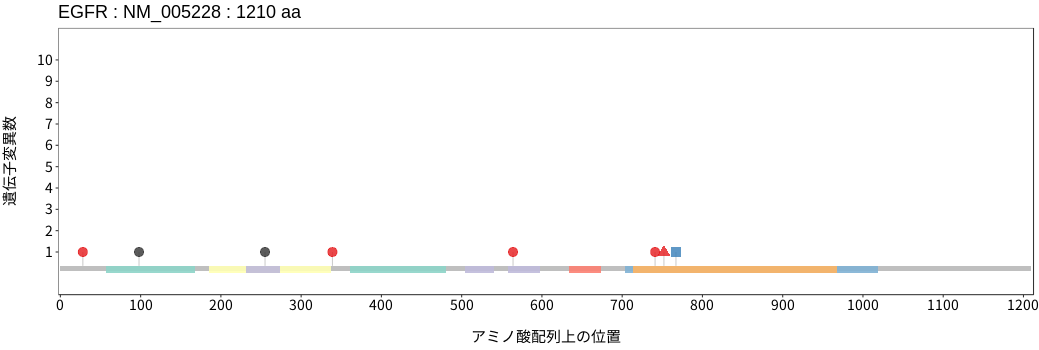
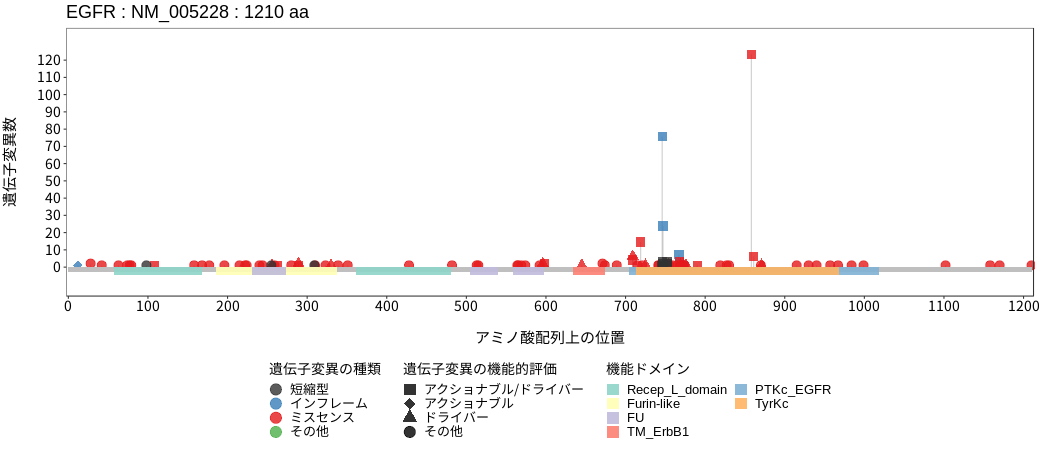
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr7:55211080 | G | A | 3 / 28 | p.R108K | c.323G>A | COSV51766433 | Tier1 | 1 / 355 |
| chr7:55221722 | G | T | 7 / 28 | p.D256Y | c.766G>T | COSV51855357 | Tier2 | 1 / 355 |
| chr7:55221743 | A | C | 7 / 28 | p.T263P | c.787A>C | COSV51768130 | Tier1 | 1 / 355 |
| chr7:55221821 | G | A | 7 / 28 | p.A289T | c.865G>A | COSV51770322 | Tier2 | 1 / 355 |
| chr7:55221822 | C | A | 7 / 28 | p.A289D | c.866C>A | COSV51768139 | Tier2 | 1 / 355 |
| chr7:55221822 | C | T | 7 / 28 | p.A289V | c.866C>T | COSV51765841 | Tier1 | 1 / 355 |
| chr7:55223621 | G | A | 8 / 28 | p.E330K | c.988G>A | COSV51831642 | Tier1 | 1 / 355 |
| chr7:55233037 | C | T | 15 / 28 | p.P596L | c.1787C>T | COSV51779162 | Tier2 | 2 / 355 |
| chr7:55233043 | G | T | 15 / 28 | p.G598V | c.1793G>T | COSV51769031 | Tier1 | 2 / 355 |
| chr7:55240690 | C | G | 17 / 28 | p.S645C | c.1934C>G | COSV51772147 | Tier2 | 1 / 355 |
| chr7:55241677 | G | A | 18 / 28 | p.E709K | c.2125G>A | COSV51767260 | Tier1 | 4 / 355 |
| chr7:55241678 | A | C | 18 / 28 | p.E709A | c.2126A>C | COSV51772956 | Tier1 | 2 / 355 |
| chr7:55241678 | A | T | 18 / 28 | p.E709V | c.2126A>T | COSV51766584 | Tier2 | 1 / 355 |
| chr7:55241678 | A | G | 18 / 28 | p.E709G | c.2126A>G | COSV51765252 | Tier2 | 3 / 355 |
| chr7:55241707 | G | A | 18 / 28 | p.G719S | c.2155G>A | COSV51767289 | Tier1 | 2 / 355 |
| chr7:55241707 | G | T | 18 / 28 | p.G719C | c.2155G>T | COSV51766606 | Tier1 | 1 / 355 |
| chr7:55241708 | G | C | 18 / 28 | p.G719A | c.2156G>C | COSV51769339 | Tier1 | 10 / 355 |
| chr7:55241708 | G | A | 18 / 28 | p.G719D | c.2156G>A | COSV51779524 | Tier2 | 1 / 355 |
| chr7:55241726 | C | T | 18 / 28 | p.T725M | c.2174C>T | COSV51779794 | Tier2 | 1 / 355 |
| chr7:55242462 | CAAGGAATTAAGAG | C | 19 / 28 | p.E746fs | c.2236_2248delGAATTAAGAGAAG | Tier1 | 1 / 355 | |
| chr7:55242463 | AAGGAATTAAGAGAAGCAAC | A | 19 / 28 | p.K745fs | c.2235_2253delGGAATTAAGAGAAGCAACA | Tier1 | 1 / 355 | |
| chr7:55242464 | AGGAATTAAGAGAAGC | A | 19 / 28 | p.E746_A750del | c.2235_2249delGGAATTAAGAGAAGC | COSV51765119 | Tier1 | 48 / 355 |
| chr7:55242464 | AGGAATTAAGAGAAGCAAC | A | 19 / 28 | p.E746_T751del | c.2235_2252delGGAATTAAGAGAAGCAAC | COSV51810835 | Tier1 | 1 / 355 |
| chr7:55242465 | GGAATTAAGAGAAGCA | G | 19 / 28 | p.E746_A750del | c.2236_2250delGAATTAAGAGAAGCA | COSV51765066 | Tier1 | 23 / 355 |
| chr7:55242466 | GAATTAAGAGAAGC | G | 19 / 28 | p.L747fs | c.2239_2251delTTAAGAGAAGCAA | Tier1 | 1 / 355 | |
| chr7:55242466 | GAATTAAGAGAAGCAACA | G | 19 / 28 | p.E746fs | c.2237_2253delAATTAAGAGAAGCAACA | Tier1 | 2 / 355 | |
| chr7:55242466 | GAATTAAGAGAAGCAA | G | 19 / 28 | p.E746_T751delinsA | c.2237_2251delAATTAAGAGAAGCAA | COSV51769364 | Tier1 | 1 / 355 |
| chr7:55242467 | AATTAAGAGAAGC | A | 19 / 28 | p.L747_A750del | c.2239_2250delTTAAGAGAAGCA | COSV99289022 | Tier1 | 3 / 355 |
| chr7:55242467 | AATTAAGAGAAGCAAC | A | 19 / 28 | p.L747_T751del | c.2240_2254delTAAGAGAAGCAACAT | COSV51766247 | Tier1 | 8 / 355 |
| chr7:55242468 | ATTAAGAGAAG | A | 19 / 28 | p.L747fs | c.2239_2248delTTAAGAGAAG | Tier1 | 1 / 355 | |
| chr7:55242469 | TTAAGAGAAGCAACATCTC | T | 19 / 28 | p.L747_P753delinsS | c.2240_2257delTAAGAGAAGCAACATCTC | COSV51767961 | Tier1 | 12 / 355 |
| chr7:55242481 | ACATCT | A | 19 / 28 | p.S752fs | c.2253_2257delATCTC | COSV51857889 | Tier1 | 1 / 355 |
| chr7:55242482 | CATCTCCGAAAGCCAACAAGGAAAT | C | 19 / 28 | p.S752_I759del | c.2253_2276delATCTCCGAAAGCCAACAAGGAAAT | COSV51766231 | Tier1 | 2 / 355 |
| chr7:55242483 | ATCTCC | A | 19 / 28 | p.S752fs | c.2254_2258delTCTCC | Tier1 | 1 / 355 | |
| chr7:55242484 | TCTCCGAAA | T | 19 / 28 | p.S752fs | c.2255_2262delCTCCGAAA | Tier1 | 1 / 355 | |
| chr7:55242485 | C | A | 19 / 28 | p.S752Y | c.2255C>A | COSV51866106 | Tier2 | 1 / 355 |
| chr7:55242486 | TC | T | 19 / 28 | p.P753fs | c.2258delC | Tier1 | 2 / 355 | |
| chr7:55242490 | A | G | 19 / 28 | p.K754E | c.2260A>G | COSV51830809 | Tier2 | 1 / 355 |
| chr7:55248998 | A | ATGGCCAGCG | 20 / 28 | p.A767_V769dup | c.2300_2308dupCCAGCGTGG | COSV51766549 | Tier1 | 7 / 355 |
| chr7:55249002 | C | CCAGCGTGGA | 20 / 28 | p.S768_D770dup | c.2303_2311dupGCGTGGACA | COSV51774688 | Tier1 | 2 / 355 |
| chr7:55249005 | G | T | 20 / 28 | p.S768I | c.2303G>T | COSV51768106 | Tier1 | 3 / 355 |
| chr7:55249007 | G | A | 20 / 28 | p.V769M | c.2305G>A | COSV51802272 | Tier2 | 1 / 355 |
| chr7:55249010 | G | GACAACCCCC | 20 / 28 | p.N771_H773dup | c.2311_2319dupAACCCCCAC | COSV51772596 | Tier1 | 1 / 355 |
| chr7:55249011 | A | ACAACCCCCC | 20 / 28 | p.P772_H773insPNP | c.2317_2318insCCAACCCCC | Tier1 | 1 / 355 | |
| chr7:55249017 | C | CCCA | 20 / 28 | p.H773dup | c.2317_2319dupCAC | COSV51781591 | Tier1 | 1 / 355 |
| chr7:55249020 | A | T | 20 / 28 | p.H773L | c.2318A>T | COSV51792805 | Tier1 | 1 / 355 |
| chr7:55249029 | G | A | 20 / 28 | p.R776H | c.2327G>A | COSV51780730 | Tier2 | 1 / 355 |
| chr7:55249071 | C | T | 20 / 28 | p.T790M | c.2369C>T | COSV51765492 | Tier1 | 1 / 355 |
| chr7:55259515 | T | G | 21 / 28 | p.L858R | c.2573T>G | COSV51765161 | Tier1 | 119 / 355 |
| chr7:55259524 | T | A | 21 / 28 | p.L861Q | c.2582T>A | COSV51766344 | Tier1 | 6 / 355 |
| chr7:55259554 | C | G | 21 / 28 | p.A871G | c.2612C>G | COSV51774247 | Tier2 | 1 / 355 |