Side Menu
FAT1
基本情報
- 遺伝子名
-
FAT atypical cadherin 1
- 慣用名
-
CDHF7, CDHR8, FAT, ME5, hFat1
- 遺伝子分類
-
がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
分化
- シグナル伝達経路
-
WNT
- 染色体上の位置 (GRCh37/hg19)
-
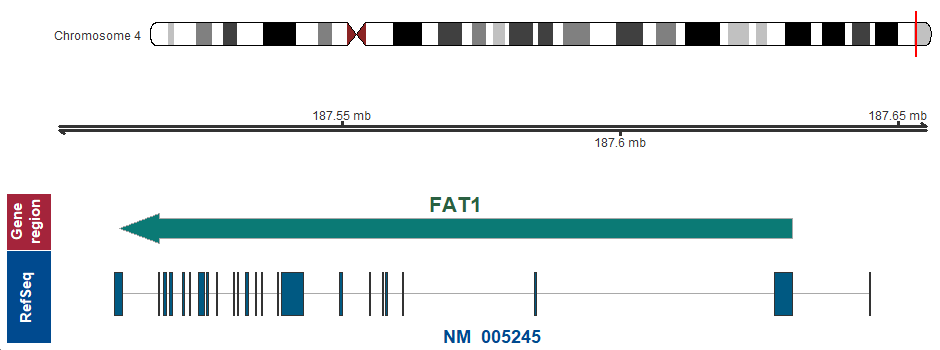
4q35.2 (chr4:187509746..187630981, complement)
- アミノ酸配列の長さ
-
4,588
遺伝子マップ
解説
FAT1は、ショウジョウバエ脂肪遺伝子のオルソログであり、ショウジョウバエ発生過程の細胞増殖の制御に不可欠な腫瘍抑制因子をコードする。この遺伝子は、カドヘリン型リピートの存在が特徴の内在性膜タンパク質群であるカドヘリンスーパーファミリーで、34のタンデムなカドヘリン型リピートに加え、5つの上皮成長因子(EGF)様リピートおよび1つのラミニンA-Gドメインを有する。この遺伝子は多数の胎児上皮で多量に発現する。その産物は、接着分子および/またはシグナル伝達受容体として機能し、発生過程および細胞伝達に重要であると思われる。
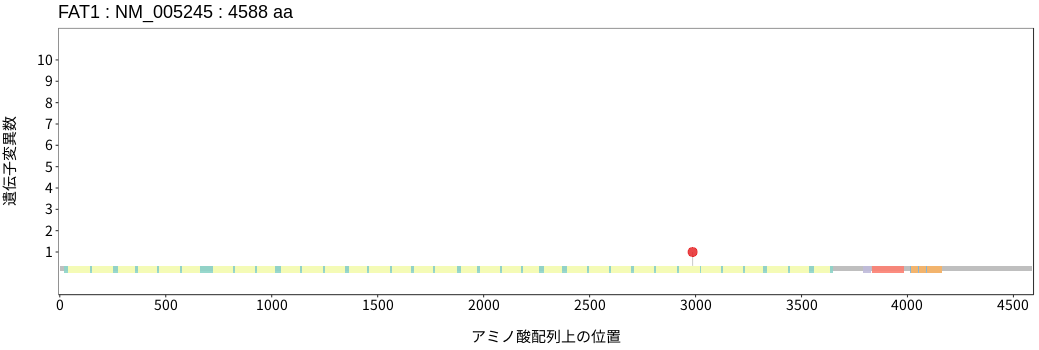
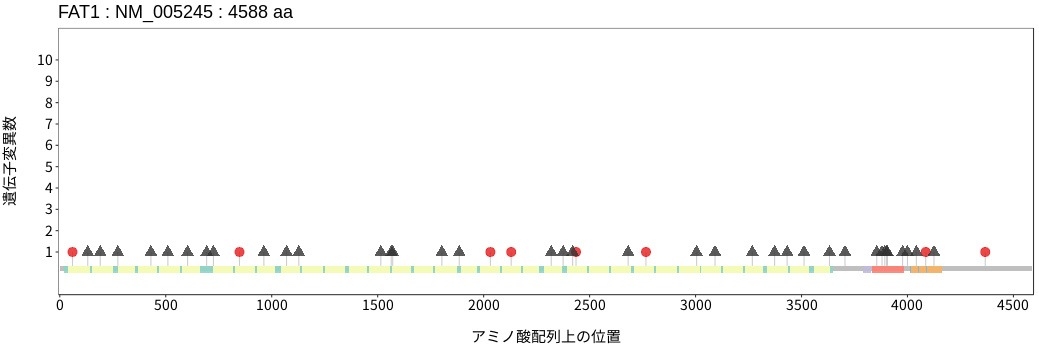
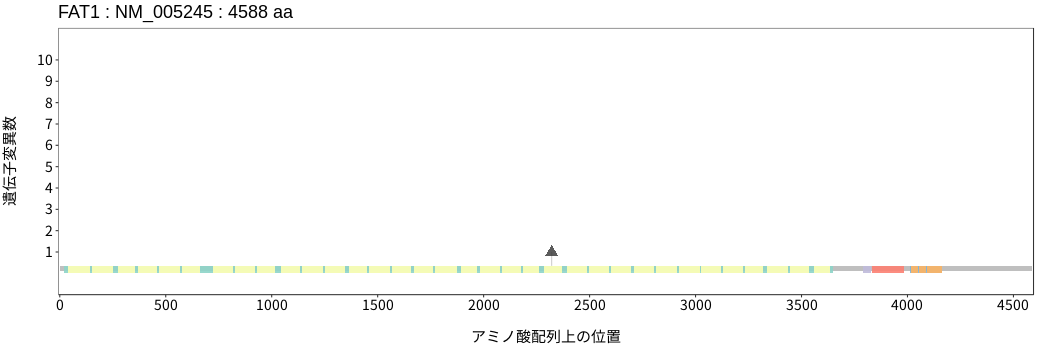
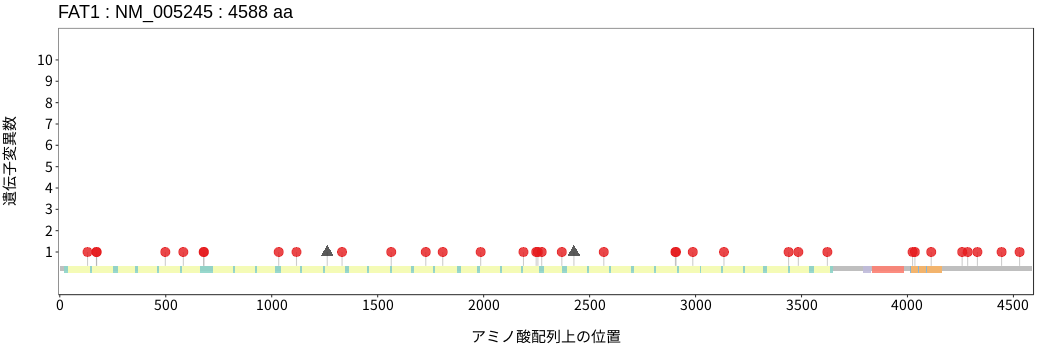
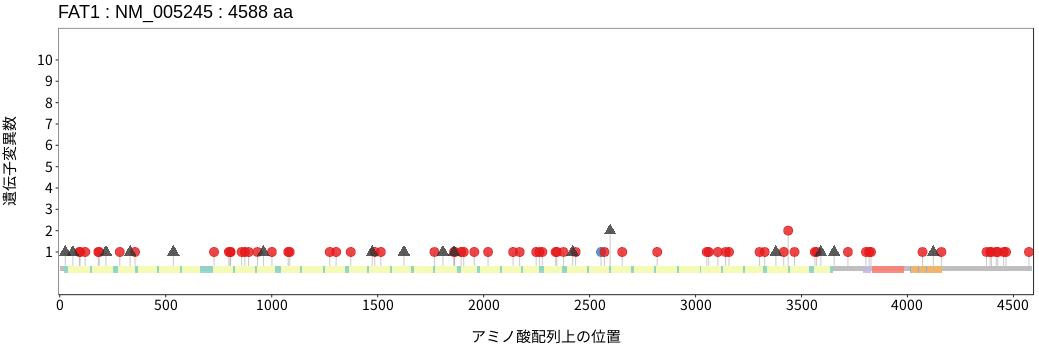
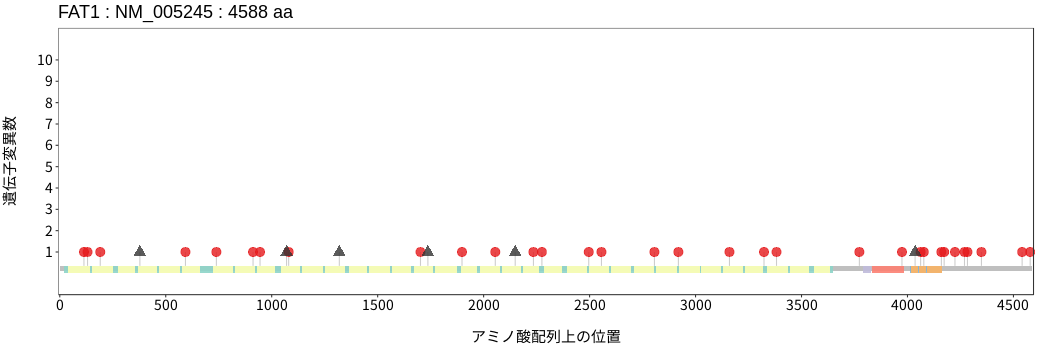
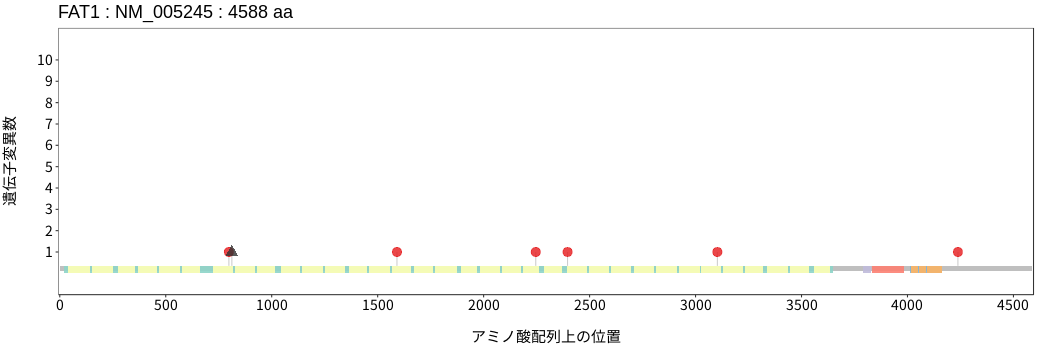
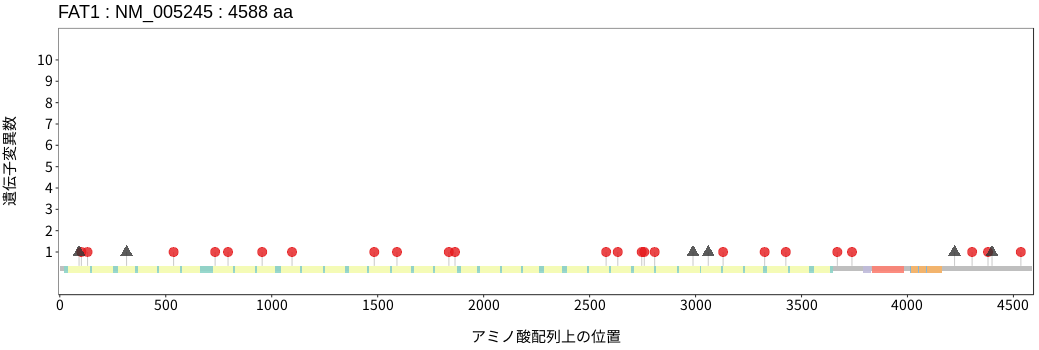
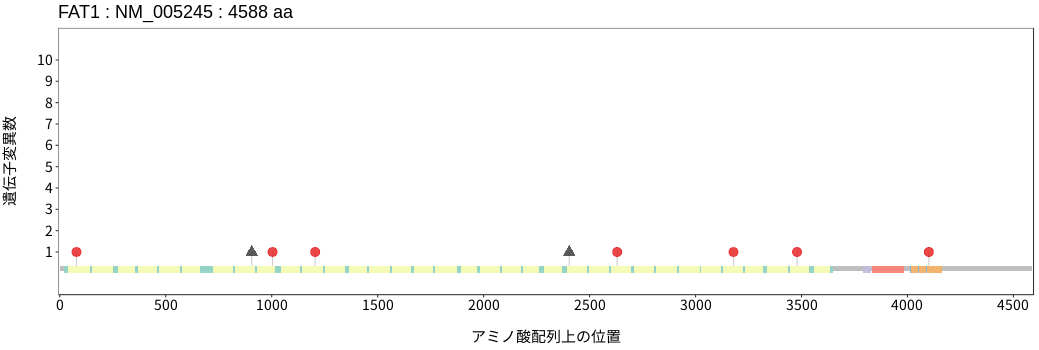
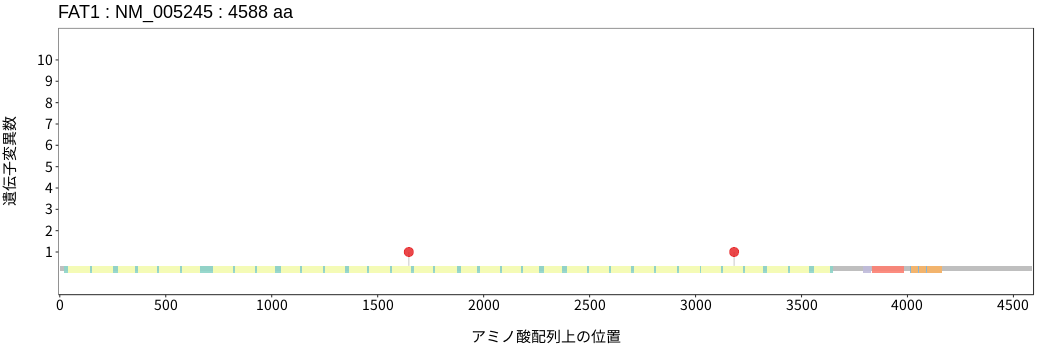
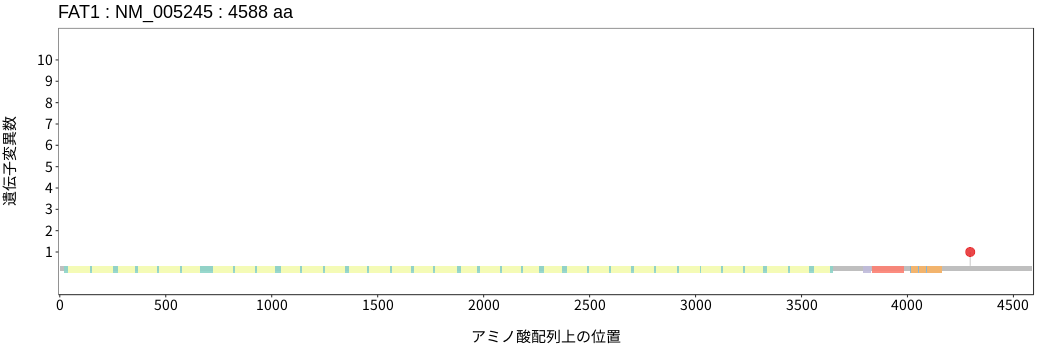
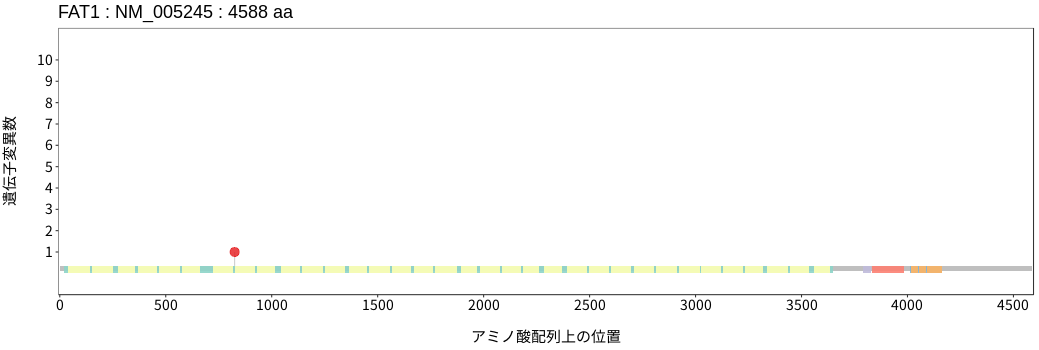
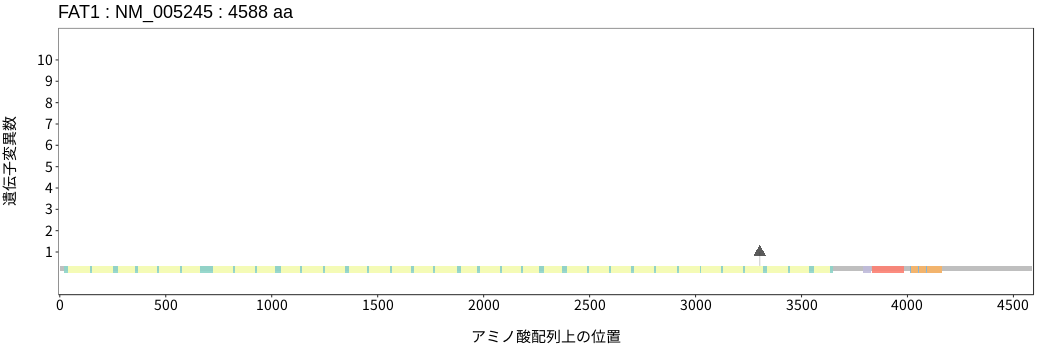
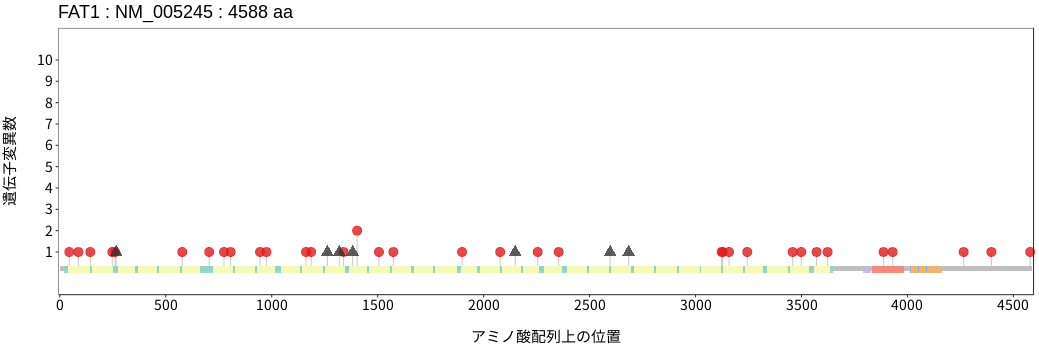
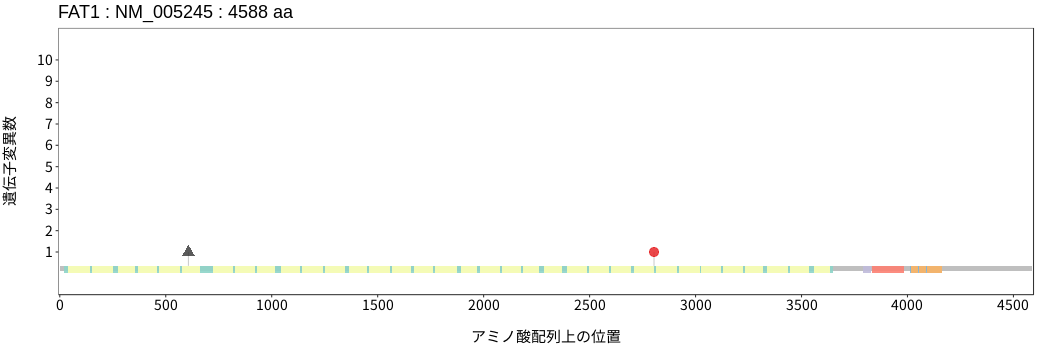
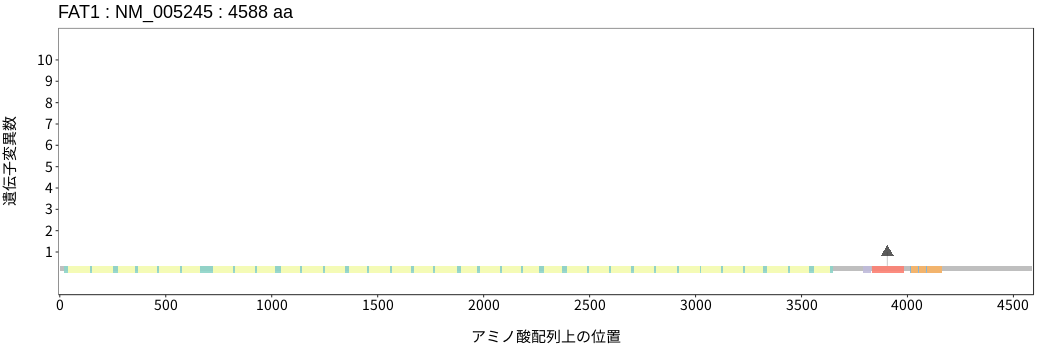
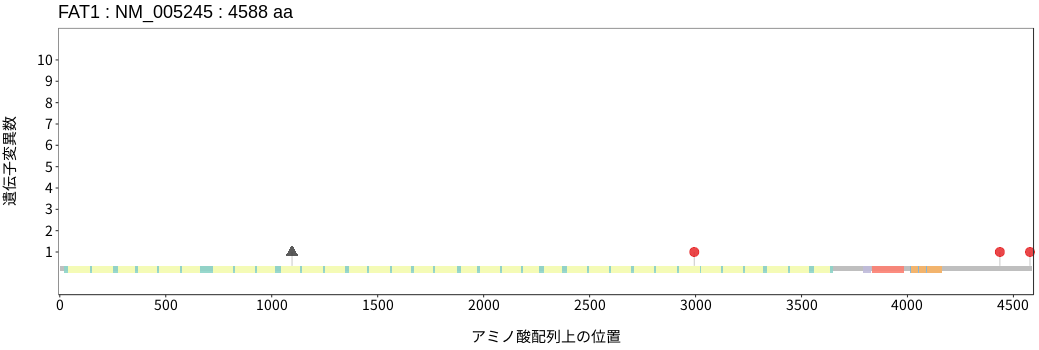
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

アミノ酸配列上の変異分布
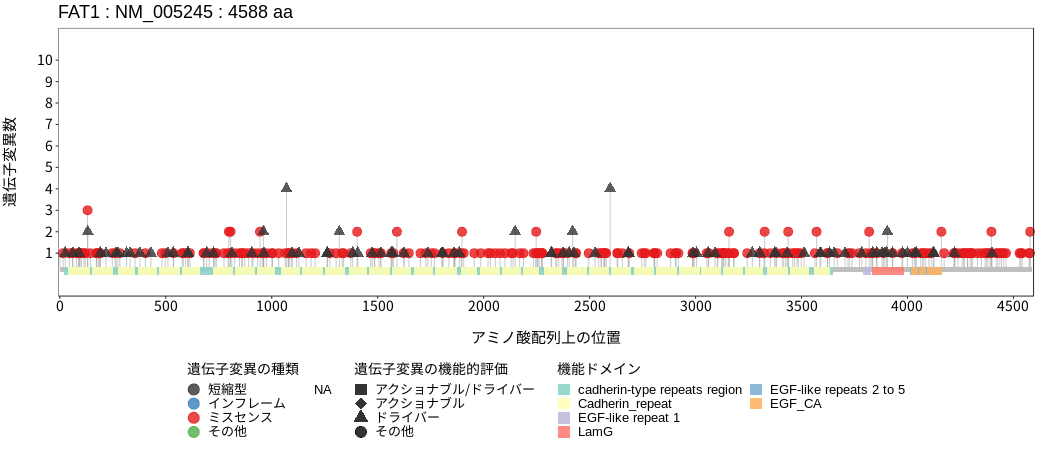
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
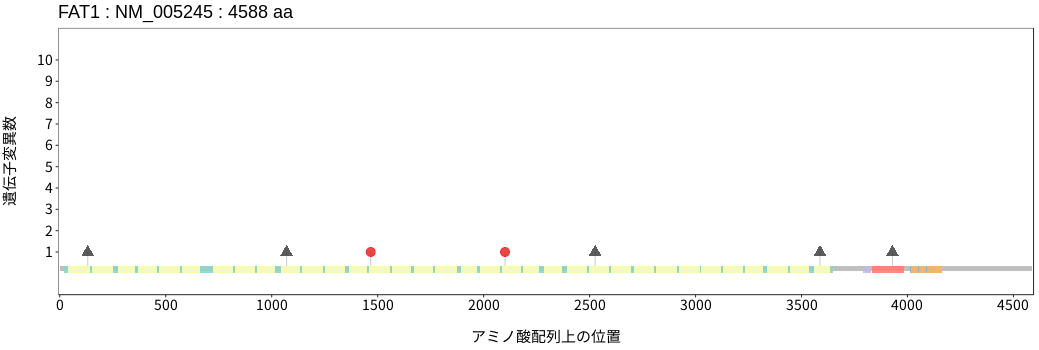
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr4:187627774 | G | A | 2 / 27 | p.R1070* | c.3208C>T | COSV71674659 | Tier2 | 4 / 310 |
| chr4:187628095 | T | A | 2 / 27 | p.R963* | c.2887A>T | Tier2 | 1 / 310 | |
| chr4:187628101 | G | A | 2 / 27 | p.Q961* | c.2881C>T | COSV101498593 | Tier2 | 2 / 310 |
| chr4:187628266 | G | A | 2 / 27 | p.Q906* | c.2716C>T | COSV71675913 | Tier2 | 1 / 310 |
| chr4:187628547 | AC | A | 2 / 27 | p.V812fs | c.2434delG | Tier2 | 1 / 310 | |
| chr4:187628809 | G | A | 2 / 27 | p.Q725* | c.2173C>T | COSV71675113 | Tier2 | 1 / 310 |
| chr4:187628904 | A | C | 2 / 27 | p.L693* | c.2078T>G | Tier2 | 1 / 310 | |
| chr4:187629163 | C | A | 2 / 27 | p.E607* | c.1819G>T | Tier2 | 1 / 310 | |
| chr4:187629175 | G | A | 2 / 27 | p.Q603* | c.1807C>T | Tier2 | 1 / 310 | |
| chr4:187629373 | TCA | T | 2 / 27 | p.L536fs | c.1607_1608delTG | Tier2 | 1 / 310 | |
| chr4:187629454 | AC | A | 2 / 27 | p.F510fs | c.1527delG | Tier2 | 1 / 310 | |
| chr4:187629687 | GCTGCC | G | 2 / 27 | p.Q430fs | c.1290_1294delGGCAG | Tier2 | 1 / 310 | |
| chr4:187629850 | C | A | 2 / 27 | p.E378* | c.1132G>T | Tier2 | 1 / 310 | |
| chr4:187629986 | G | C | 2 / 27 | p.Y332* | c.996C>G | Tier2 | 1 / 310 | |
| chr4:187630037 | A | T | 2 / 27 | p.Y315* | c.945T>A | Tier2 | 1 / 310 | |
| chr4:187630162 | C | CA | 2 / 27 | p.A274fs | c.819dupT | COSV101499379 | Tier2 | 1 / 310 |
| chr4:187630186 | C | A | 2 / 27 | p.E266* | c.796G>T | Tier2 | 1 / 310 | |
| chr4:187630328 | A | C | 2 / 27 | p.Y218* | c.654T>G | Tier2 | 1 / 310 | |
| chr4:187630411 | G | A | 2 / 27 | p.R191* | c.571C>T | COSV71672617 | Tier2 | 1 / 310 |
| chr4:187630588 | G | A | 2 / 27 | p.R132* | c.394C>T | COSV71671964 | Tier2 | 2 / 310 |
| chr4:187630711 | C | A | 2 / 27 | p.G91* | c.271G>T | Tier2 | 1 / 310 | |
| chr4:187630797 | T | TAAAC | 2 / 27 | p.Y62fs | c.181_184dupGTTT | Tier2 | 1 / 310 | |
| chr4:187630906 | G | A | 2 / 27 | p.R26* | c.76C>T | Tier2 | 1 / 310 | |
| chr4:187584647 | ACCTC | A | 3 / 27 | p.E1128fs | c.3382_3385delGAGG | Tier2 | 1 / 310 | |
| chr4:187584747 | G | A | 3 / 27 | p.R1096* | c.3286C>T | COSV71672426 | Tier2 | 1 / 310 |
| chr4:187557756 | C | A | 5 / 27 | p.E1319* | c.3955G>T | Tier2 | 2 / 310 | |
| chr4:187557924 | C | A | 5 / 27 | p.E1263* | c.3787G>T | Tier2 | 1 / 310 | |
| chr4:187557927 | G | A | 5 / 27 | p.R1262* | c.3784C>T | COSV71675829(SNP) | Tier2 | 1 / 310 |
| chr4:187557215 | CCA | C | 6 / 27 | p.V1382fs | c.4145_4146delTG | Tier2 | 1 / 310 | |
| chr4:187554945 | T | A | 7 / 27 | p.K1406* | c.4216A>T | Tier2 | 1 / 310 | |
| chr4:187549689 | CAGAAGTAT | C | 8 / 27 | p.Y1515fs | c.4544_4551delATACTTCT | Tier2 | 1 / 310 | |
| chr4:187549821 | C | A | 8 / 27 | p.E1474* | c.4420G>T | Tier2 | 1 / 310 | |
| chr4:187549406 | AC | A | 9 / 27 | p.V1571fs | c.4711delG | Tier2 | 1 / 310 | |
| chr4:187549427 | G | GCGGTGAACCA | 9 / 27 | p.A1564fs | c.4681_4690dupTGGTTCACCG | Tier2 | 1 / 310 | |
| chr4:187539690 | C | A | 10 / 27 | p.E2684* | c.8050G>T | COSV71672478 | Tier2 | 1 / 310 |
| chr4:187539695 | G | GA | 10 / 27 | p.S2682fs | c.8044dupT | Tier2 | 1 / 310 | |
| chr4:187539951 | G | A | 10 / 27 | p.R2597* | c.7789C>T | COSV71672084 | Tier2 | 4 / 310 |
| chr4:187540163 | TG | T | 10 / 27 | p.H2526fs | c.7576delC | Tier2 | 1 / 310 | |
| chr4:187540464 | G | A | 10 / 27 | p.Q2426* | c.7276C>T | Tier2 | 1 / 310 | |
| chr4:187540481 | G | C | 10 / 27 | p.S2420* | c.7259C>G | Tier2 | 2 / 310 | |
| chr4:187540528 | AGG | A | 10 / 27 | p.P2404fs | c.7210_7211delCC | Tier2 | 1 / 310 | |
| chr4:187540614 | CAA | C | 10 / 27 | p.I2375fs | c.7124_7125delTT | Tier2 | 1 / 310 | |
| chr4:187540779 | G | A | 10 / 27 | p.Q2321* | c.6961C>T | COSV71675559 | Tier2 | 1 / 310 |
| chr4:187540784 | G | C | 10 / 27 | p.S2319* | c.6956C>G | COSV71674364 | Tier2 | 1 / 310 |
| chr4:187541295 | C | A | 10 / 27 | p.E2149* | c.6445G>T | COSV71671772 | Tier2 | 2 / 310 |
| chr4:187542065 | GTTGGTAACAAAAGAGATGCT | G | 10 / 27 | p.E1885fs | c.5655_5674delAGCATCTCTTTTGTTACCAA | Tier2 | 1 / 310 | |
| chr4:187542162 | C | A | 10 / 27 | p.E1860* | c.5578G>T | Tier2 | 1 / 310 | |
| chr4:187542318 | C | CA | 10 / 27 | p.V1808fs | c.5421dupT | Tier2 | 1 / 310 | |
| chr4:187542332 | G | C | 10 / 27 | p.S1803* | c.5408C>G | COSV71672193 | Tier2 | 1 / 310 |
| chr4:187542494 | GTATTAGTGGACAAACCAGCCATGTTAGTTCCTTGTAT | G | 10 / 27 | p.I1737fs | c.5209_5245delATACAAGGAACTAACATGGCTGGTTTGTCCACTAATA | Tier2 | 1 / 310 | |
| chr4:187542870 | C | A | 10 / 27 | p.E1624* | c.4870G>T | COSV101499639 | Tier2 | 1 / 310 |
| chr4:187538220 | G | C | 11 / 27 | p.S3005* | c.9014C>G | COSV71673041 | Tier2 | 1 / 310 |
| chr4:187538272 | T | A | 11 / 27 | p.K2988* | c.8962A>T | Tier2 | 1 / 310 | |
| chr4:187535394 | G | C | 12 / 27 | p.Y3060* | c.9180C>G | Tier2 | 1 / 310 | |
| chr4:187534453 | A | AG | 13 / 27 | p.V3092fs | c.9272dupC | Tier2 | 1 / 310 | |
| chr4:187532590 | G | T | 14 / 27 | p.S3268* | c.9803C>A | Tier2 | 1 / 310 | |
| chr4:187531111 | TGTTAG | T | 15 / 27 | p.L3303fs | c.9907_9911delCTAAC | Tier2 | 1 / 310 | |
| chr4:187530408 | G | A | 16 / 27 | p.Q3379* | c.10135C>T | COSV104716925 | Tier2 | 1 / 310 |
| chr4:187530421 | AATTG | A | 16 / 27 | p.S3373fs | c.10118_10121delCAAT | Tier2 | 1 / 310 | |
| chr4:187527274 | TGACATCG | T | 17 / 27 | p.D3432fs | c.10293_10299delCGATGTC | Tier2 | 1 / 310 | |
| chr4:187525541 | AGT | A | 18 / 27 | p.L3512fs | c.10536_10537delAC | Tier2 | 1 / 310 | |
| chr4:187524330 | C | A | 19 / 27 | p.E3784* | c.11350G>T | Tier2 | 1 / 310 | |
| chr4:187524567 | G | A | 19 / 27 | p.Q3705* | c.11113C>T | COSV71673051 | Tier2 | 1 / 310 |
| chr4:187524720 | C | A | 19 / 27 | p.E3654* | c.10960G>T | COSV71674882 | Tier2 | 1 / 310 |
| chr4:187524787 | G | GA | 19 / 27 | p.R3632fs | c.10892dupT | Tier2 | 1 / 310 | |
| chr4:187524906 | CAG | C | 19 / 27 | p.S3591fs | c.10772_10773delCT | Tier2 | 1 / 310 | |
| chr4:187524917 | TTGTC | T | 19 / 27 | p.D3587fs | c.10759_10762delGACA | Tier2 | 1 / 310 | |
| chr4:187522425 | C | A | 21 / 27 | p.E3880* | c.11638G>T | Tier2 | 1 / 310 | |
| chr4:187522495 | GGTCA | G | 21 / 27 | p.L3855fs | c.11564_11567delTGAC | Tier2 | 1 / 310 | |
| chr4:187522557 | C | A | 21 / 27 | p.G3836* | c.11506G>T | Tier2 | 1 / 310 | |
| chr4:187521157 | C | A | 22 / 27 | p.E4000* | c.11998G>T | Tier2 | 1 / 310 | |
| chr4:187521224 | CA | C | 22 / 27 | p.M3977fs | c.11930delT | Tier2 | 1 / 310 | |
| chr4:187521370 | G | A | 22 / 27 | p.Q3929* | c.11785C>T | COSV71672375 | Tier2 | 1 / 310 |
| chr4:187521442 | G | A | 22 / 27 | p.Q3905* | c.11713C>T | COSV71676602 | Tier2 | 2 / 310 |
| chr4:187521451 | G | A | 22 / 27 | p.Q3902* | c.11704C>T | COSV71673132 | Tier2 | 1 / 310 |
| chr4:187521478 | C | A | 22 / 27 | p.G3893* | c.11677G>T | Tier2 | 1 / 310 | |
| chr4:187519258 | G | GC | 23 / 27 | p.A4042fs | c.12124dupG | Tier2 | 1 / 310 | |
| chr4:187519275 | A | T | 23 / 27 | p.Y4036* | c.12108T>A | Tier2 | 1 / 310 | |
| chr4:187518840 | C | A | 24 / 27 | p.E4122* | c.12364G>T | Tier2 | 1 / 310 | |
| chr4:187518030 | C | A | 25 / 27 | p.G4222* | c.12664G>T | Tier2 | 1 / 310 | |
| chr4:187518311 | ATATCACTCTG | A | 25 / 27 | p.Q4125fs | c.12373_12382delCAGAGTGATA | Tier2 | 1 / 310 | |
| chr4:187510318 | C | A | 27 / 27 | p.E4399* | c.13195G>T | Tier2 | 1 / 310 |