Side Menu
KMT2D
基本情報
- 遺伝子名
-
lysine methyltransferase 2D
- 慣用名
-
AAD10, ALR, CAGL114, KABUK1, KMS, MLL2, MLL4, TNRC21
- 遺伝子分類
-
がん遺伝子 / がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
エピジェネティック制御
- シグナル伝達経路
-
Epigenetic modification
- 染色体上の位置 (GRCh37/hg19)
-
12q13.12 (chr12:49415563..49449107, complement)
- アミノ酸配列の長さ
-
5,537
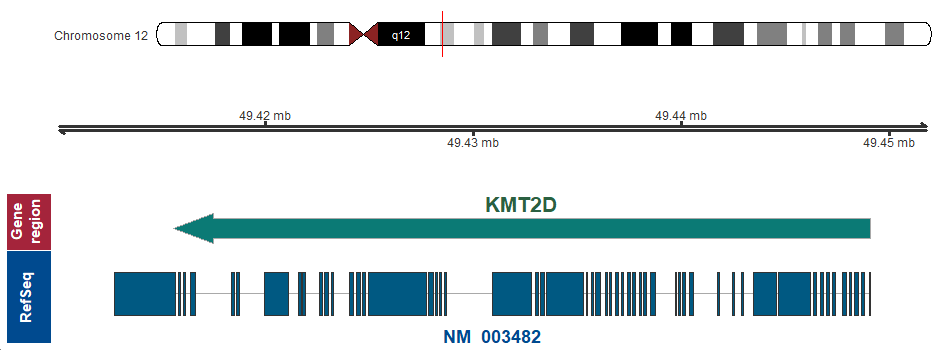
遺伝子マップ
解説
KMT2D(MLL2)はヒストンメチル基転移酵素であり、ヒストンH3の4番目のリジン残基(H3K4)のメチル化を行うことで、発生や細胞分化における転写の活性化に関与する。また、KMT2Dの欠失はゲノム不安定性を引き起こすことが報告されている。体細胞変異は、濾胞性リンパ腫、びまん性大細胞型B細胞リンパ腫において認められる。
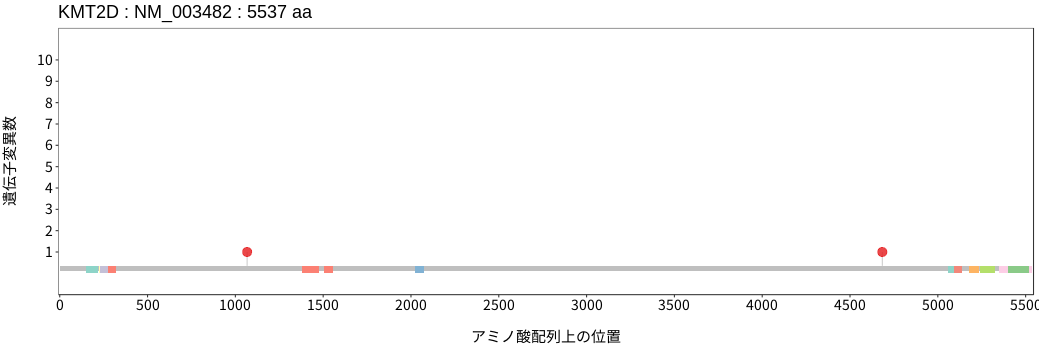
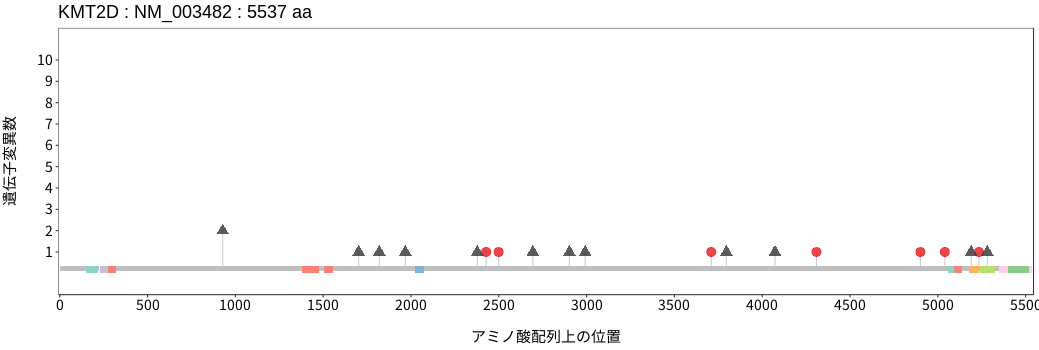
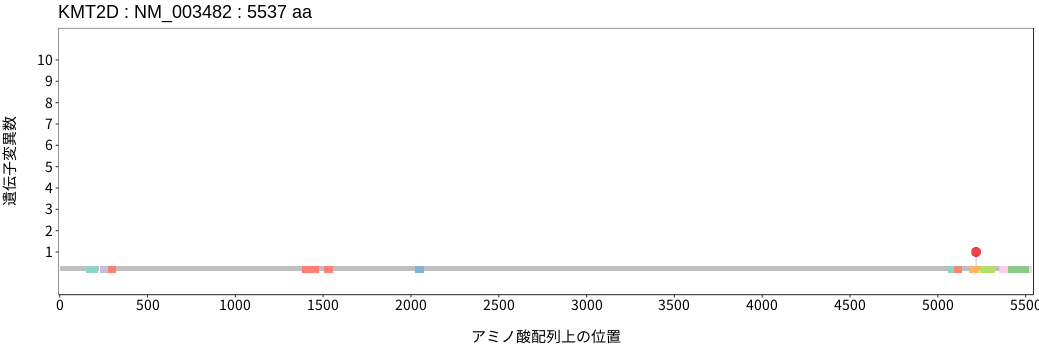
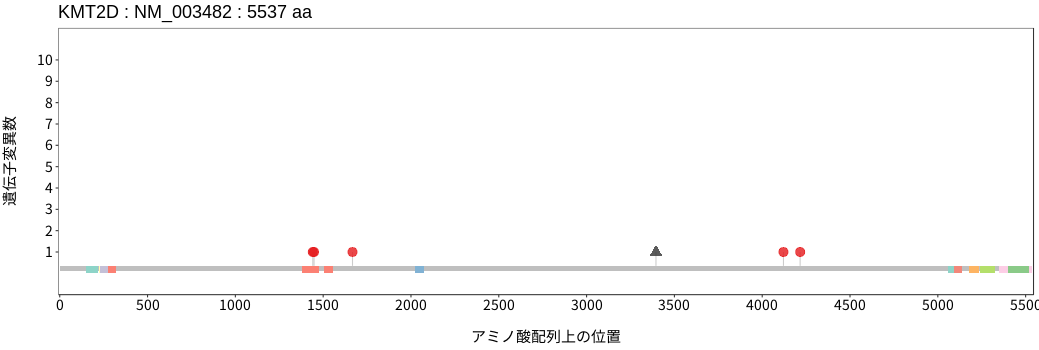
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

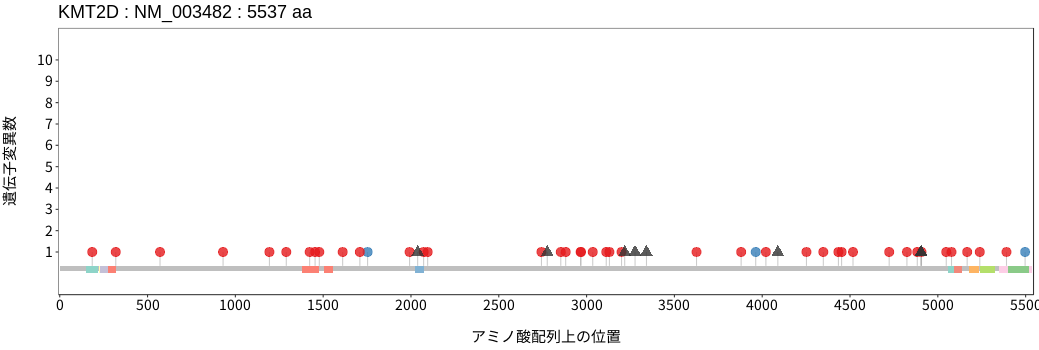
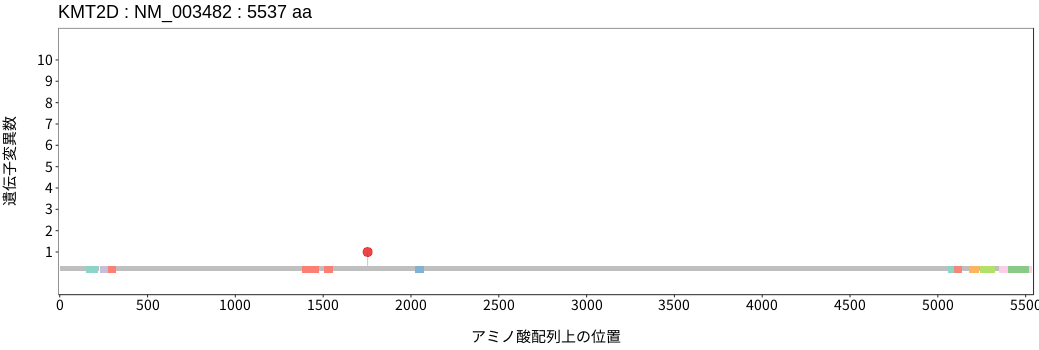
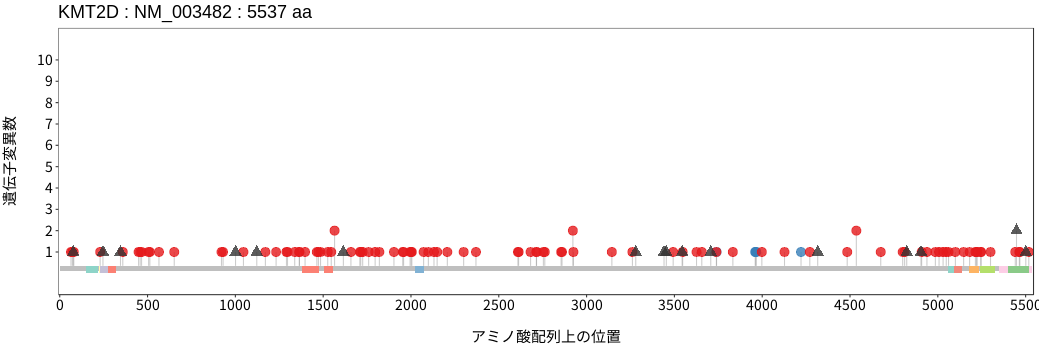
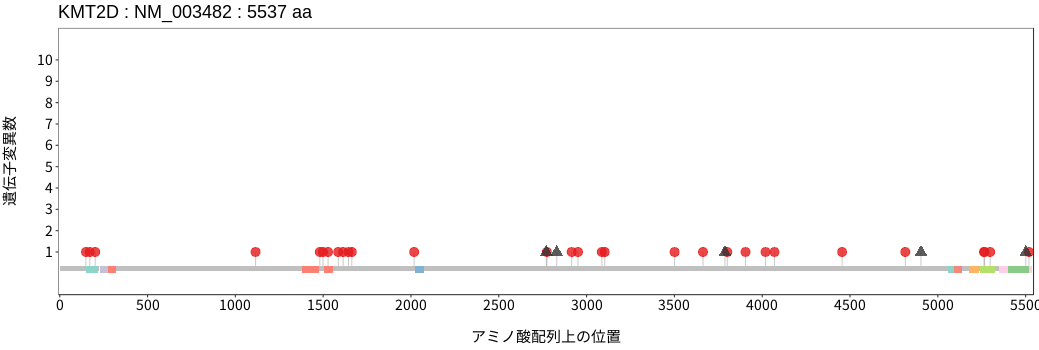
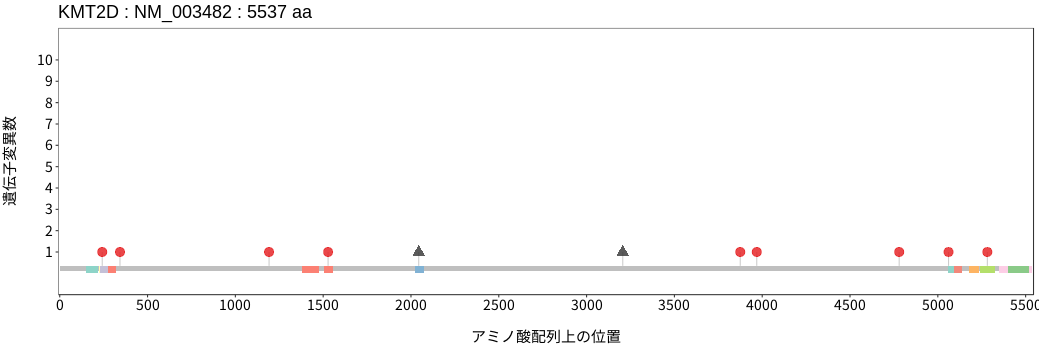
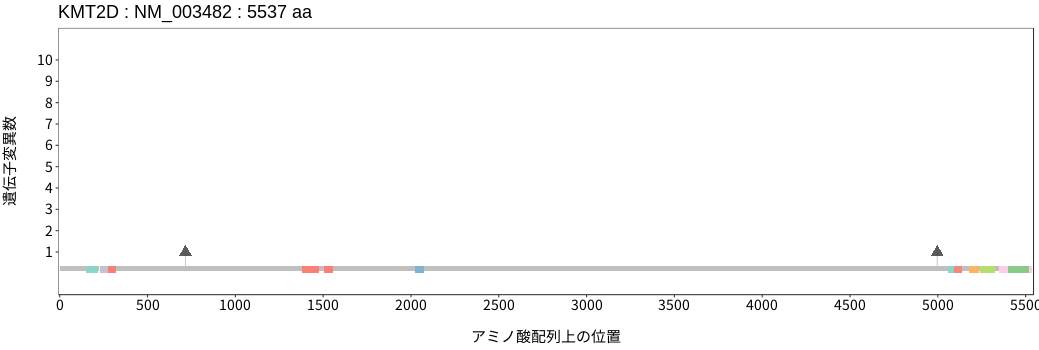
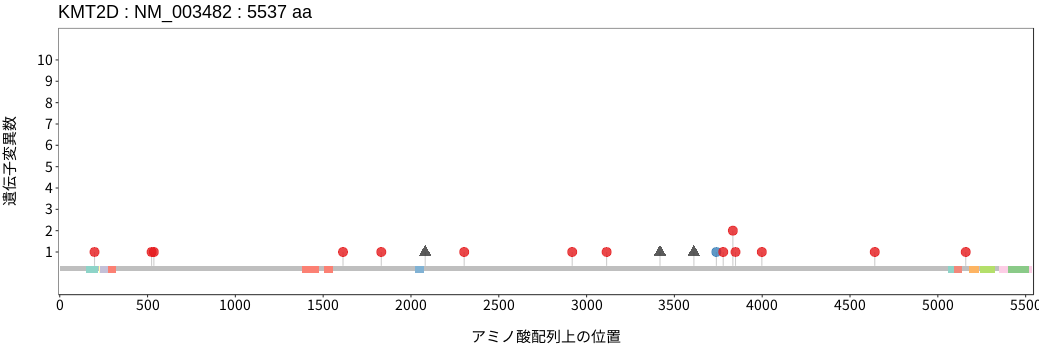
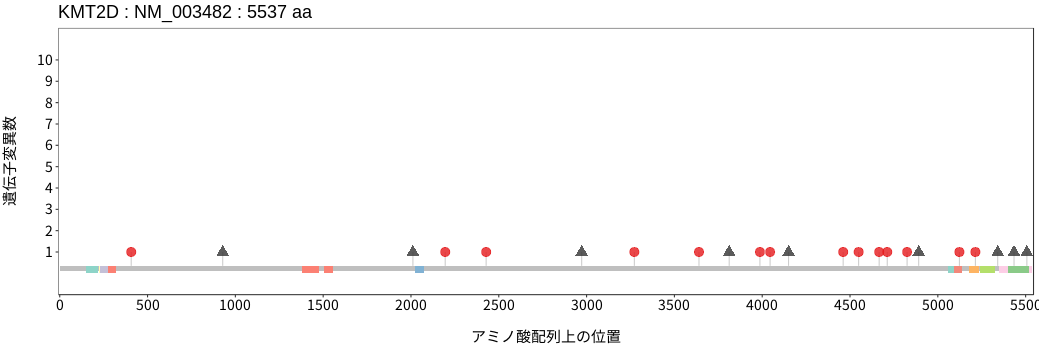
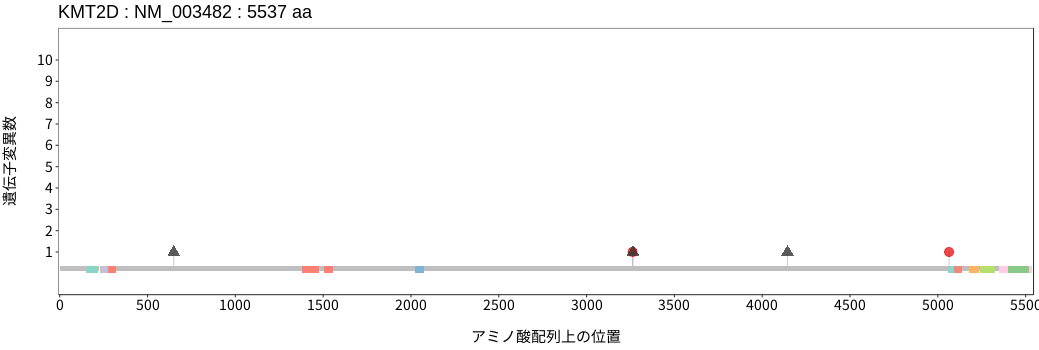
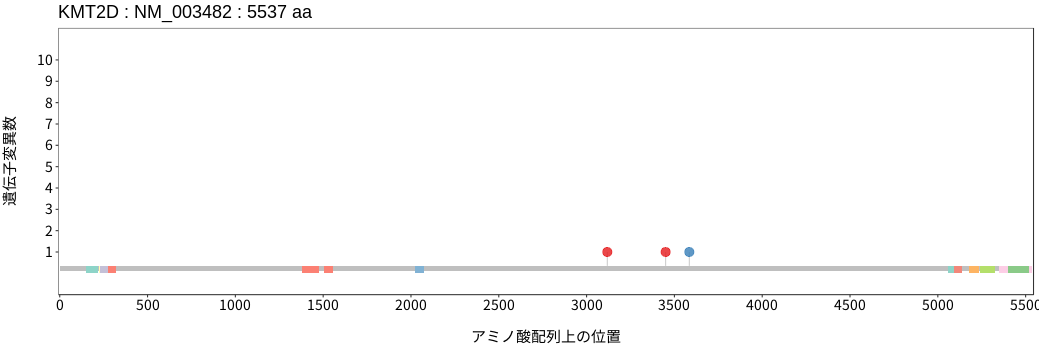
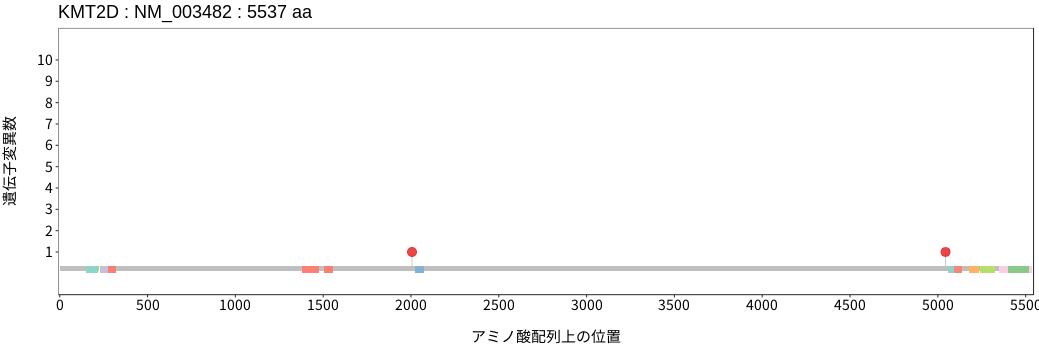
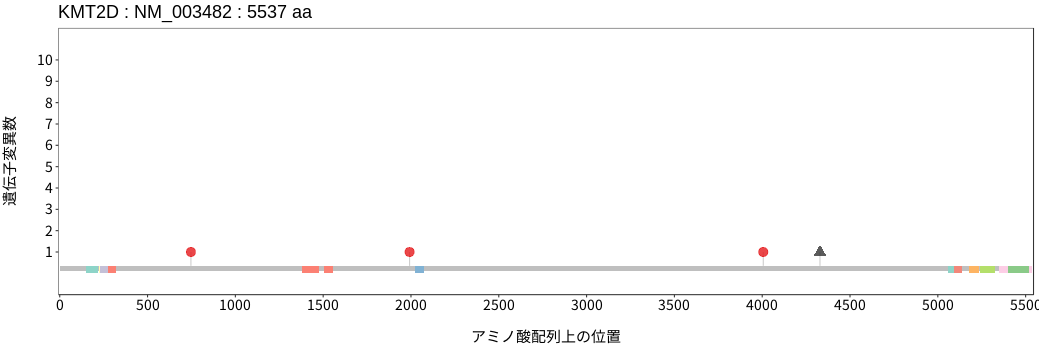
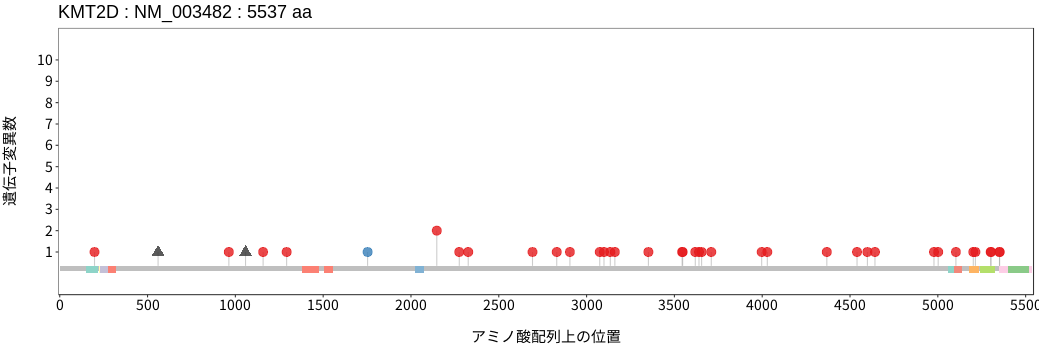
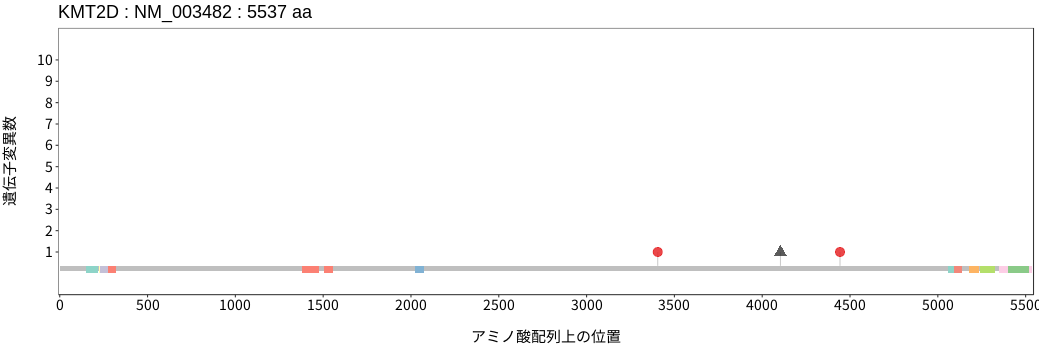
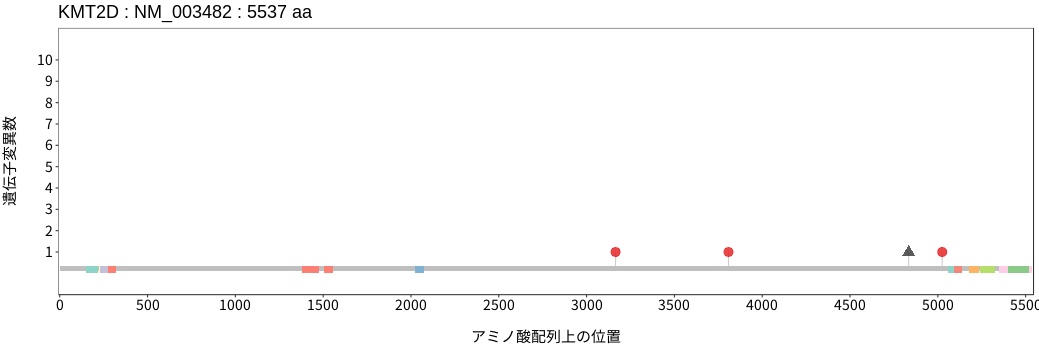
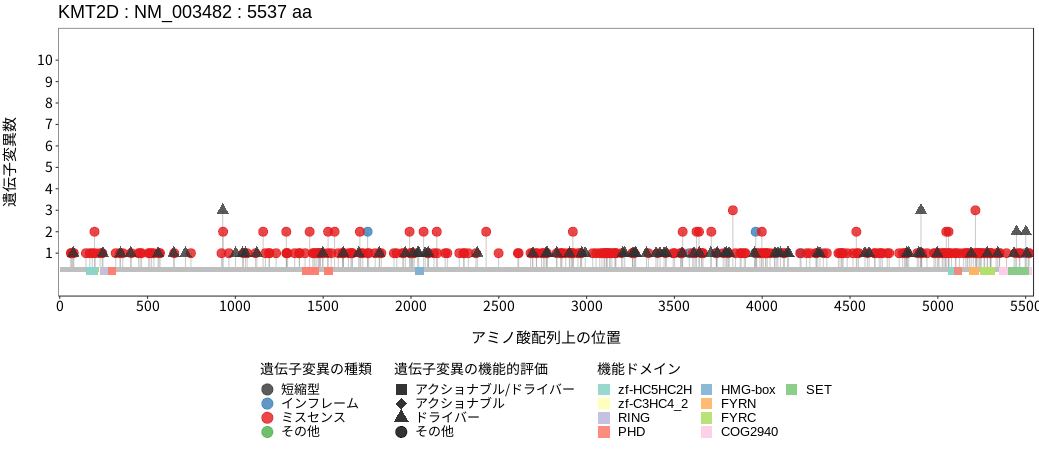
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr12:49448479 | CG | C | 3 / 54 | p.H77fs | c.231delC | COSV56471398 | Tier2 | 1 / 372 |
| chr12:49447361 | CT | C | 6 / 54 | p.S246fs | c.736delA | Tier2 | 1 / 372 | |
| chr12:49446773 | CAG | C | 8 / 54 | p.C346fs | c.1035_1036delCT | COSV56420417 | Tier2 | 1 / 372 |
| chr12:49446393 | C | CA | 9 / 54 | p.M404fs | c.1211dupT | Tier2 | 1 / 372 | |
| chr12:49444684 | G | A | 10 / 54 | p.Q928* | c.2782C>T | COSV56458548 | Tier2 | 3 / 372 |
| chr12:49445317 | AC | A | 10 / 54 | p.M716fs | c.2148delG | Tier2 | 1 / 372 | |
| chr12:49445521 | C | A | 10 / 54 | p.E649* | c.1945G>T | COSV99985669 | Tier2 | 1 / 372 |
| chr12:49445788 | C | A | 10 / 54 | p.E560* | c.1678G>T | Tier2 | 1 / 372 | |
| chr12:49444004 | CTG | C | 11 / 54 | p.T1122fs | c.3365_3366delCA | Tier2 | 1 / 372 | |
| chr12:49444196 | CTA | C | 11 / 54 | p.I1058fs | c.3173_3174delTA | Tier2 | 1 / 372 | |
| chr12:49444250 | G | A | 11 / 54 | p.Q1041* | c.3121C>T | Tier2 | 1 / 372 | |
| chr12:49444369 | AG | A | 11 / 54 | p.L1001fs | c.3001delC | Tier2 | 1 / 372 | |
| chr12:49440134 | AGT | A | 16 / 54 | p.H1497fs | c.4490_4491delAC | COSV99983599 | Tier2 | 1 / 372 |
| chr12:49438647 | G | A | 19 / 54 | p.R1615* | c.4843C>T | COSV56420226 | Tier2 | 1 / 372 |
| chr12:49438067 | G | A | 21 / 54 | p.R1702* | c.5104C>T | COSV56407907 | Tier2 | 1 / 372 |
| chr12:49437426 | G | T | 23 / 54 | p.S1820* | c.5459C>A | Tier2 | 1 / 372 | |
| chr12:49435950 | C | A | 28 / 54 | p.E2011* | c.6031G>T | COSV56494116 | Tier2 | 1 / 372 |
| chr12:49436079 | C | A | 28 / 54 | p.E1968* | c.5902G>T | COSV56493501 | Tier2 | 1 / 372 |
| chr12:49435753 | G | A | 29 / 54 | p.Q2044* | c.6130C>T | COSV56433680 | Tier2 | 1 / 372 |
| chr12:49435769 | C | T | 29 / 54 | p.W2038* | c.6114G>A | Tier2 | 1 / 372 | |
| chr12:49434421 | A | AT | 31 / 54 | p.Y2378fs | c.7131dupA | Tier2 | 1 / 372 | |
| chr12:49435258 | G | A | 31 / 54 | p.R2099* | c.6295C>T | COSV56487468 | Tier2 | 1 / 372 |
| chr12:49435312 | C | A | 31 / 54 | p.E2081* | c.6241G>T | Tier2 | 1 / 372 | |
| chr12:49433367 | G | A | 32 / 54 | p.Q2694* | c.8080C>T | Tier2 | 1 / 372 | |
| chr12:49433040 | TGGAGG | T | 33 / 54 | p.P2776fs | c.8326_8330delCCTCC | Tier2 | 1 / 372 | |
| chr12:49433060 | G | A | 33 / 54 | p.R2771* | c.8311C>T | COSV56428815 | Tier2 | 1 / 372 |
| chr12:49430956 | G | A | 34 / 54 | p.Q3395* | c.10183C>T | Tier2 | 1 / 372 | |
| chr12:49431118 | G | A | 34 / 54 | p.Q3341* | c.10021C>T | COSV56482593 | Tier2 | 1 / 372 |
| chr12:49431301 | G | A | 34 / 54 | p.Q3280* | c.9838C>T | Tier2 | 1 / 372 | |
| chr12:49431313 | G | A | 34 / 54 | p.Q3276* | c.9826C>T | Tier2 | 1 / 372 | |
| chr12:49431349 | G | A | 34 / 54 | p.Q3264* | c.9790C>T | Tier2 | 1 / 372 | |
| chr12:49431488 | ACT | A | 34 / 54 | p.S3217fs | c.9649_9650delAG | Tier2 | 1 / 372 | |
| chr12:49431523 | C | A | 34 / 54 | p.G3206* | c.9616G>T | Tier2 | 1 / 372 | |
| chr12:49432165 | C | A | 34 / 54 | p.E2992* | c.8974G>T | Tier2 | 1 / 372 | |
| chr12:49432225 | C | A | 34 / 54 | p.E2972* | c.8914G>T | Tier2 | 1 / 372 | |
| chr12:49432435 | G | A | 34 / 54 | p.Q2902* | c.8704C>T | COSV56498164 | Tier2 | 1 / 372 |
| chr12:49432651 | G | A | 34 / 54 | p.R2830* | c.8488C>T | COSV56427366 | Tier2 | 1 / 372 |
| chr12:49428629 | G | A | 35 / 54 | p.Q3441* | c.10321C>T | COSV56427390 | Tier2 | 1 / 372 |
| chr12:49428698 | C | A | 35 / 54 | p.E3418* | c.10252G>T | Tier2 | 1 / 372 | |
| chr12:49428445 | G | A | 36 / 54 | p.Q3454* | c.10360C>T | COSV56427412 | Tier2 | 1 / 372 |
| chr12:49427959 | GCA | G | 38 / 54 | p.A3544fs | c.10629_10630delTG | Tier2 | 1 / 372 | |
| chr12:49425503 | G | A | 39 / 54 | p.Q4329* | c.12985C>T | COSV56445048 | Tier2 | 1 / 372 |
| chr12:49425542 | C | A | 39 / 54 | p.E4316* | c.12946G>T | Tier2 | 1 / 372 | |
| chr12:49426040 | G | A | 39 / 54 | p.Q4150* | c.12448C>T | Tier2 | 1 / 372 | |
| chr12:49426058 | G | A | 39 / 54 | p.Q4144* | c.12430C>T | Tier2 | 1 / 372 | |
| chr12:49426178 | G | A | 39 / 54 | p.Q4104* | c.12310C>T | Tier2 | 1 / 372 | |
| chr12:49426221 | CAG | C | 39 / 54 | p.L4089fs | c.12265_12266delCT | COSV56461303 | Tier2 | 1 / 372 |
| chr12:49426270 | G | T | 39 / 54 | p.S4073* | c.12218C>A | Tier2 | 1 / 372 | |
| chr12:49426622 | G | A | 39 / 54 | p.Q3956* | c.11866C>T | Tier2 | 1 / 372 | |
| chr12:49427054 | G | A | 39 / 54 | p.Q3812* | c.11434C>T | COSV104616224 | Tier2 | 1 / 372 |
| chr12:49427102 | G | A | 39 / 54 | p.Q3796* | c.11386C>T | COSV56483498 | Tier2 | 1 / 372 |
| chr12:49427126 | G | A | 39 / 54 | p.Q3788* | c.11362C>T | COSV56438266 | Tier2 | 1 / 372 |
| chr12:49427258 | G | A | 39 / 54 | p.Q3744* | c.11230C>T | COSV99985372 | Tier2 | 1 / 372 |
| chr12:49427369 | G | A | 39 / 54 | p.R3707* | c.11119C>T | COSV56422615 | Tier2 | 1 / 372 |
| chr12:49427564 | G | A | 39 / 54 | p.Q3642* | c.10924C>T | Tier2 | 1 / 372 | |
| chr12:49427657 | G | A | 39 / 54 | p.Q3611* | c.10831C>T | COSV56407766 | Tier2 | 1 / 372 |
| chr12:49424398 | G | A | 41 / 54 | p.Q4609* | c.13825C>T | Tier2 | 1 / 372 | |
| chr12:49424466 | TTGAC | T | 41 / 54 | p.V4585fs | c.13753_13756delGTCA | Tier2 | 1 / 372 | |
| chr12:49421807 | C | A | 46 / 54 | p.E4834* | c.14500G>T | Tier2 | 1 / 372 | |
| chr12:49421817 | TGGCTCAGTGCCTGCCCGGGCGG | T | 46 / 54 | p.P4823fs | c.14468_14489delCCGCCCGGGCAGGCACTGAGCC | Tier2 | 1 / 372 | |
| chr12:49420178 | GCTGT | G | 48 / 54 | p.Q5190fs | c.15567_15570delACAG | Tier2 | 1 / 372 | |
| chr12:49420760 | G | A | 48 / 54 | p.Q4997* | c.14989C>T | Tier2 | 1 / 372 | |
| chr12:49421036 | G | A | 48 / 54 | p.Q4905* | c.14713C>T | Tier2 | 1 / 372 | |
| chr12:49421039 | G | A | 48 / 54 | p.R4904* | c.14710C>T | COSV56414273 | Tier2 | 3 / 372 |
| chr12:49421078 | G | A | 48 / 54 | p.Q4891* | c.14671C>T | Tier2 | 1 / 372 | |
| chr12:49418670 | G | A | 49 / 54 | p.R5282* | c.15844C>T | COSV56407274 | Tier2 | 1 / 372 |
| chr12:49418391 | G | C | 50 / 54 | p.S5341* | c.16022C>G | Tier2 | 1 / 372 | |
| chr12:49416411 | C | A | 51 / 54 | p.E5434* | c.16300G>T | Tier2 | 1 / 372 | |
| chr12:49416133 | G | A | 52 / 54 | p.R5448* | c.16342C>T | COSV56418474 | Tier2 | 2 / 372 |
| chr12:49415828 | C | A | 53 / 54 | p.E5507* | c.16519G>T | COSV56445827 | Tier2 | 1 / 372 |
| chr12:49415846 | G | A | 53 / 54 | p.R5501* | c.16501C>T | COSV56407191 | Tier2 | 2 / 372 |