Side Menu
KRAS
基本情報
- 遺伝子名
-
KRAS proto-oncogene, GTPase
- 慣用名
-
'C-K-RAS, C-K-RAS, CFC2, K-RAS2A, K-RAS2B, K-RAS4A, K-RAS4B, K-Ras, K-Ras 2, KI-RAS, KRAS1, KRAS2, NS, NS3, OES, RALD, RASK2, c-Ki-ras, c-Ki-ras2
- 遺伝子分類
-
がん遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
腫瘍形成・増殖
- シグナル伝達経路
-
MAPK
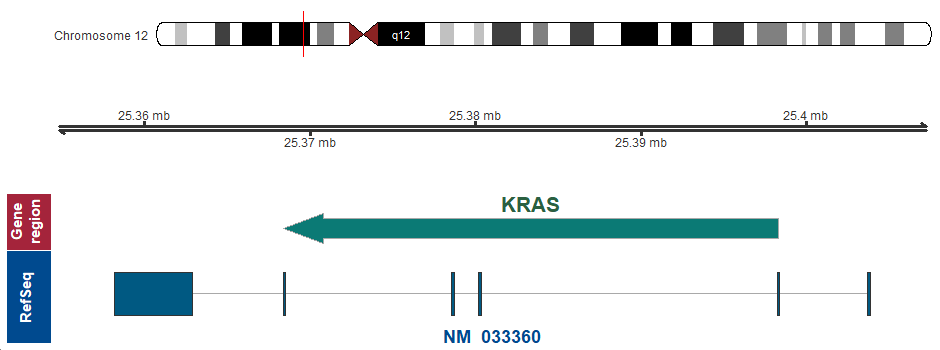
- 染色体上の位置 (GRCh37/hg19)
-
12p12.1 (chr12:25368375..25398318, complement)
- アミノ酸配列の長さ
-
189
遺伝子マップ
解説
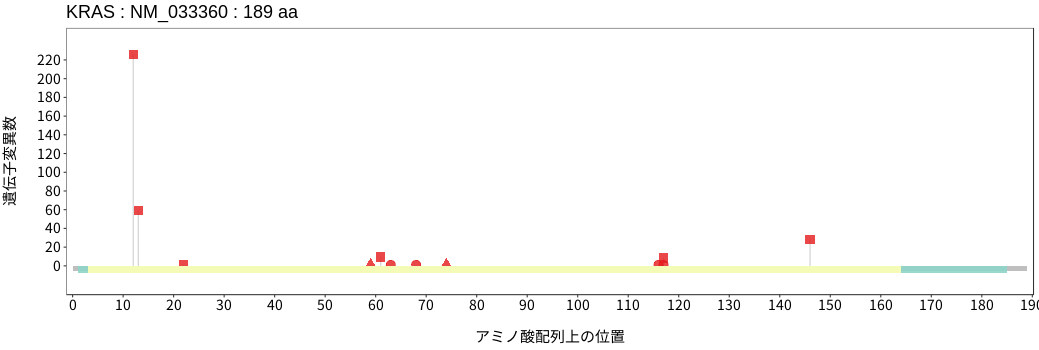
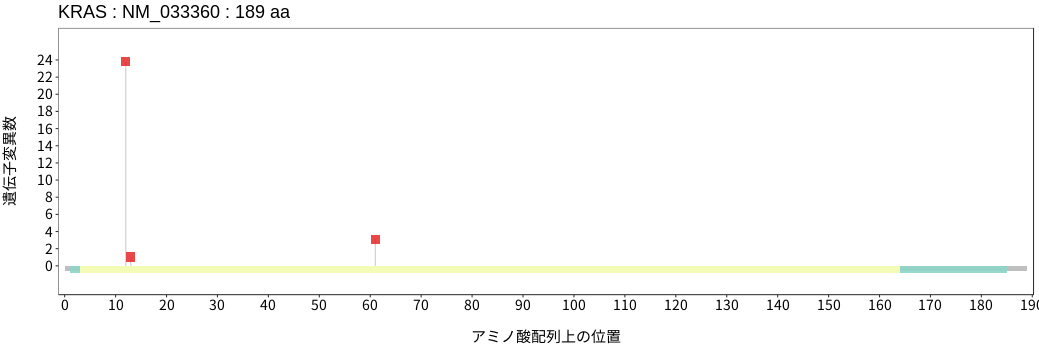
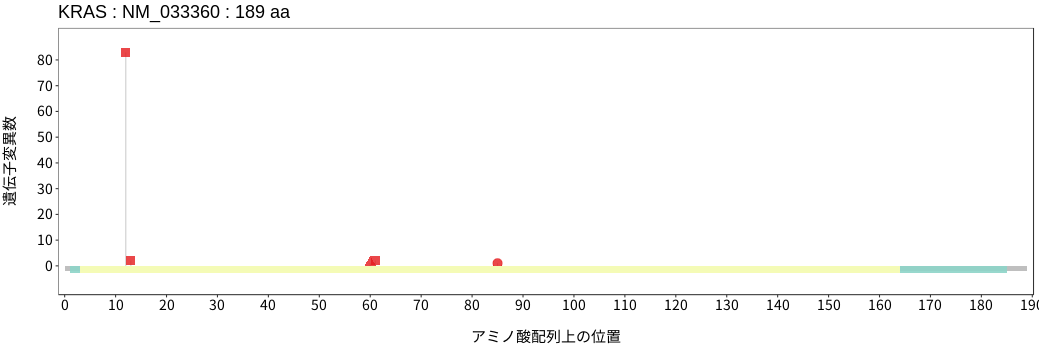
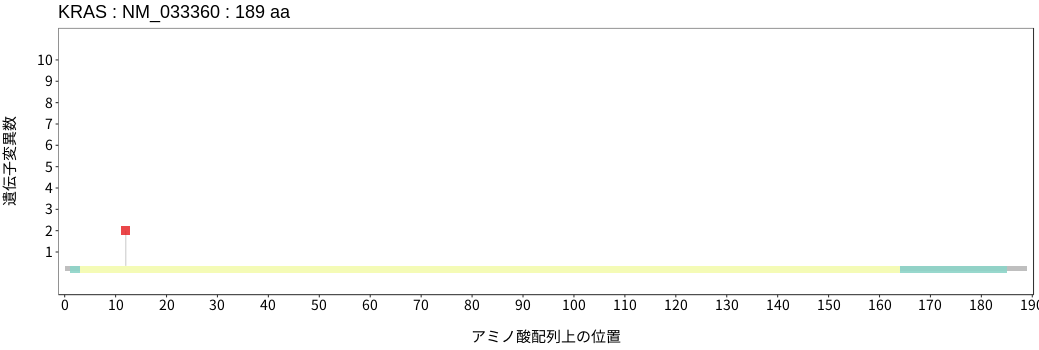
KRASはHRASおよびNRASと同じく、低分子GTP結合タンパク質のRASファミリーに属する。RASは上流の受容体チロシンキナーゼにおける活性化シグナルを受け、guanine nucleotide exchange factor (GEF)であるSOS1により活性型のGTP結合型RASに変換されることで、細胞増殖に関連する下流のMAPKおよびPI3K/Akt/mTORシグナル経路の活性化を引き起こす。一方で、GTPase-activating protein(GAP)であるNF1およびRASA1によりRASの内在性GTPase活性が増強され、非活性型のGDP結合型RASに変換されることでその活性は抑制される。体細胞バリアントは膵臓癌、大腸癌、子宮内膜癌、卵巣癌、そして胆管癌において高い頻度で検出され、そのホットスポットは12番目および13番目のアミノ酸グリシン、そして61番目のアミノ酸グルタミンである。これらホットスポットに位置するバリアントによりKRASのGTPase活性が阻害され、活性型のGTP結合型RASの蓄積が引き起こされるため、下流の細胞増殖関連シグナル経路の恒常的な活性化が誘導される。
シグナル伝達経路

遺伝子変異総量(Tumor Mutation Burden)の分布TMB

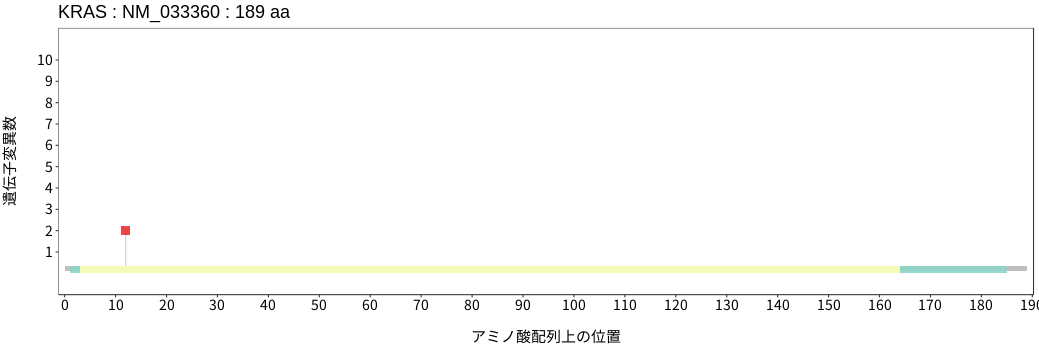
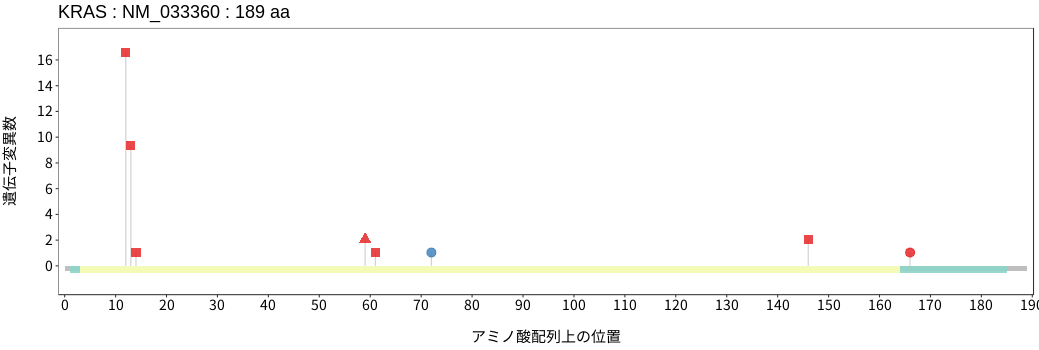
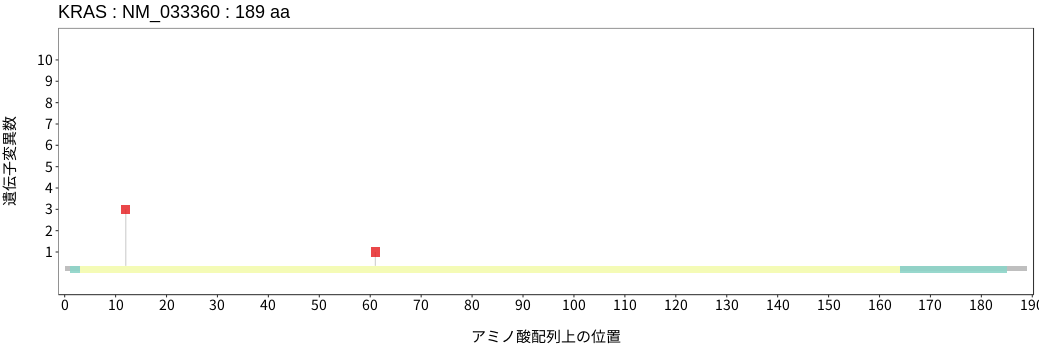
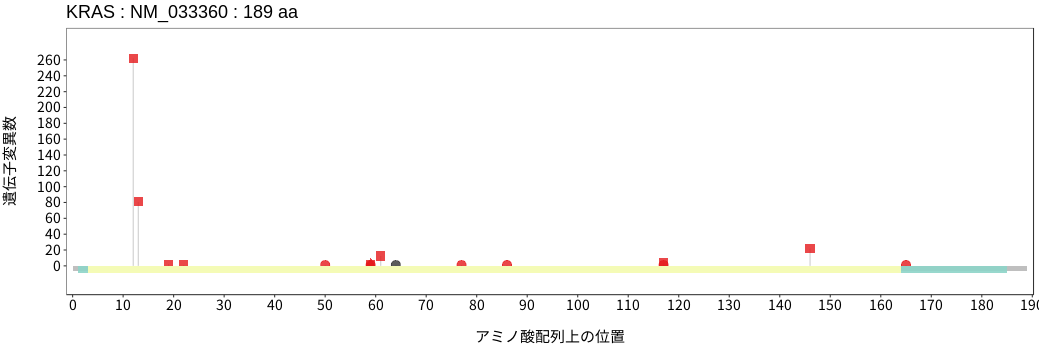
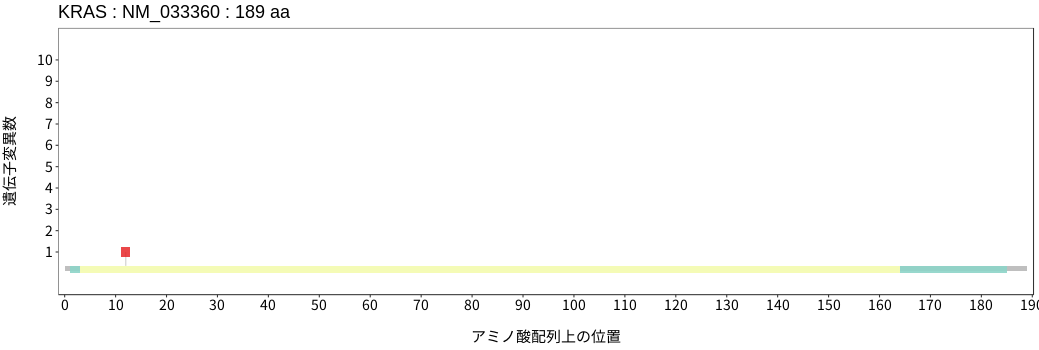
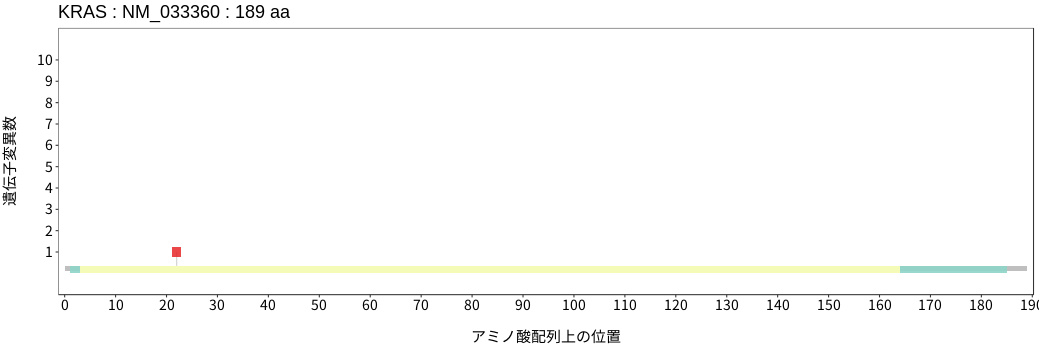
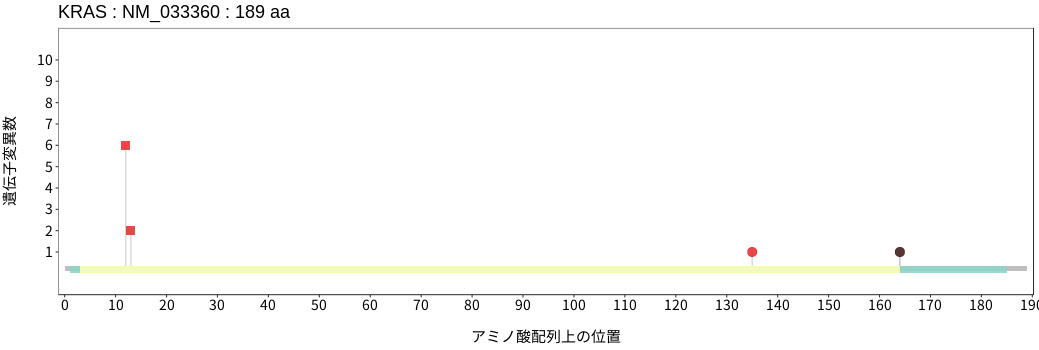
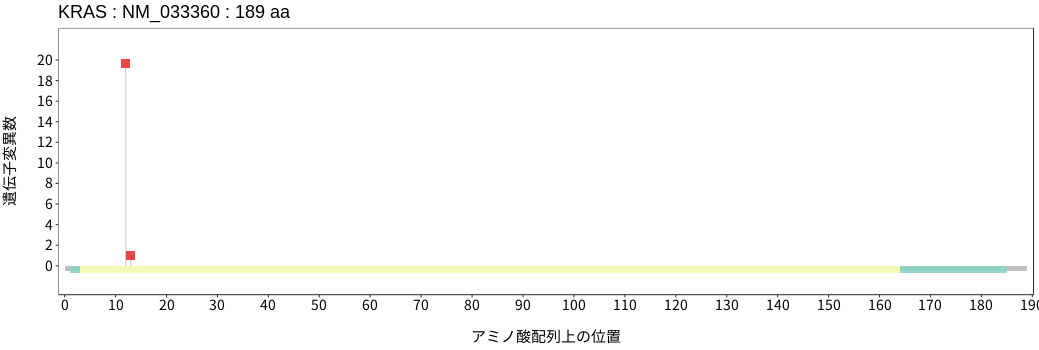
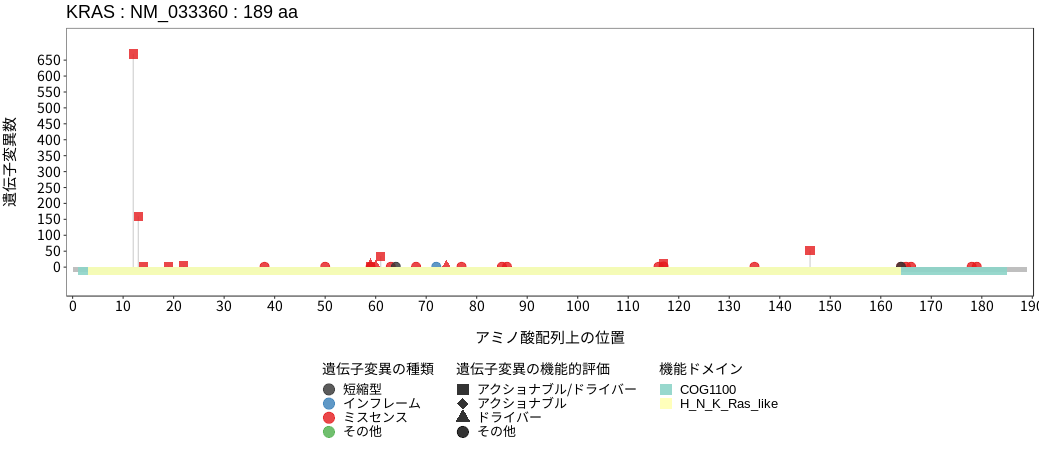
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr12:25398255 | G | T | 2 / 6 | p.Q22K | c.64C>A | COSV55526911 | Tier1 | 3 / 928 |
| chr12:25398262 | C | A | 2 / 6 | p.L19F | c.57G>T | COSV55502103 | Tier1 | 2 / 928 |
| chr12:25398279 | C | T | 2 / 6 | p.V14I | c.40G>A | COSV55501342 | Tier2 | 1 / 928 |
| chr12:25398281 | C | T | 2 / 6 | p.G13D | c.38G>A | COSV55497388 | Tier1 | 143 / 928 |
| chr12:25398281 | C | A | 2 / 6 | p.G13V | c.38G>T | COSV55522580 | Tier1 | 1 / 928 |
| chr12:25398282 | C | A | 2 / 6 | p.G13C | c.37G>T | COSV55497378 | Tier1 | 9 / 928 |
| chr12:25398282 | C | G | 2 / 6 | p.G13R | c.37G>C | COSV55502117 | Tier1 | 1 / 928 |
| chr12:25398284 | C | A | 2 / 6 | p.G12V | c.35G>T | COSV55497419 | Tier1 | 176 / 928 |
| chr12:25398284 | C | T | 2 / 6 | p.G12D | c.35G>A | COSV55497369 | Tier1 | 291 / 928 |
| chr12:25398284 | C | G | 2 / 6 | p.G12A | c.35G>C | COSV55497479 | Tier1 | 47 / 928 |
| chr12:25398285 | C | A | 2 / 6 | p.G12C | c.34G>T | COSV55497469 | Tier1 | 73 / 928 |
| chr12:25398285 | C | G | 2 / 6 | p.G12R | c.34G>C | COSV55497582 | Tier1 | 14 / 928 |
| chr12:25398285 | C | T | 2 / 6 | p.G12S | c.34G>A | COSV55497461 | Tier1 | 45 / 928 |
| chr12:25380238 | T | G | 3 / 6 | p.T74P | c.220A>C | COSV55722485 | Tier2 | 1 / 928 |
| chr12:25380275 | T | A | 3 / 6 | p.Q61H | c.183A>T | COSV55499223 | Tier1 | 6 / 928 |
| chr12:25380275 | T | G | 3 / 6 | p.Q61H | c.183A>C | COSV55498802 | Tier1 | 19 / 928 |
| chr12:25380276 | T | C | 3 / 6 | p.Q61R | c.182A>G | COSV55498739 | Tier1 | 4 / 928 |
| chr12:25380276 | T | A | 3 / 6 | p.Q61L | c.182A>T | COSV55504296 | Tier1 | 4 / 928 |
| chr12:25380279 | C | A | 3 / 6 | p.G60V | c.179G>T | COSV55708344 | Tier2 | 1 / 928 |
| chr12:25380282 | G | C | 3 / 6 | p.A59G | c.176C>G | COSV55604554 | Tier1 | 1 / 928 |
| chr12:25380283 | C | T | 3 / 6 | p.A59T | c.175G>A | COSV55499283 | Tier1 | 5 / 928 |
| chr12:25378561 | G | A | 4 / 6 | p.A146V | c.437C>T | COSV55498939 | Tier1 | 6 / 928 |
| chr12:25378562 | C | T | 4 / 6 | p.A146T | c.436G>A | COSV55501778 | Tier1 | 41 / 928 |
| chr12:25378562 | C | G | 4 / 6 | p.A146P | c.436G>C | COSV55541748 | Tier1 | 3 / 928 |
| chr12:25378647 | T | A | 4 / 6 | p.K117N | c.351A>T | COSV55504752 | Tier1 | 10 / 928 |
| chr12:25378647 | T | G | 4 / 6 | p.K117N | c.351A>C | COSV55545304 | Tier1 | 2 / 928 |