Side Menu
MAP3K1
基本情報
- 遺伝子名
-
mitogen-activated protein kinase kinase kinase 1
- 慣用名
-
MAPKKK1, MEKK, MEKK 1, MEKK1, SRXY6
- 遺伝子分類
-
がん遺伝子 / がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
腫瘍形成・増殖
- シグナル伝達経路
-
MAPK
- 染色体上の位置 (GRCh37/hg19)
-
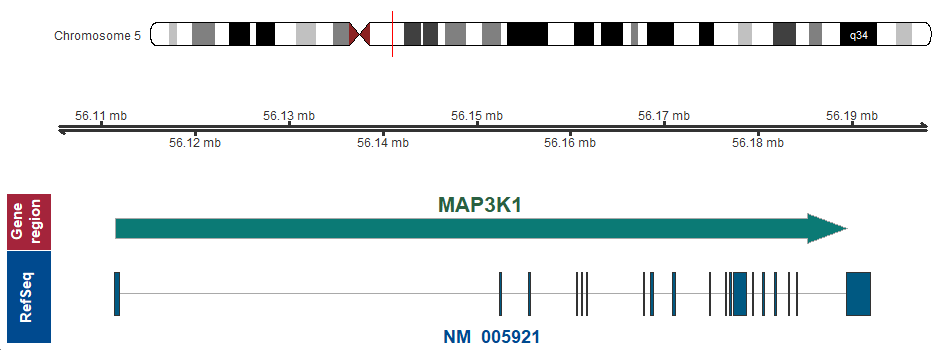
5q11.2 (chr5:56111401..56189507)
- アミノ酸配列の長さ
-
1,512
遺伝子マップ
解説
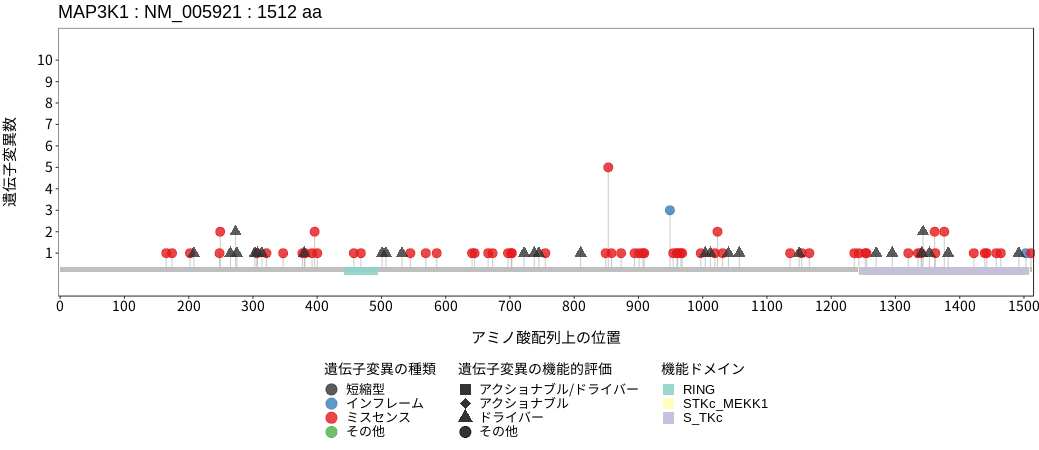
MAP3K1 (MEKK1)は紫外線や電離放射線などの環境ストレスに応答し、下流のMAP2K4 (MKK4)をリン酸化することでJNK経路を活性化させる。また、MAP3K1はスモイル化およびユビキチン化によりMEKおよびERKの活性調節に働くことが知られている。MAP3K1の短縮型変異もしくは欠失は、乳癌のルミナルAタイプにおいて高い頻度で認められる。
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

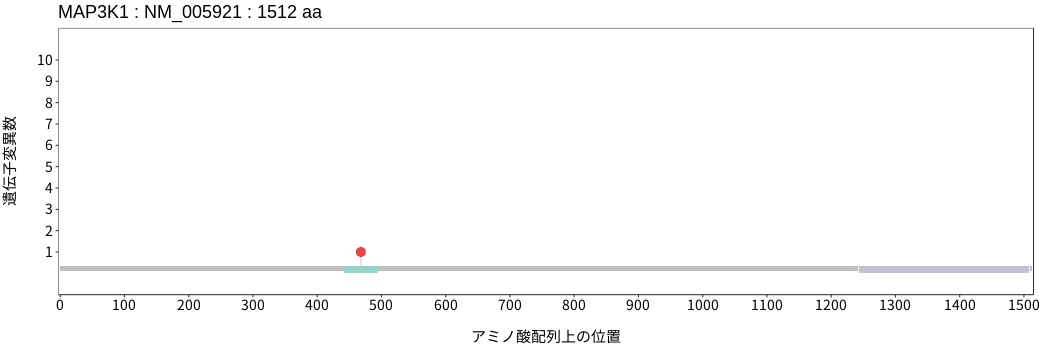
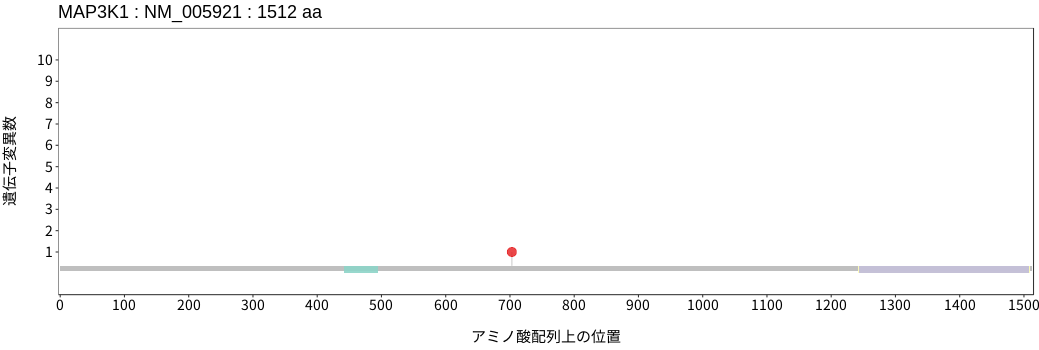
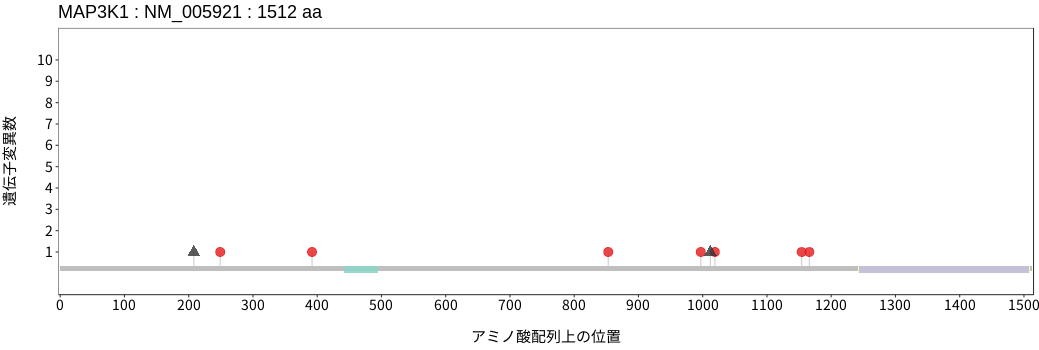
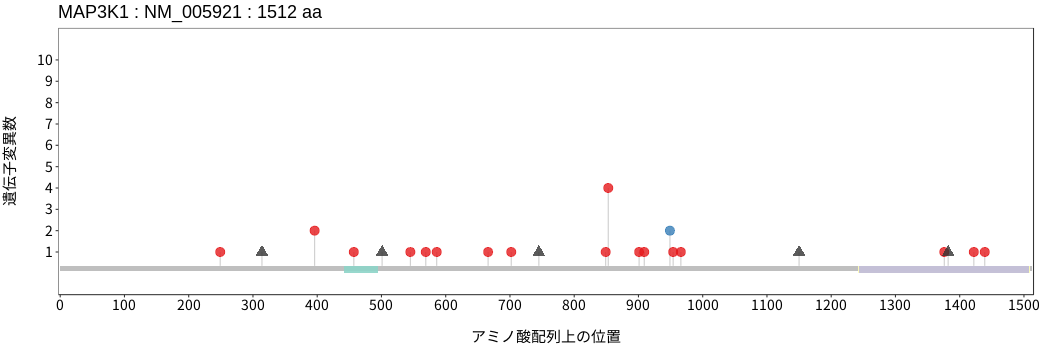
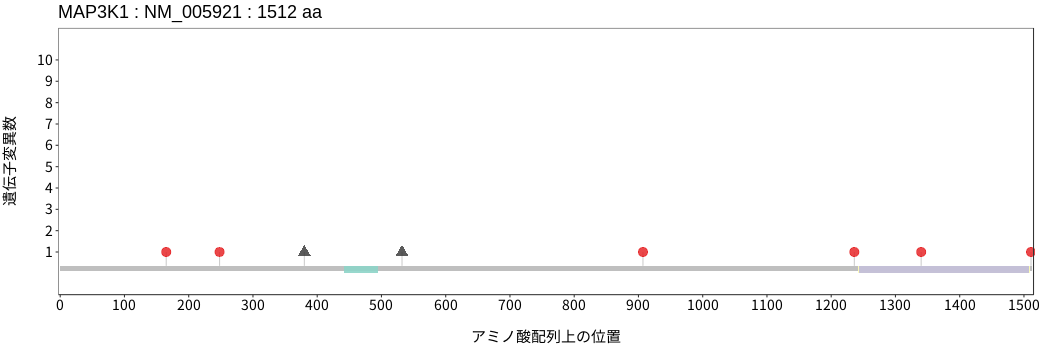
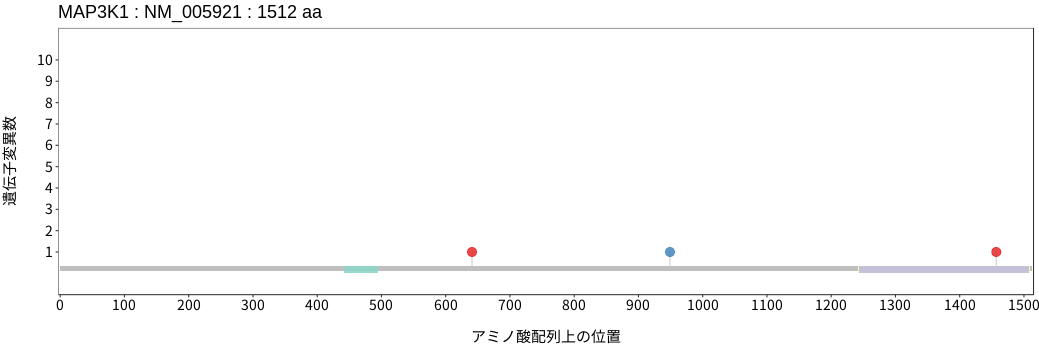
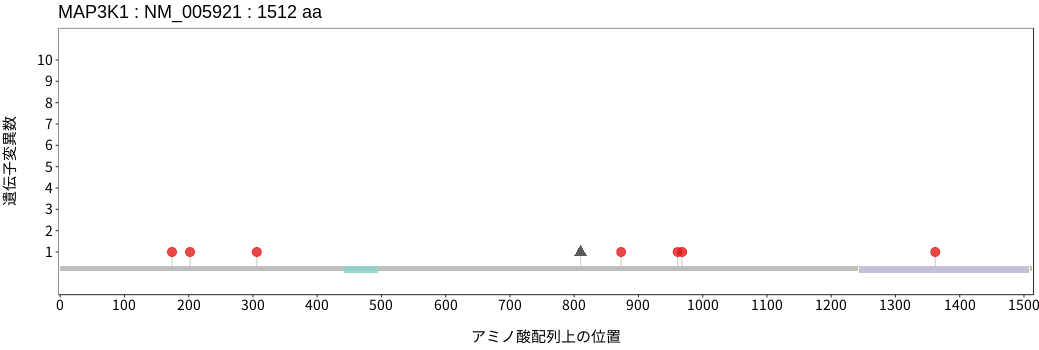
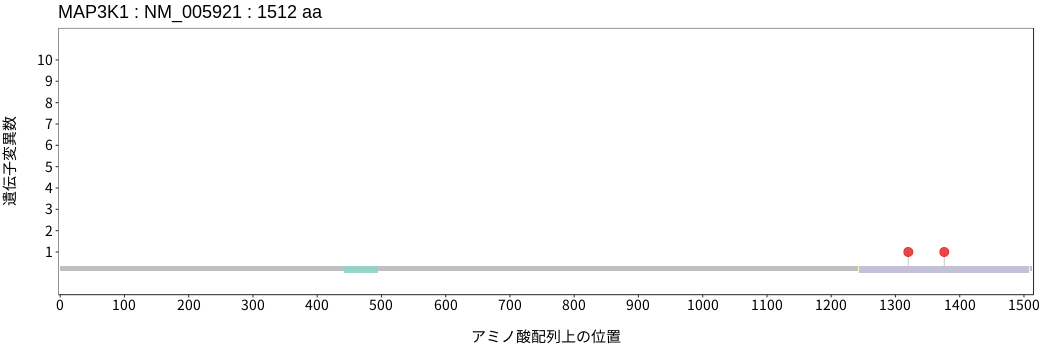
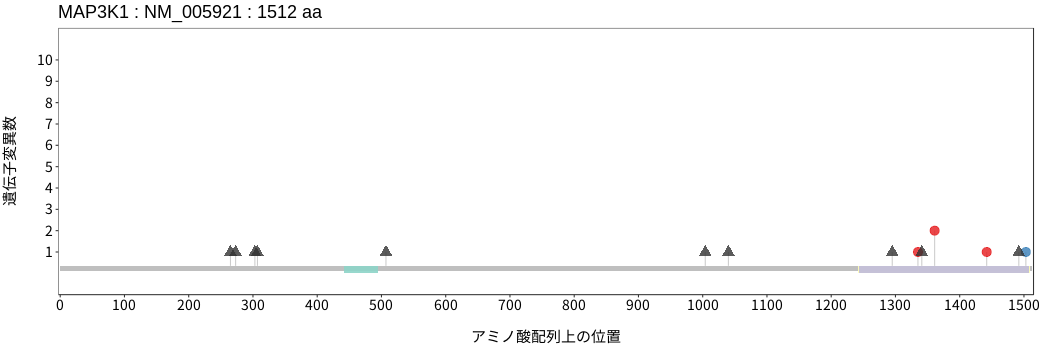
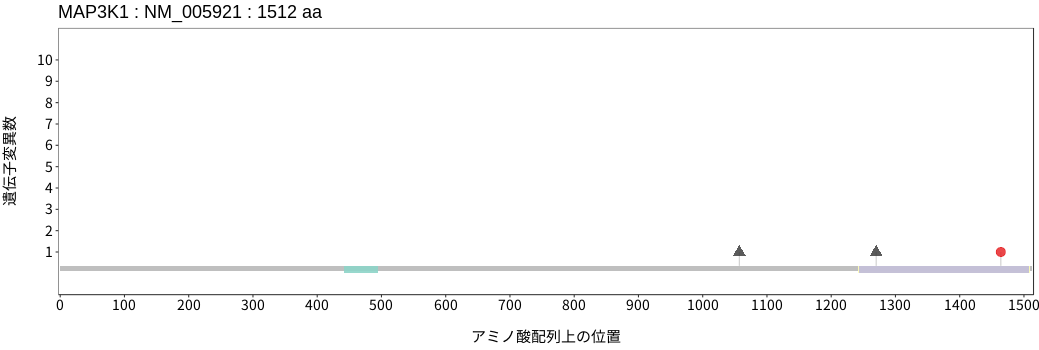
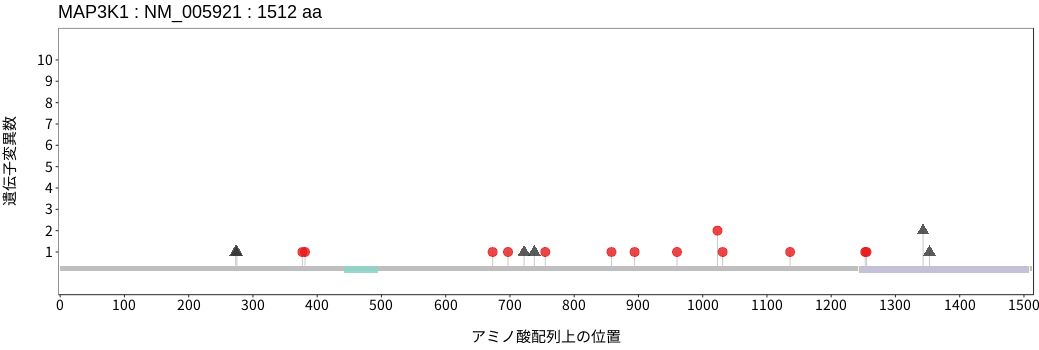
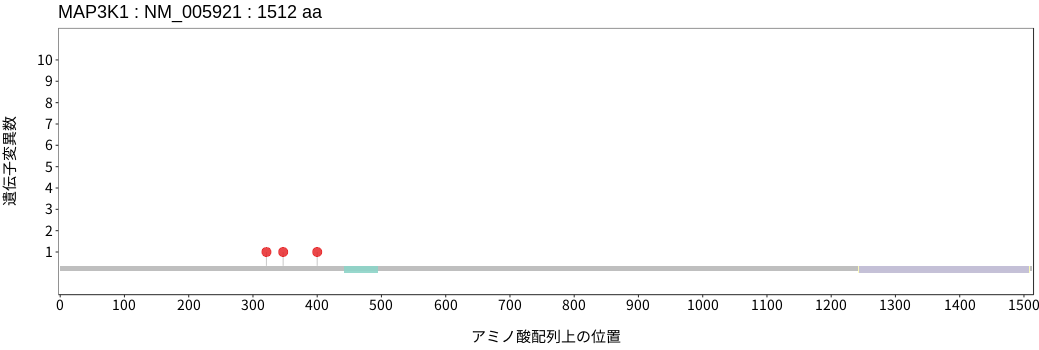
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr5:56152566 | C | T | 2 / 20 | p.R208* | c.622C>T | COSV68125794 | Tier2 | 1 / 106 |
| chr5:56155699 | CAG | C | 3 / 20 | p.E265fs | c.793_794delGA | COSV99063982 | Tier2 | 1 / 106 |
| chr5:56155720 | GAA | G | 3 / 20 | p.R273fs | c.816_817delAA | COSV68126639 | Tier2 | 1 / 106 |
| chr5:56155724 | AAG | A | 3 / 20 | p.R273fs | c.819_820delAG | COSV68121778 | Tier2 | 1 / 106 |
| chr5:56155728 | G | GTT | 3 / 20 | p.S275fs | c.822_823dupTT | Tier2 | 1 / 106 | |
| chr5:56160633 | GAA | G | 4 / 20 | p.E303fs | c.909_910delAA | Tier2 | 1 / 106 | |
| chr5:56160643 | GC | G | 4 / 20 | p.R307fs | c.919delC | COSV68125708 | Tier2 | 1 / 106 |
| chr5:56160653 | C | CAAAGTGATGCG | 4 / 20 | p.A314fs | c.928_938dupAAAGTGATGCG | Tier2 | 1 / 106 | |
| chr5:56161270 | T | G | 5 / 20 | p.L380* | c.1139T>G | Tier2 | 1 / 106 | |
| chr5:56168547 | C | G | 8 / 20 | p.Y501* | c.1503C>G | Tier2 | 1 / 106 | |
| chr5:56168663 | CA | C | 9 / 20 | p.S507fs | c.1519delA | Tier2 | 1 / 106 | |
| chr5:56168740 | C | T | 9 / 20 | p.R532* | c.1594C>T | COSV68123693 | Tier2 | 1 / 106 |
| chr5:56176614 | G | T | 12 / 20 | p.E722* | c.2164G>T | COSV68128615 | Tier2 | 1 / 106 |
| chr5:56176943 | T | G | 13 / 20 | p.L738* | c.2213T>G | Tier2 | 1 / 106 | |
| chr5:56176963 | C | T | 13 / 20 | p.Q745* | c.2233C>T | Tier2 | 1 / 106 | |
| chr5:56177456 | C | A | 14 / 20 | p.S810* | c.2429C>A | Tier2 | 1 / 106 | |
| chr5:56178036 | GCATAGACT | G | 14 / 20 | p.H1004fs | c.3010_3017delCATAGACT | Tier2 | 1 / 106 | |
| chr5:56178063 | C | A | 14 / 20 | p.C1012* | c.3036C>A | Tier2 | 1 / 106 | |
| chr5:56178143 | CA | C | 14 / 20 | p.D1040fs | c.3117delA | Tier2 | 1 / 106 | |
| chr5:56178195 | CAT | C | 14 / 20 | p.I1057fs | c.3170_3171delTA | Tier2 | 1 / 106 | |
| chr5:56178470 | C | CTT | 14 / 20 | p.K1150fs | c.3446_3447dupTT | Tier2 | 1 / 106 | |
| chr5:56179492 | ATGGC | A | 15 / 20 | p.A1270fs | c.3808_3811delGCTG | Tier2 | 1 / 106 | |
| chr5:56180553 | GAT | G | 16 / 20 | p.I1295fs | c.3884_3885delTA | COSV68123228 | Tier2 | 1 / 106 |
| chr5:56181789 | A | ATGGAGCCT | 17 / 20 | p.F1341fs | c.4015_4022dupGGAGCCTT | Tier2 | 1 / 106 | |
| chr5:56181803 | G | T | 17 / 20 | p.E1343* | c.4027G>T | COSV68127321 | Tier2 | 2 / 106 |
| chr5:56181834 | T | A | 17 / 20 | p.L1353* | c.4058T>A | Tier2 | 1 / 106 | |
| chr5:56183234 | C | T | 18 / 20 | p.Q1382* | c.4144C>T | COSV68126240 | Tier2 | 1 / 106 |
| chr5:56189442 | C | T | 20 / 20 | p.Q1492* | c.4474C>T | COSV68127064 | Tier2 | 1 / 106 |