Side Menu
NOTCH3
基本情報
- 遺伝子名
-
notch receptor 3
- 慣用名
-
CADASIL, CADASIL1, CASIL, IMF2, LMNS
- 遺伝子分類
-
がん遺伝子 / がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
分化
- シグナル伝達経路
-
NOTCH
- 染色体上の位置 (GRCh37/hg19)
-
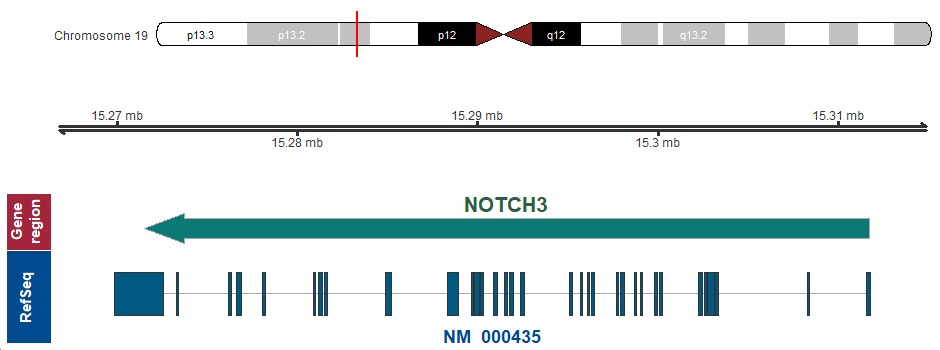
19p13.12 (chr19:15271473..15311716, complement)
- アミノ酸配列の長さ
-
2,321
遺伝子マップ
解説
NOTCH3はNOTCHファミリー受容体の一つであり、隣接した細胞間のシグナル伝達に関与し細胞の分化制御に働く。NOTCH3受容体と隣接する細胞のリガンドとの相互作用が起きることで、NOTCH3はγ-セクレターゼで分解され、遊離した細胞内ドメインが転写制御に働く。NOTCH3はがん遺伝子およびがん抑制遺伝子で働く二面性を有しており、それはがん種によって異なることが知られている。
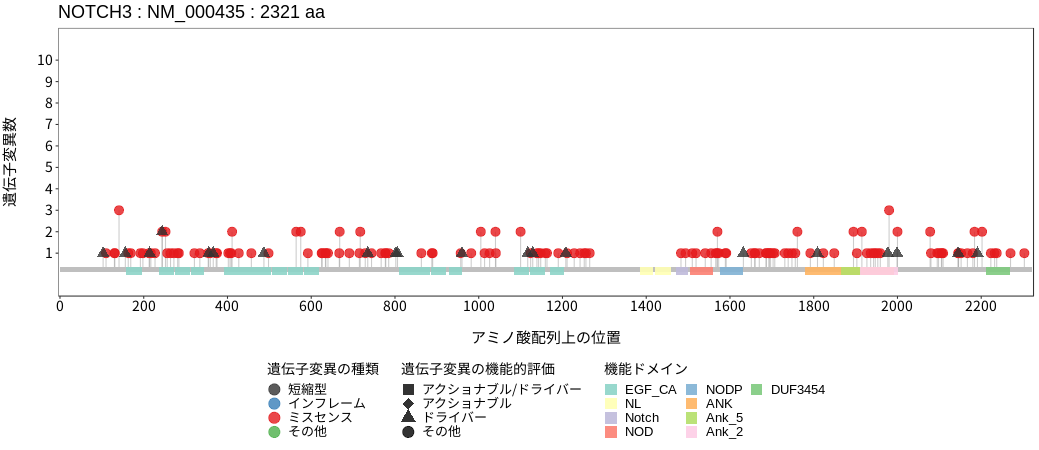
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

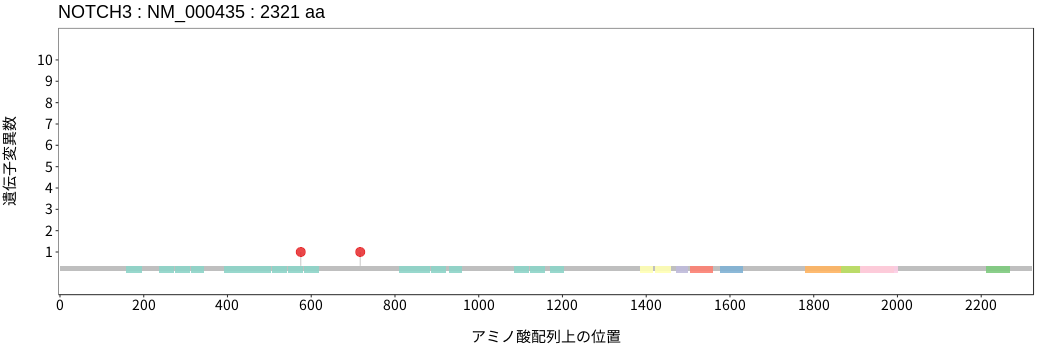
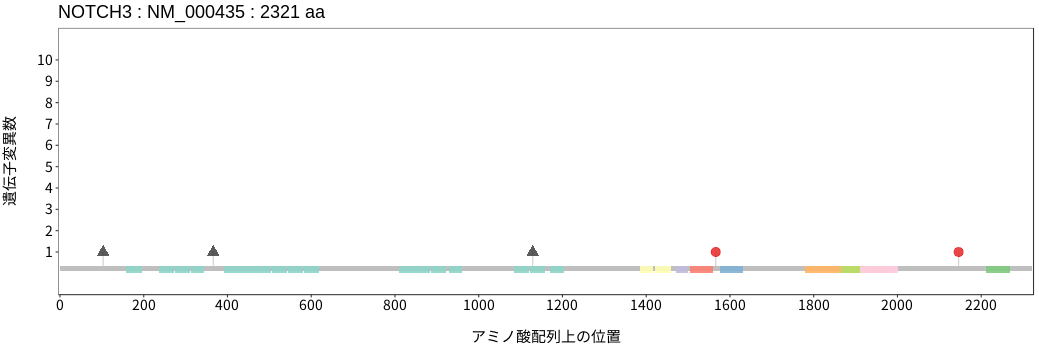
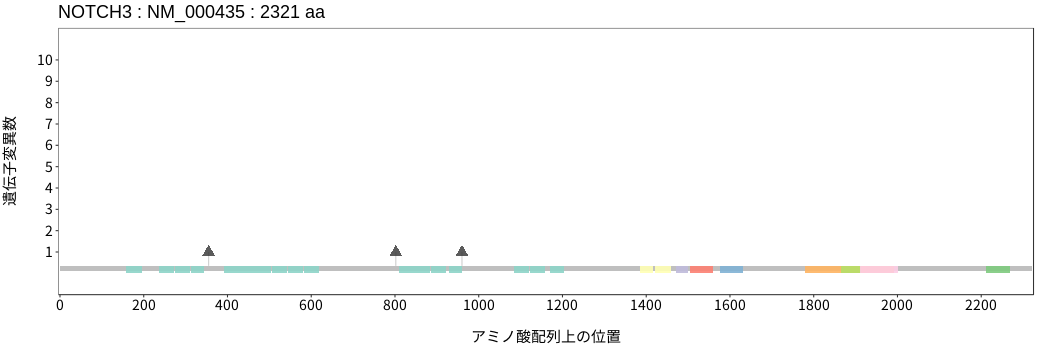
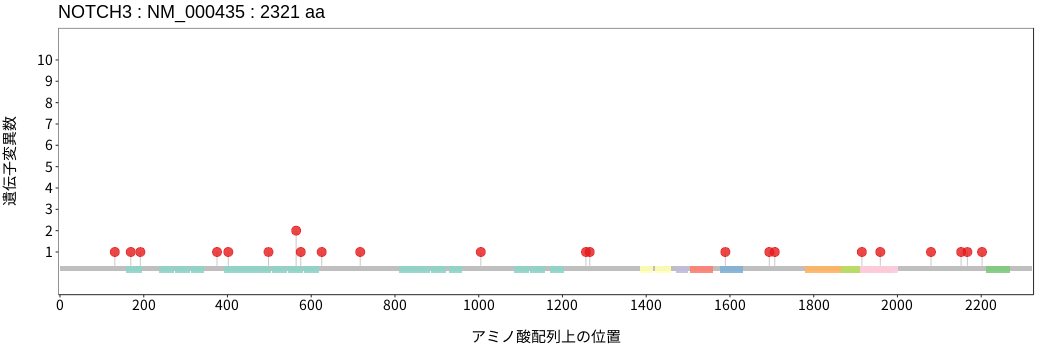
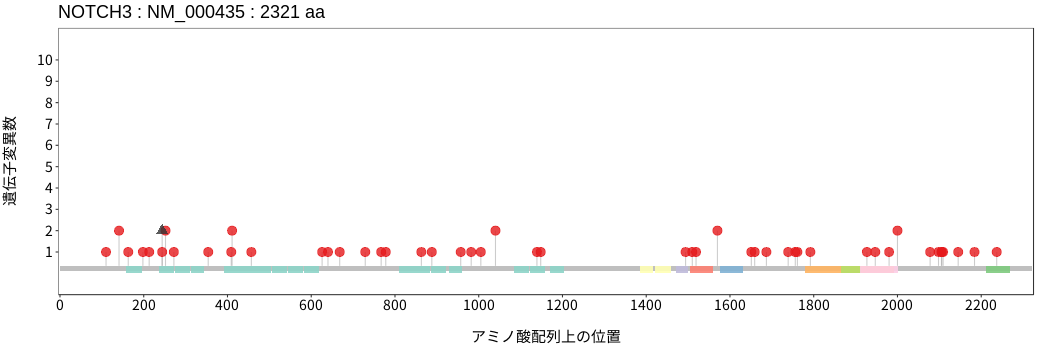
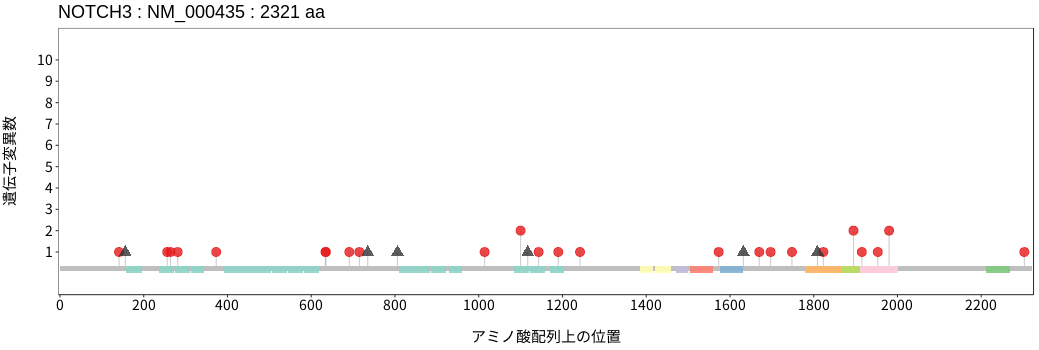
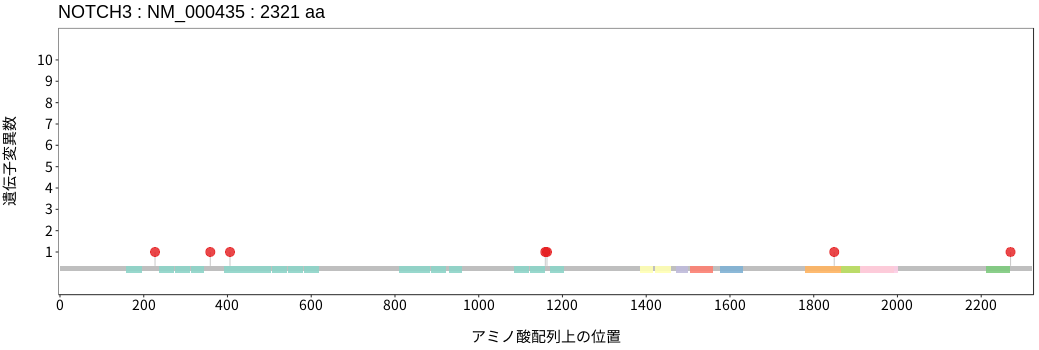
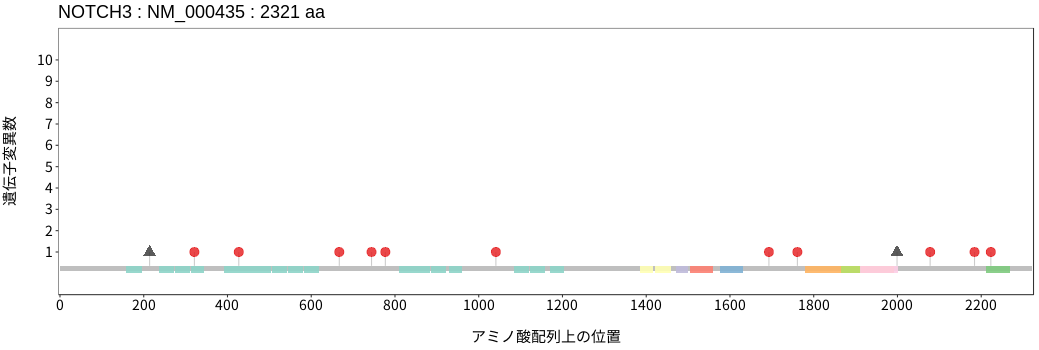
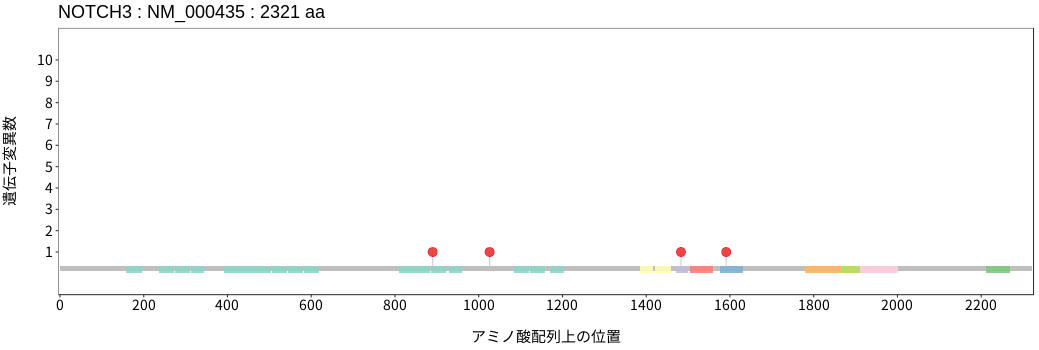
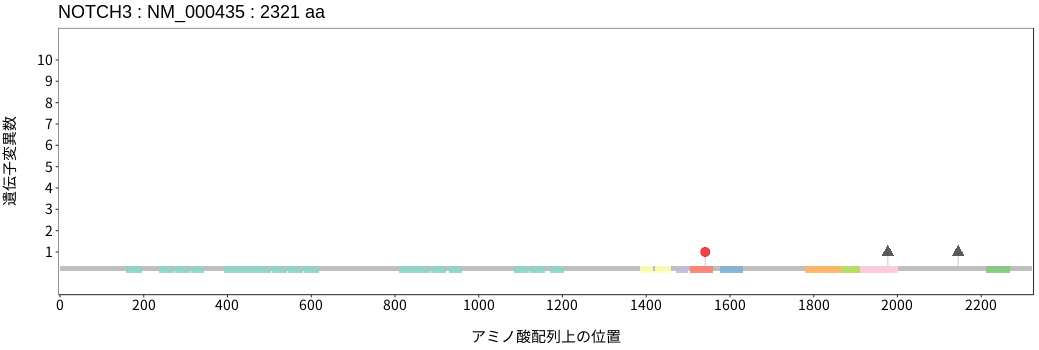
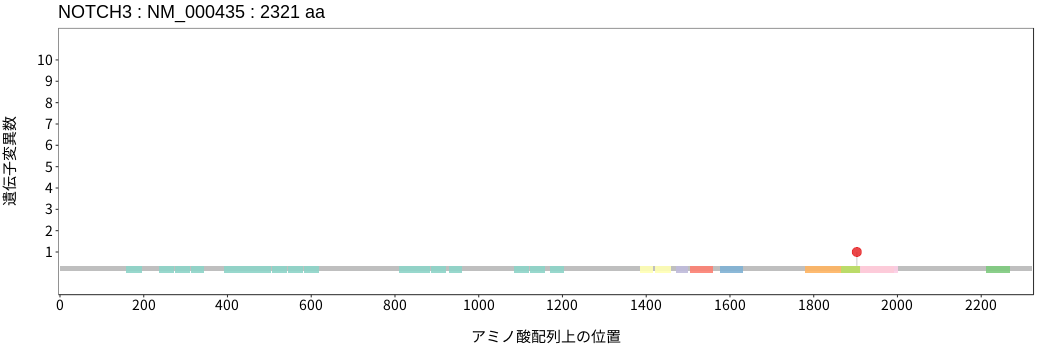
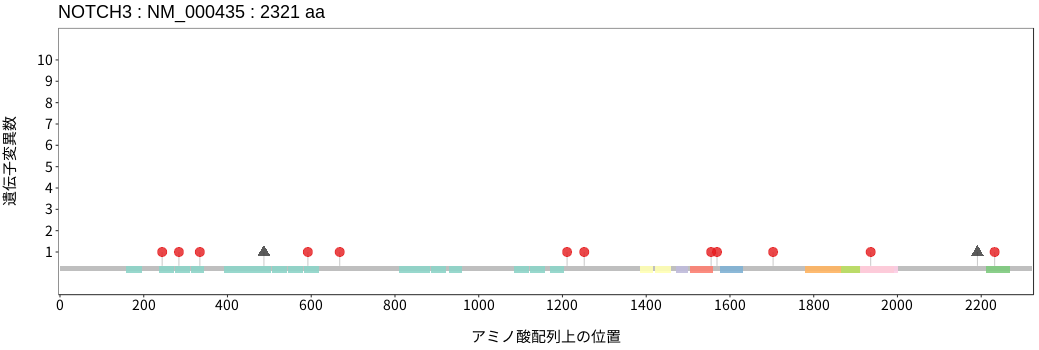
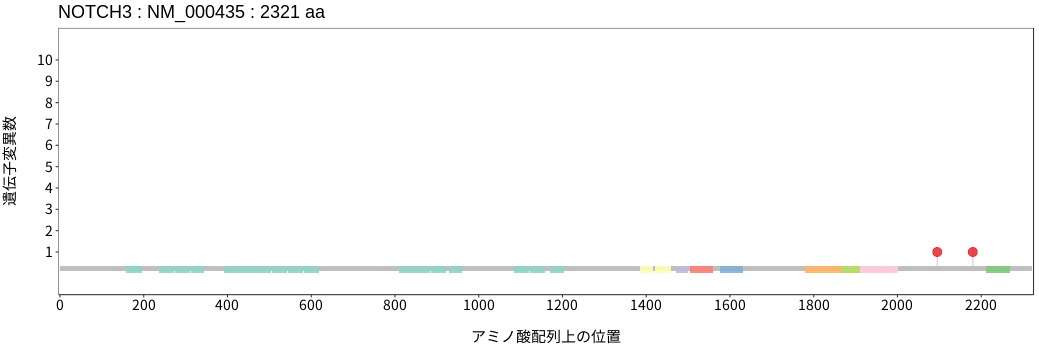
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
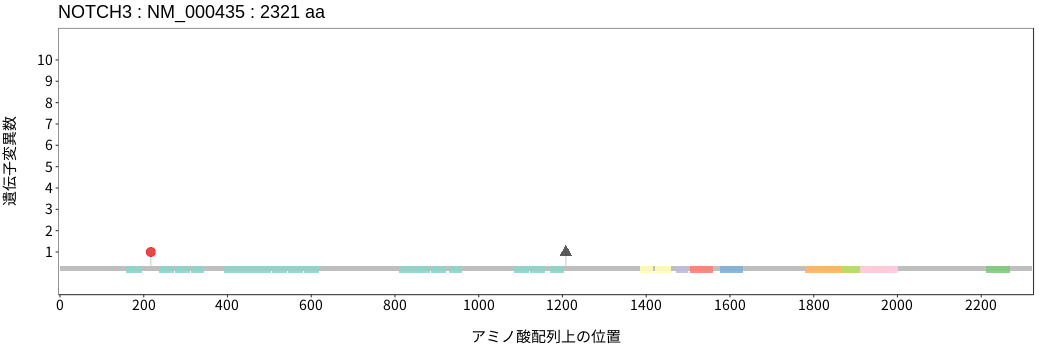
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr19:15303221 | G | A | 3 / 33 | p.R103* | c.307C>T | COSV54655561 | Tier2 | 1 / 213 |
| chr19:15302810 | G | A | 4 / 33 | p.Q214* | c.640C>T | Tier2 | 1 / 213 | |
| chr19:15302984 | G | A | 4 / 33 | p.R156* | c.466C>T | COSV54631988 | Tier2 | 1 / 213 |
| chr19:15302628 | G | A | 5 / 33 | p.R244* | c.730C>T | Tier2 | 2 / 213 | |
| chr19:15300181 | G | GAT | 7 / 33 | p.C366fs | c.1093_1094dupAT | Tier2 | 1 / 213 | |
| chr19:15300211 | A | T | 7 / 33 | p.C355* | c.1065T>A | Tier2 | 1 / 213 | |
| chr19:15299079 | G | A | 9 / 33 | p.R487* | c.1459C>T | COSV54646925 | Tier2 | 1 / 213 |
| chr19:15296161 | G | A | 14 / 33 | p.R735* | c.2203C>T | COSV54639420 | Tier2 | 1 / 213 |
| chr19:15295721 | C | T | 15 / 33 | p.W802* | c.2406G>A | Tier2 | 1 / 213 | |
| chr19:15295256 | G | A | 16 / 33 | p.R806* | c.2416C>T | COSV54647420 | Tier2 | 1 / 213 |
| chr19:15291888 | G | A | 18 / 33 | p.Q960* | c.2878C>T | Tier2 | 1 / 213 | |
| chr19:15290250 | G | A | 21 / 33 | p.Q1129* | c.3385C>T | Tier2 | 1 / 213 | |
| chr19:15290282 | TTA | T | 21 / 33 | p.D1117fs | c.3351_3352delTA | Tier2 | 1 / 213 | |
| chr19:15289932 | C | A | 22 / 33 | p.E1208* | c.3622G>T | Tier2 | 1 / 213 | |
| chr19:15281362 | C | A | 27 / 33 | p.E1632* | c.4894G>T | Tier2 | 1 / 213 | |
| chr19:15276840 | C | A | 30 / 33 | p.E1809* | c.5425G>T | Tier2 | 1 / 213 | |
| chr19:15271868 | G | GT | 33 / 33 | p.P2191fs | c.6570_6571insA | Tier2 | 1 / 213 | |
| chr19:15272001 | CGCCCG | C | 33 / 33 | p.R2145fs | c.6433_6437delCGGGC | Tier2 | 1 / 213 | |
| chr19:15272444 | C | A | 33 / 33 | p.E1999* | c.5995G>T | Tier2 | 1 / 213 | |
| chr19:15272483 | CATAGCTGCCCTCGCGGGCGGCCAGGA | C | 33 / 33 | p.F1977fs | c.5930_5955delTCCTGGCCGCCCGCGAGGGCAGCTAT | Tier2 | 1 / 213 |