Side Menu
PIK3R1
基本情報
- 遺伝子名
-
phosphoinositide-3-kinase regulatory subunit 1
- 慣用名
-
AGM7, GRB1, IMD36, p85-ALPHA
- 遺伝子分類
-
がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
腫瘍形成・増殖
- シグナル伝達経路
-
PI3K/Akt/mTOR
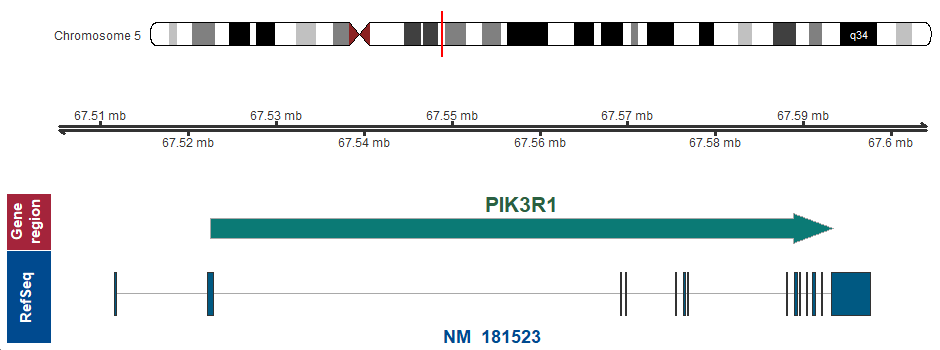
- 染色体上の位置 (GRCh37/hg19)
-
5q13.1 (chr5:67522504..67593429)
- アミノ酸配列の長さ
-
724
遺伝子マップ
解説
PIK3R1はホスファチジルイノシトール-3キナーゼ (PI3K)の調節サブユニットp85αであり、PI3Kの触媒サブユニットp110α (PIK3CA)の活性の安定化および阻害する。また、PI3Kの逆反応を司るPTENに直接結合し、そのフォスファターゼ活性を増強する。そのため、PI3K/Akt/mTORシグナル経路における抑制因子として働き、その機能喪失型バリアントではPI3K/Akt/mTORシグナル経路の活性化が引き起こされる。PIK3R1のバリアントは子宮内膜癌、神経膠腫、および大腸癌などにおいて認められる。
シグナル伝達経路

遺伝子変異総量(Tumor Mutation Burden)の分布TMB

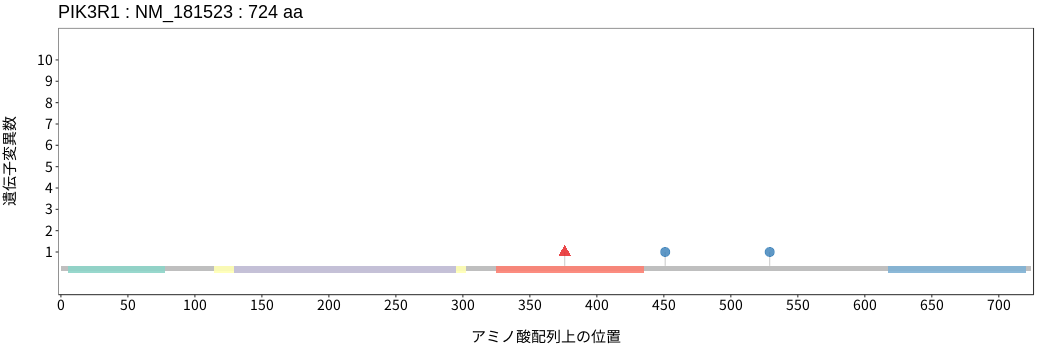
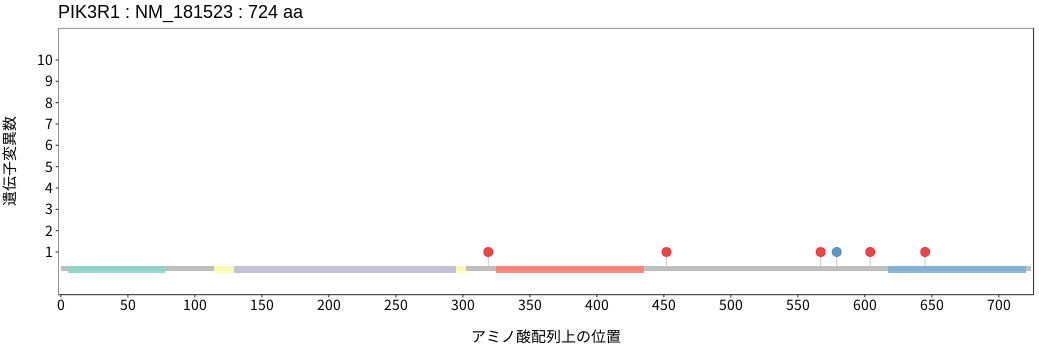
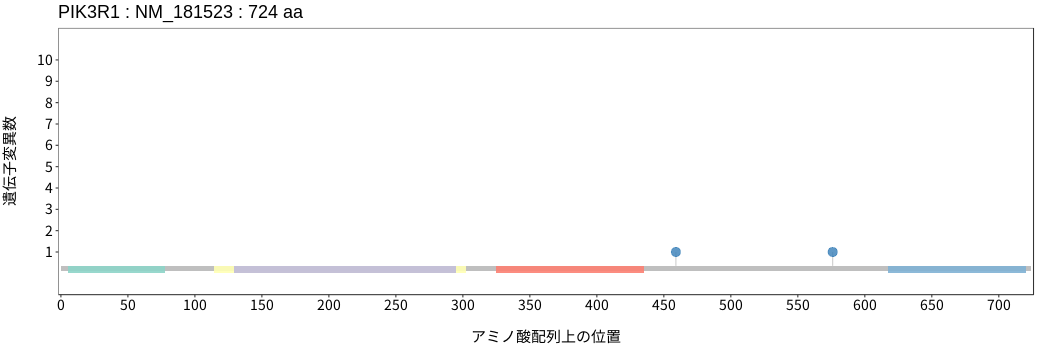
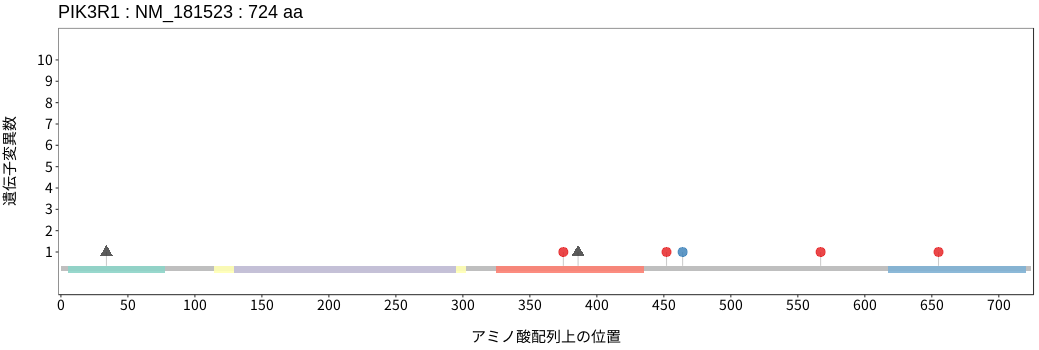
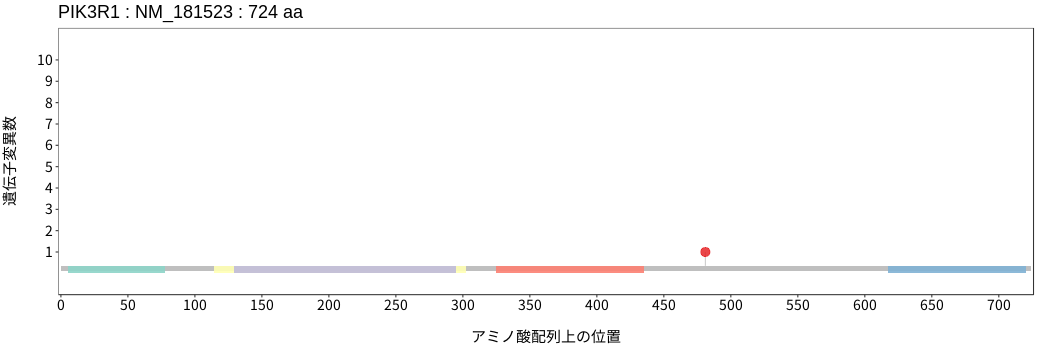
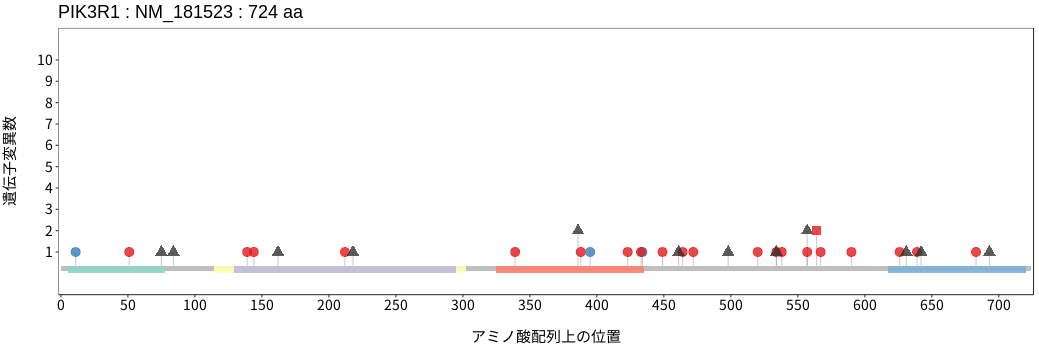
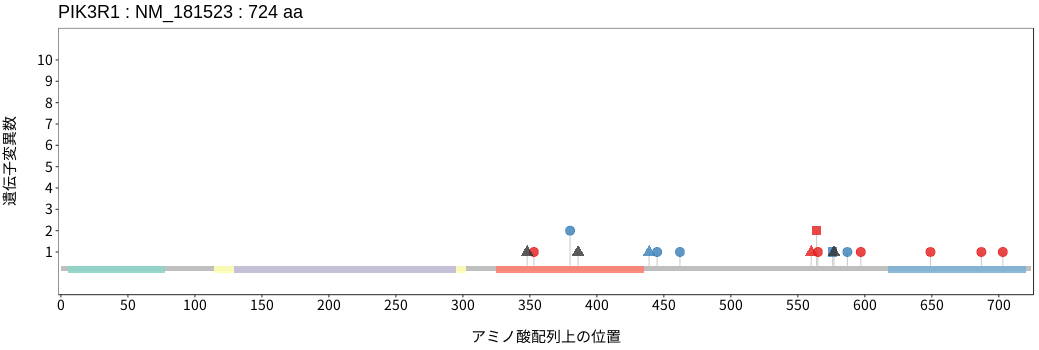
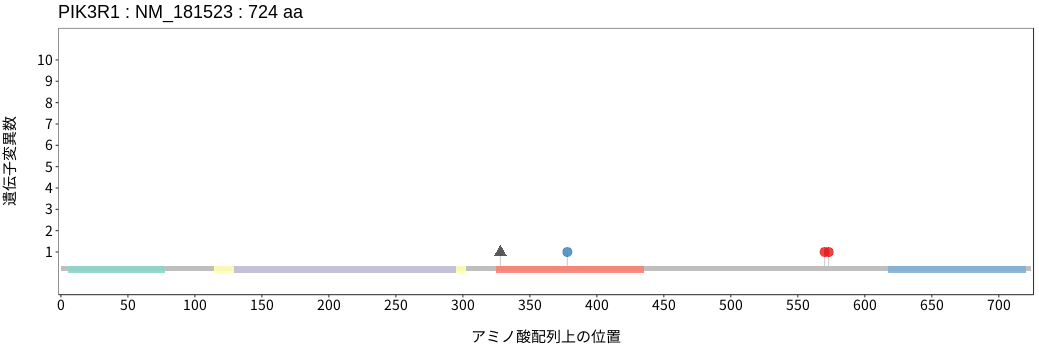
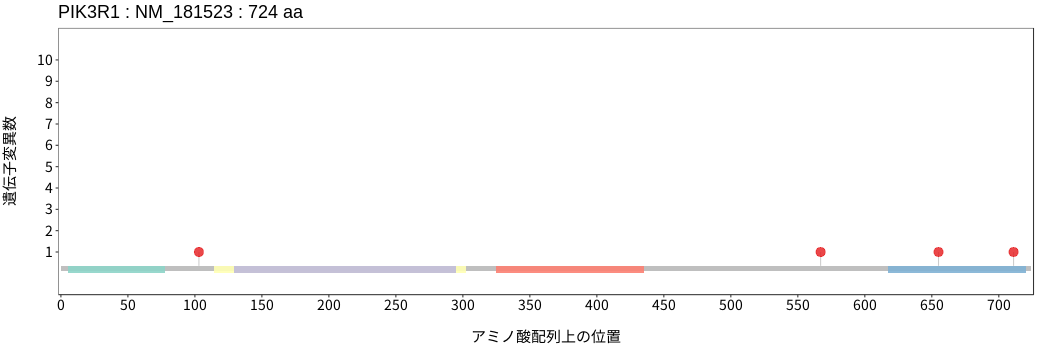
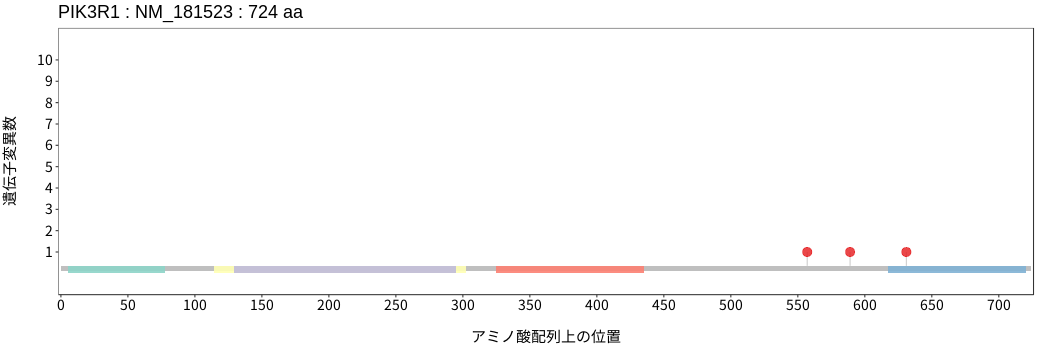
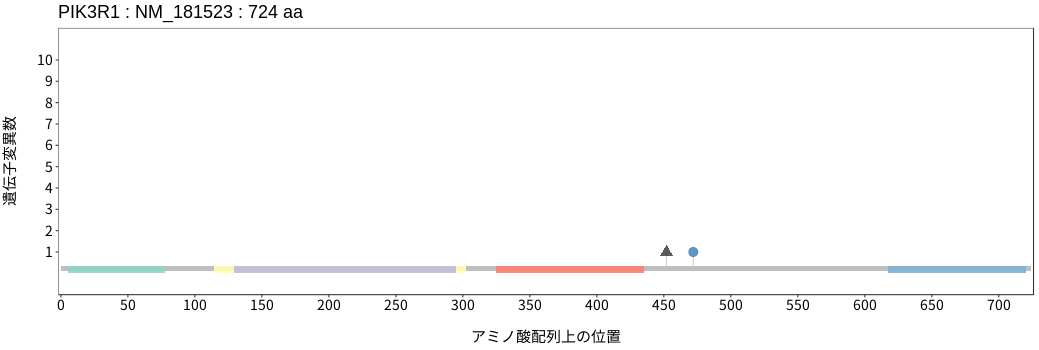
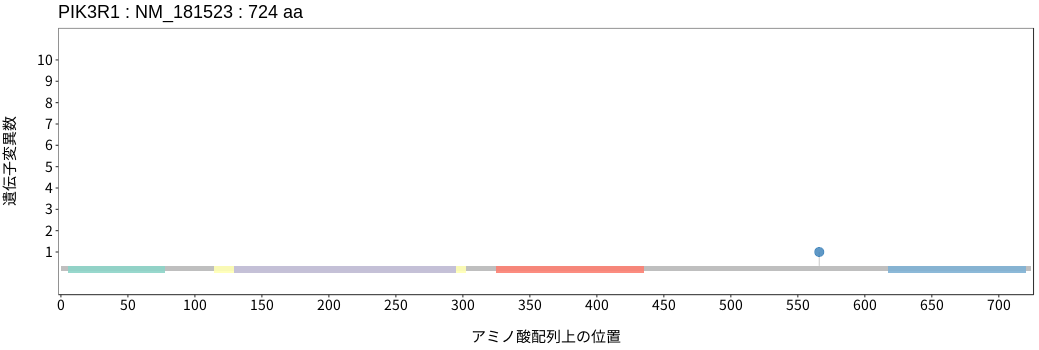
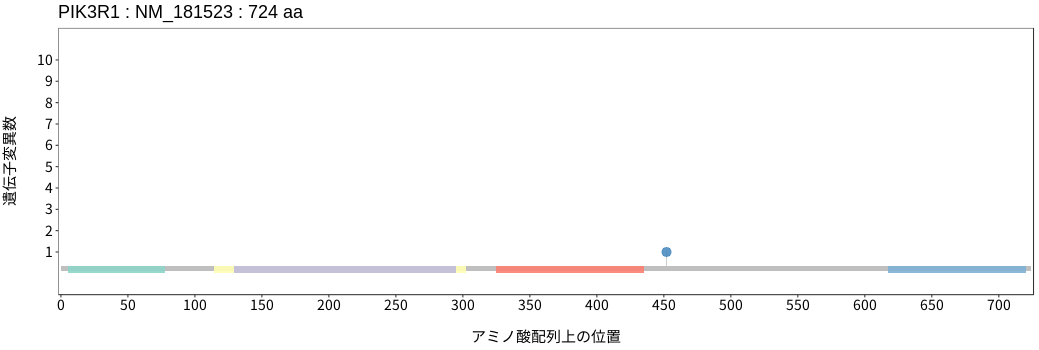
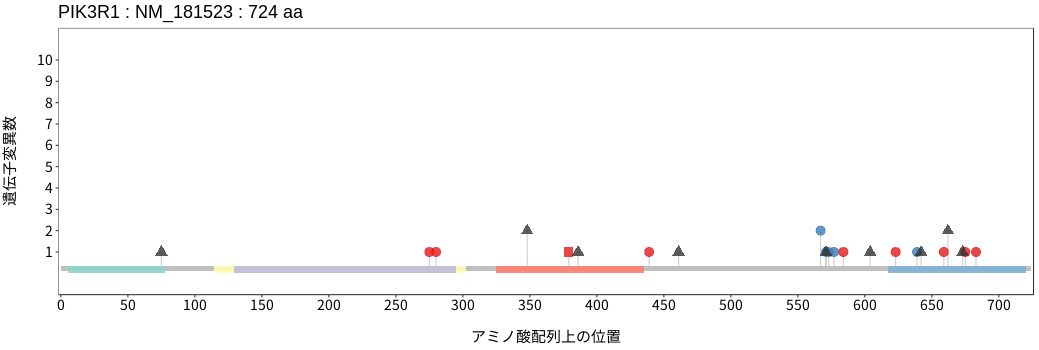
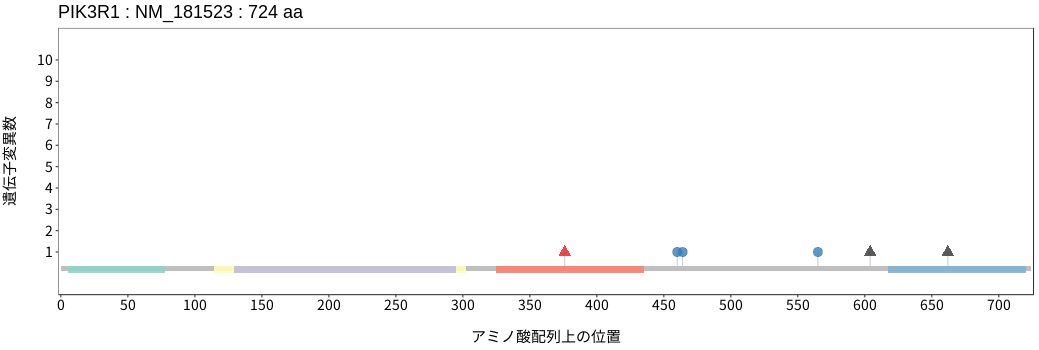
アミノ酸配列上の変異分布
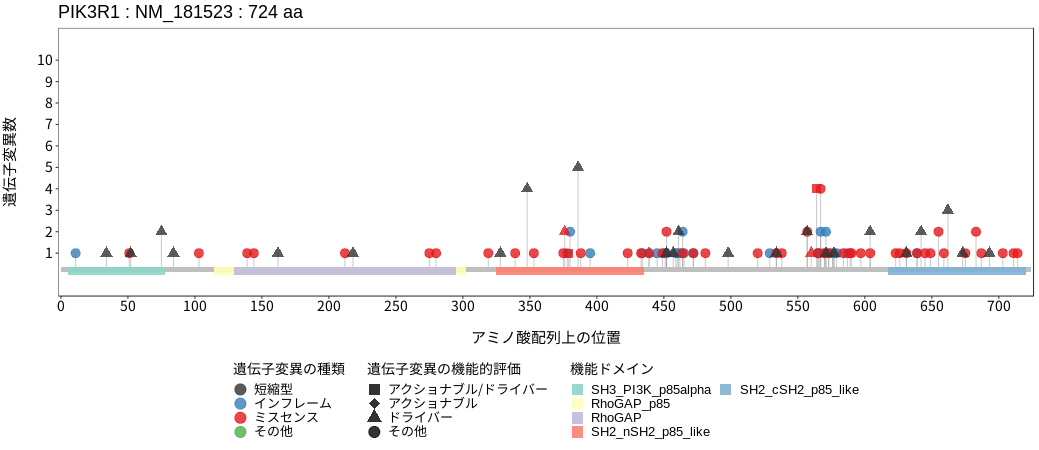
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr5:67522602 | TAA | T | 2 / 16 | p.K34fs | c.101_102delAA | Tier2 | 1 / 135 | |
| chr5:67522657 | G | T | 2 / 16 | p.E52* | c.154G>T | COSV57124346 | Tier2 | 1 / 135 |
| chr5:67522726 | G | T | 2 / 16 | p.E75* | c.223G>T | Tier2 | 2 / 135 | |
| chr5:67522748 | TCTCGC | T | 2 / 16 | p.P84fs | c.249_253delGCCTC | Tier2 | 1 / 135 | |
| chr5:67569823 | C | T | 4 / 16 | p.R162* | c.484C>T | COSV57127263 | Tier2 | 1 / 135 |
| chr5:67576373 | G | T | 6 / 16 | p.E218* | c.652G>T | COSV57137973 | Tier2 | 1 / 135 |
| chr5:67588153 | TA | T | 8 / 16 | p.L328fs | c.984delA | Tier2 | 1 / 135 | |
| chr5:67588951 | C | T | 9 / 16 | p.R348* | c.1042C>T | COSV57123475 | Tier1 | 4 / 135 |
| chr5:67589138 | G | C | 10 / 16 | p.G376R | c.1126G>C | COSV57133701 | Tier2 | 1 / 135 |
| chr5:67589138 | G | A | 10 / 16 | p.G376R | c.1126G>A | COSV57123316 | Tier2 | 1 / 135 |
| chr5:67589147 | A | G | 10 / 16 | p.K379E | c.1135A>G | COSV57124239 | Tier1 | 1 / 135 |
| chr5:67589168 | C | T | 10 / 16 | p.R386* | c.1156C>T | COSV57126203 | Tier2 | 5 / 135 |
| chr5:67589549 | AAAG | A | 11 / 16 | p.E439del | c.1317_1319delAGA | COSV57133720 | Tier2 | 1 / 135 |
| chr5:67589593 | T | A | 11 / 16 | p.Y452* | c.1356T>A | Tier2 | 1 / 135 | |
| chr5:67589606 | C | T | 11 / 16 | p.Q457* | c.1369C>T | Tier2 | 1 / 135 | |
| chr5:67589618 | C | T | 11 / 16 | p.R461* | c.1381C>T | COSV57125001 | Tier2 | 2 / 135 |
| chr5:67590432 | C | A | 12 / 16 | p.C498* | c.1494C>A | COSV57139021 | Tier2 | 1 / 135 |
| chr5:67591007 | C | T | 13 / 16 | p.R534* | c.1600C>T | COSV57137455 | Tier2 | 1 / 135 |
| chr5:67591076 | C | T | 13 / 16 | p.R557* | c.1669C>T | COSV57125577 | Tier2 | 2 / 135 |
| chr5:67591085 | G | T | 13 / 16 | p.D560Y | c.1678G>T | COSV57123942 | Tier2 | 1 / 135 |
| chr5:67591097 | A | G | 13 / 16 | p.N564D | c.1690A>G | COSV57124003 | Tier1 | 4 / 135 |
| chr5:67591113 | A | ACCTT | 13 / 16 | p.I571fs | c.1707_1710dupCCTT | Tier2 | 1 / 135 | |
| chr5:67591131 | AGAC | A | 13 / 16 | p.T576del | c.1727_1729delCGA | COSV57124497 | Tier1 | 1 / 135 |
| chr5:67591135 | GAGAGACCAATACTTGATGT | G | 13 / 16 | p.R577fs | c.1730_1745+3delGAGACCAATACTTGATGTA | COSV57137283 | Tier2 | 1 / 135 |
| chr5:67591312 | G | T | 14 / 16 | p.E604* | c.1810G>T | Tier2 | 2 / 135 | |
| chr5:67592075 | C | T | 15 / 16 | p.R631* | c.1891C>T | COSV57134805 | Tier2 | 1 / 135 |
| chr5:67592108 | C | T | 15 / 16 | p.R642* | c.1924C>T | COSV57123056 | Tier2 | 2 / 135 |
| chr5:67592163 | C | CTG | 15 / 16 | p.V662fs | c.1981_1982dupGT | COSV99140531 | Tier2 | 2 / 135 |
| chr5:67592165 | G | GT | 15 / 16 | p.V662fs | c.1982dupT | Tier2 | 1 / 135 | |
| chr5:67593269 | TAAAC | T | 16 / 16 | p.N673fs | c.2019_2022delCAAA | COSV99073799 | Tier2 | 1 / 135 |
| chr5:67593331 | G | T | 16 / 16 | p.E693* | c.2077G>T | Tier2 | 1 / 135 |