Side Menu
RBM10
基本情報
- 遺伝子名
-
RNA binding motif protein 10
- 慣用名
-
DXS8237E, GPATC9, GPATCH9, S1-1, TARPS, ZRANB5
- 遺伝子分類
-
がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
転写制御
- シグナル伝達経路
-
RNA metabolism
- 染色体上の位置 (GRCh37/hg19)
-
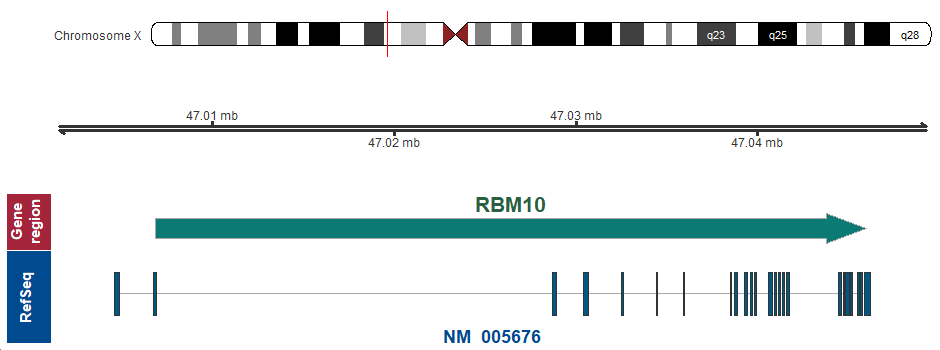
Xp11.3 (chrX:47006881..47045998)
- アミノ酸配列の長さ
-
930
遺伝子マップ
解説
RBM10は選択的スプライシングの調節に働くRNA結合モチーフを有する核タンパクである。NOTCHの抑制因子であるNUMB遺伝子のエキソン9のスキップを促進することで細胞の増殖を阻害を引き起こし、がん抑制遺伝子として機能することが報告されている。肺腺癌においてその変異頻度が高く、乳癌、結腸癌、卵巣がんにおいても変異が認められている。
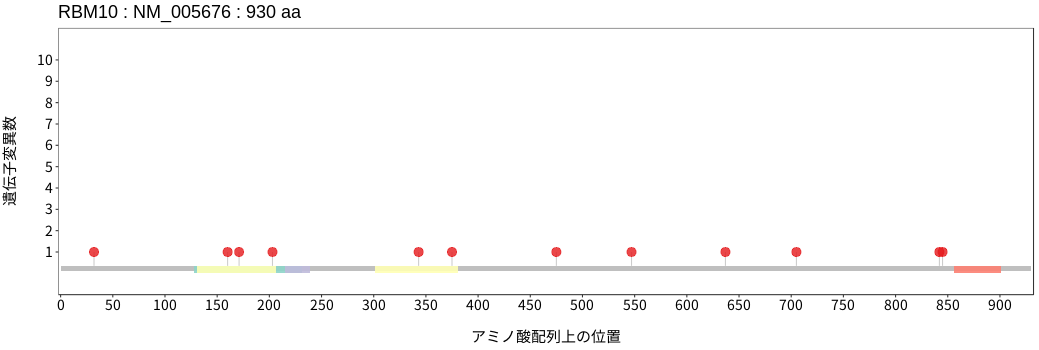
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

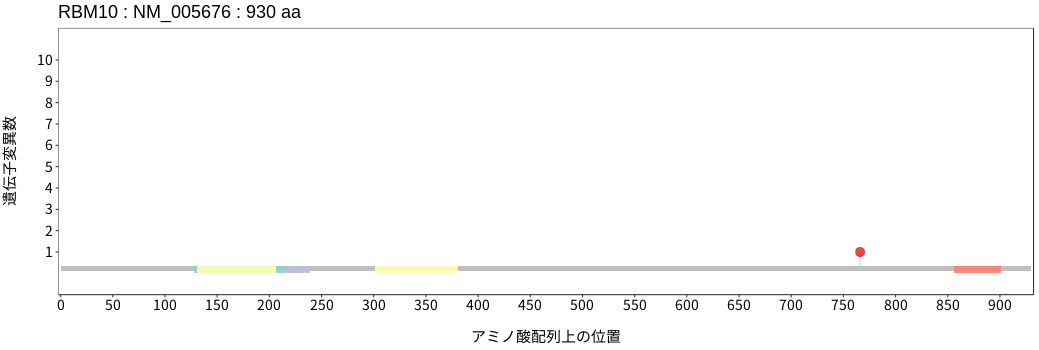
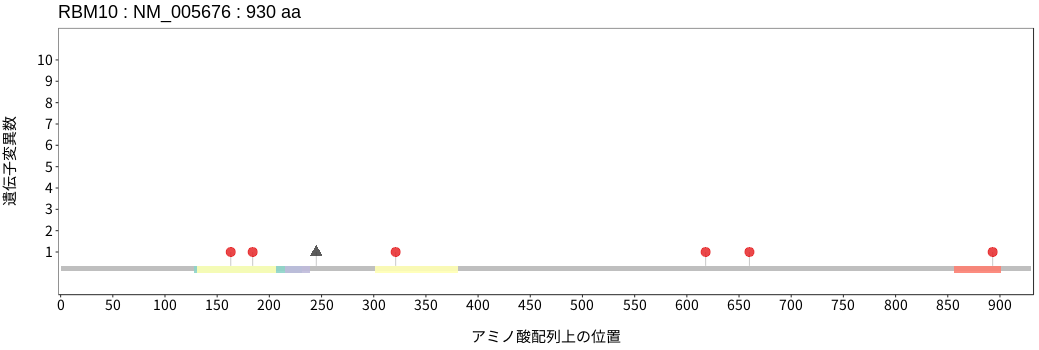
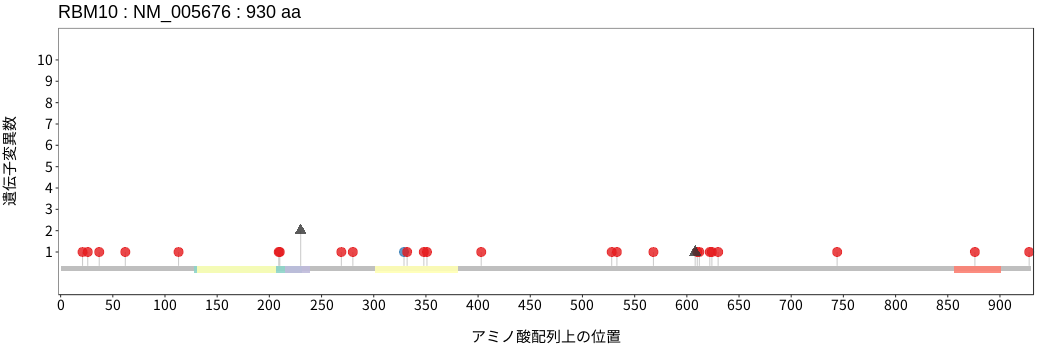
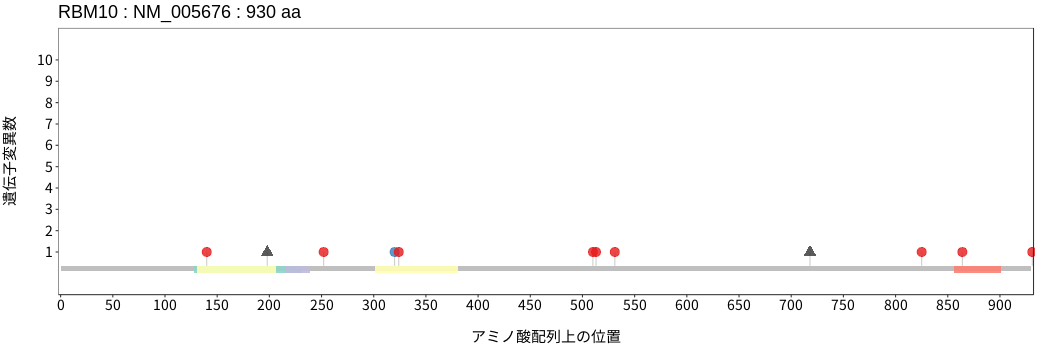
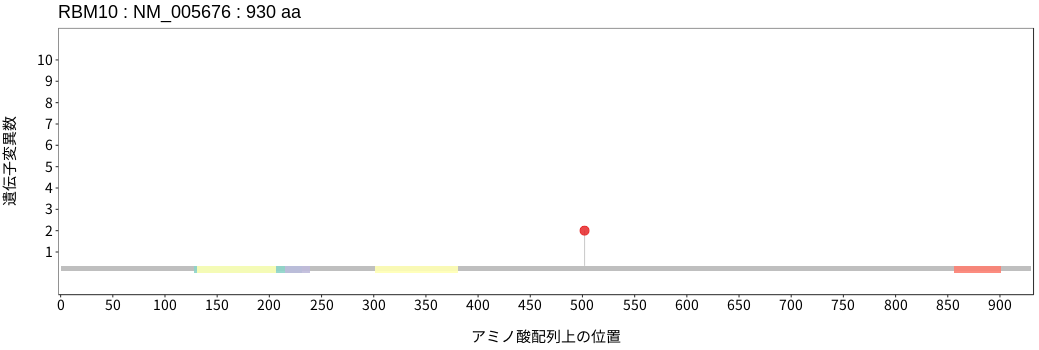
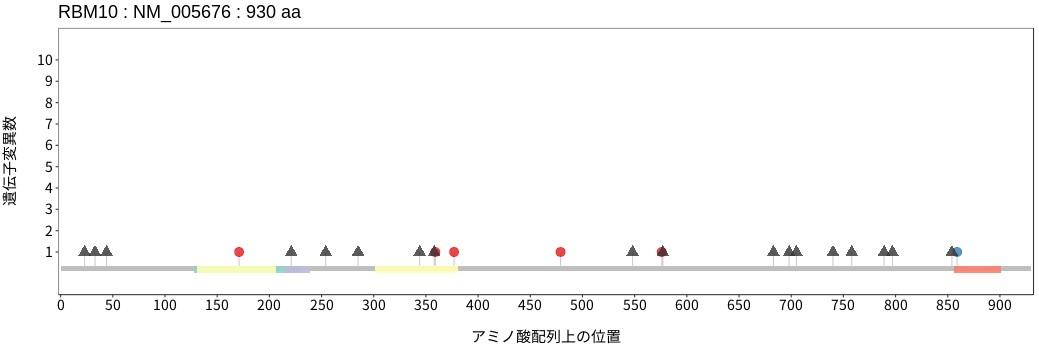
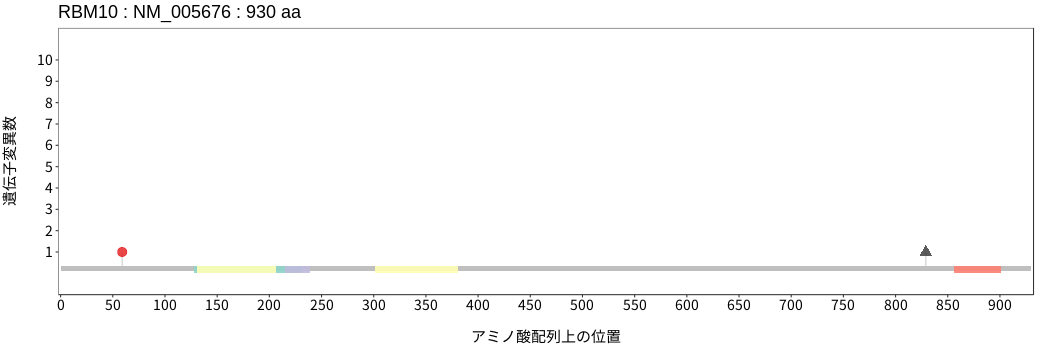
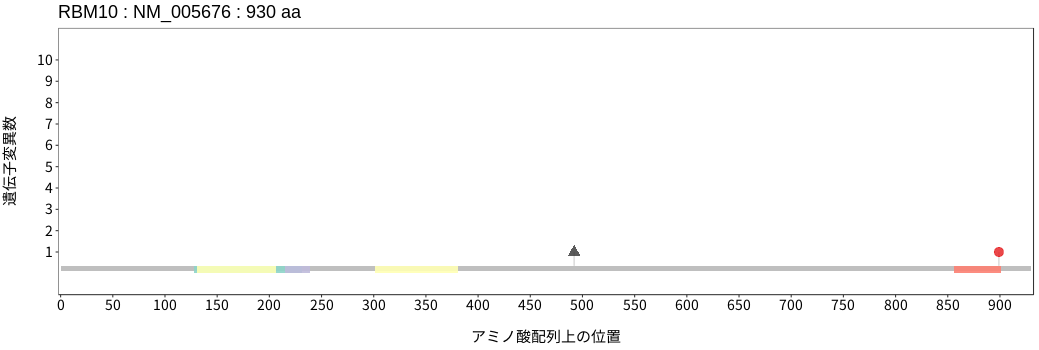
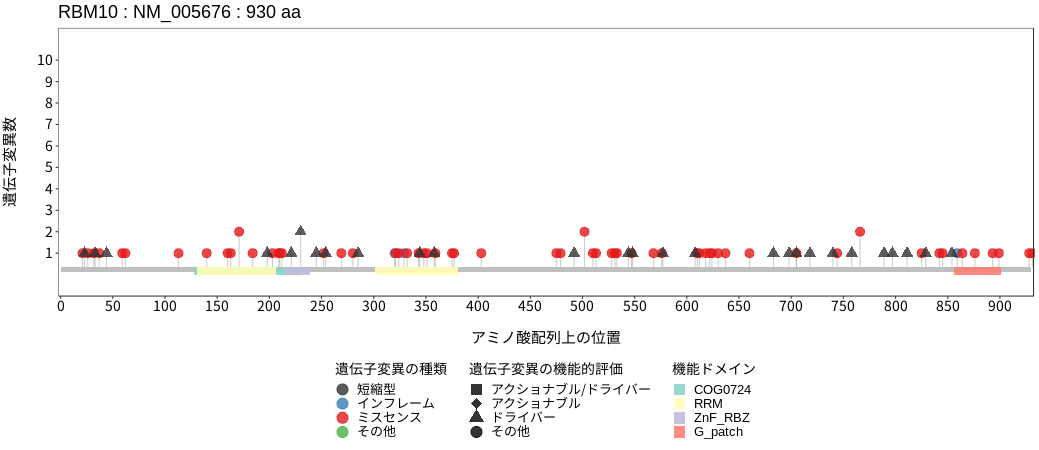
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chrX:47028763 | C | T | 3 / 24 | p.Q23* | c.67C>T | Tier2 | 1 / 127 | |
| chrX:47028790 | C | CGA | 3 / 24 | p.D33fs | c.97_98dupGA | Tier2 | 1 / 127 | |
| chrX:47028828 | T | G | 3 / 24 | p.Y44* | c.132T>G | COSV61309029 | Tier2 | 1 / 127 |
| chrX:47035912 | TC | T | 7 / 24 | p.L198fs | c.592delC | Tier2 | 1 / 127 | |
| chrX:47035983 | A | T | 7 / 24 | p.K221* | c.661A>T | Tier2 | 1 / 127 | |
| chrX:47038526 | C | T | 8 / 24 | p.R230* | c.688C>T | COSV61309636 | Tier2 | 2 / 127 |
| chrX:47038726 | C | T | 9 / 24 | p.Q245* | c.733C>T | COSV61313432 | Tier2 | 1 / 127 |
| chrX:47038751 | TG | T | 9 / 24 | p.D254fs | c.760delG | Tier2 | 1 / 127 | |
| chrX:47038846 | C | T | 9 / 24 | p.Q285* | c.853C>T | COSV61312003 | Tier2 | 1 / 127 |
| chrX:47039406 | CG | C | 10 / 24 | p.G344fs | c.1031delG | Tier2 | 1 / 127 | |
| chrX:47039613 | GGCAGCCCA | G | 11 / 24 | p.Q358fs | c.1068_1075delAGCCCAGC | Tier2 | 1 / 127 | |
| chrX:47040939 | CTG | C | 14 / 24 | p.S492fs | c.1473_1474delGT | Tier2 | 1 / 127 | |
| chrX:47041199 | ACC | A | 15 / 24 | p.H544fs | c.1629_1630delCC | Tier2 | 1 / 127 | |
| chrX:47041211 | TCTGCTCTCCCACCGGCTACCAGCCCCACTGCCCAGGAATC | T | 15 / 24 | p.A548fs | c.1642_1681delGCTCTCCCACCGGCTACCAGCCCCACTGCCCAGGAATCCT | Tier2 | 1 / 127 | |
| chrX:47041384 | GA | G | 16 / 24 | p.T577fs | c.1729delA | Tier2 | 1 / 127 | |
| chrX:47041599 | G | A | 17 / 24 | p.W608* | c.1824G>A | Tier2 | 1 / 127 | |
| chrX:47044550 | G | T | 18 / 24 | p.E683* | c.2047G>T | COSV61308334 | Tier2 | 1 / 127 |
| chrX:47044595 | G | T | 18 / 24 | p.E698* | c.2092G>T | COSV61307701 | Tier2 | 1 / 127 |
| chrX:47044713 | G | T | 19 / 24 | p.E705* | c.2113G>T | COSV61307871 | Tier2 | 1 / 127 |
| chrX:47044753 | GT | G | 19 / 24 | p.S718fs | c.2154delT | Tier2 | 1 / 127 | |
| chrX:47044892 | G | T | 20 / 24 | p.E740* | c.2218G>T | COSV61309228 | Tier2 | 1 / 127 |
| chrX:47044946 | A | T | 20 / 24 | p.K758* | c.2272A>T | Tier2 | 1 / 127 | |
| chrX:47045124 | G | T | 21 / 24 | p.E789* | c.2365G>T | COSV61309684 | Tier2 | 1 / 127 |
| chrX:47045149 | C | G | 21 / 24 | p.S797* | c.2390C>G | Tier2 | 1 / 127 | |
| chrX:47045464 | C | T | 22 / 24 | p.Q811* | c.2431C>T | Tier2 | 1 / 127 | |
| chrX:47045518 | G | T | 22 / 24 | p.E829* | c.2485G>T | COSV61311914 | Tier2 | 1 / 127 |
| chrX:47045678 | GGACGGGCTGGGCA | G | 23 / 24 | p.D854fs | c.2561_2573delACGGGCTGGGCAG | Tier2 | 1 / 127 |