Side Menu
RNF43
基本情報
- 遺伝子名
-
ring finger protein 43
- 慣用名
-
RNF124, SSPCS, URCC
- 遺伝子分類
-
がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
分化
- シグナル伝達経路
-
WNT
- 染色体上の位置 (GRCh37/hg19)
-
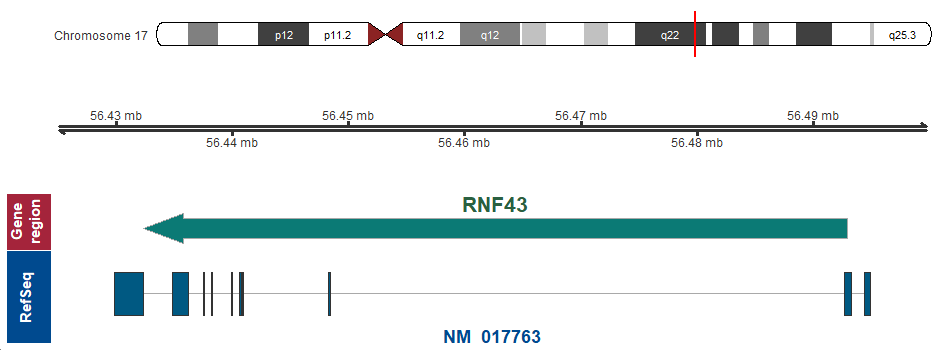
17q22 (chr17:56432304..56492938, complement)
- アミノ酸配列の長さ
-
783
遺伝子マップ
解説
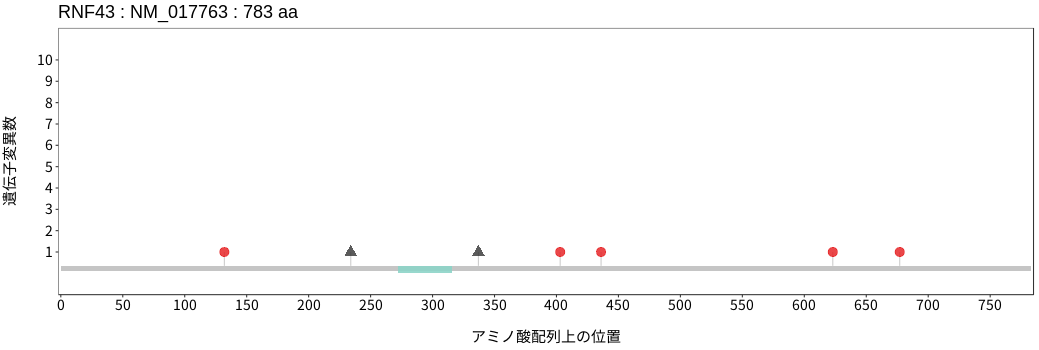
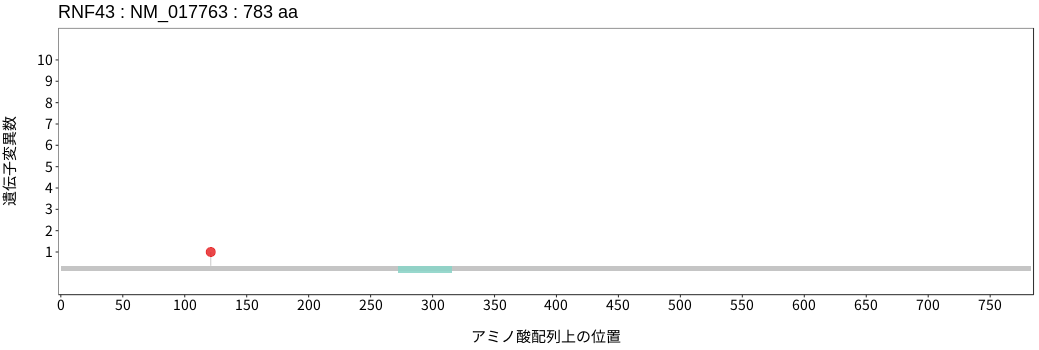
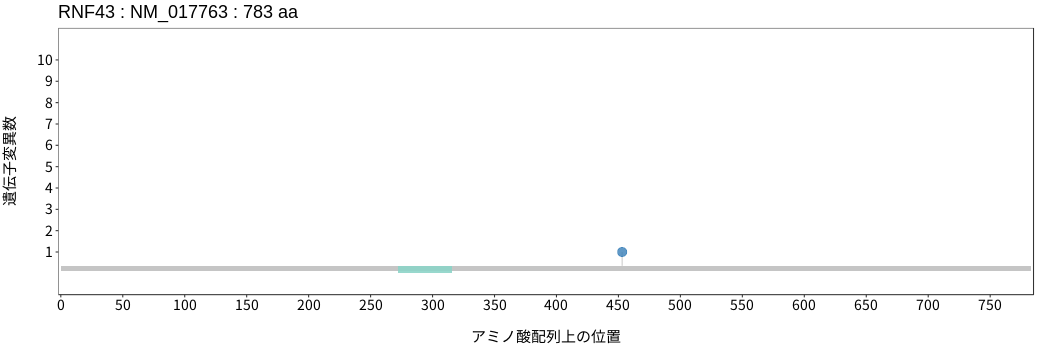
RNF43は、E3ユビキチンリガーゼである。WNTシグナルの活性化因子であるR-spondinタンパク非存在下において、同じくE3ユビキチンリガーゼであるZNRF3と共にWNTリガンドの受容体であるFrizzled (FZD)をユビキチン化することで分解誘導し、WNTシグナルの抑制因子として機能する。結腸直腸癌および子宮内膜癌において変異が高い頻度で検出される。
シグナル伝達経路

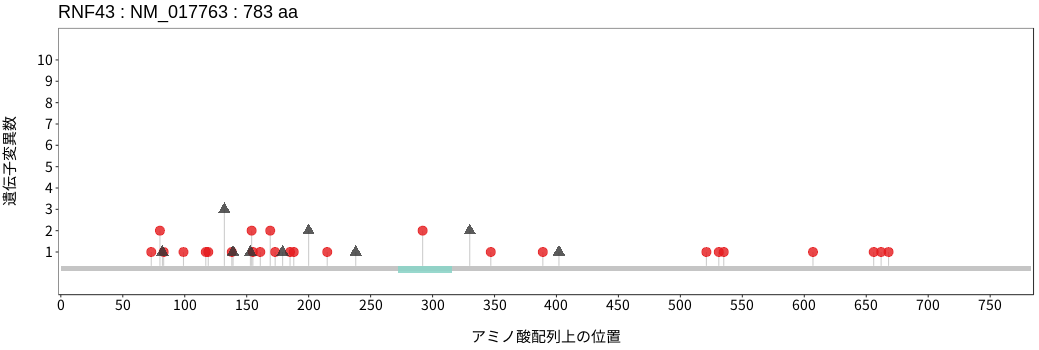
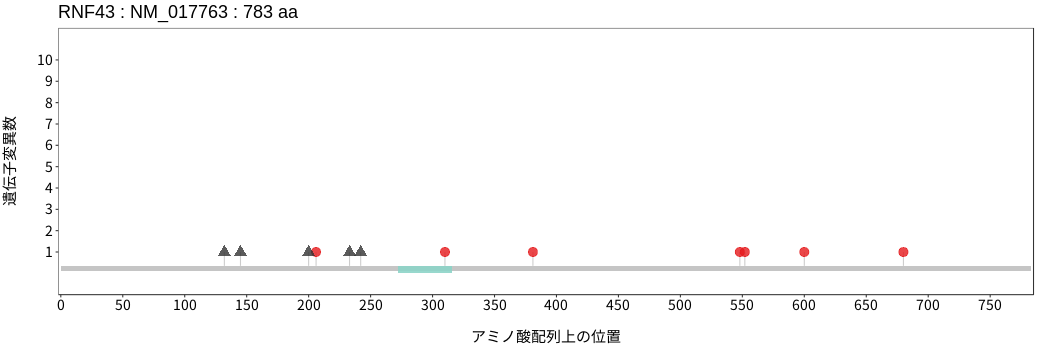
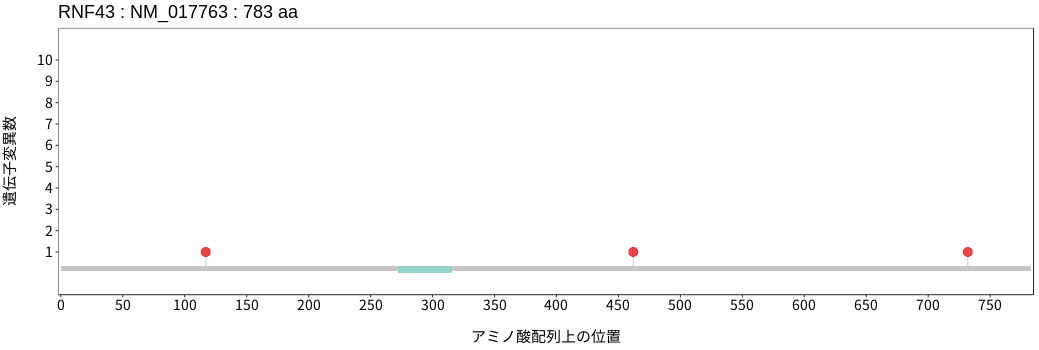
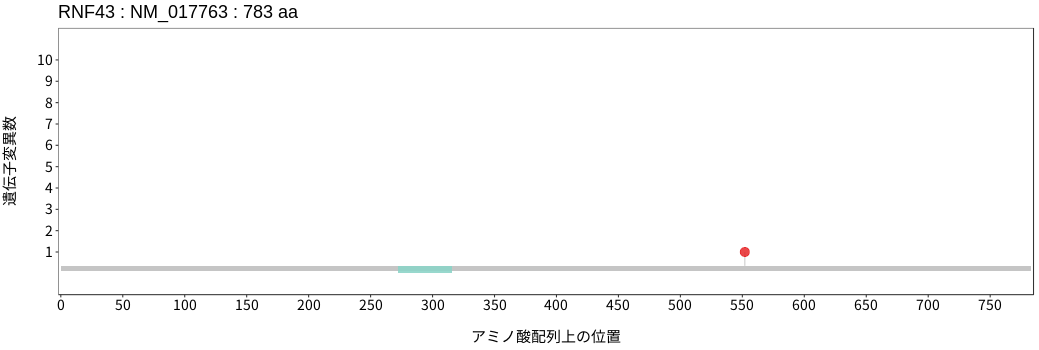
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

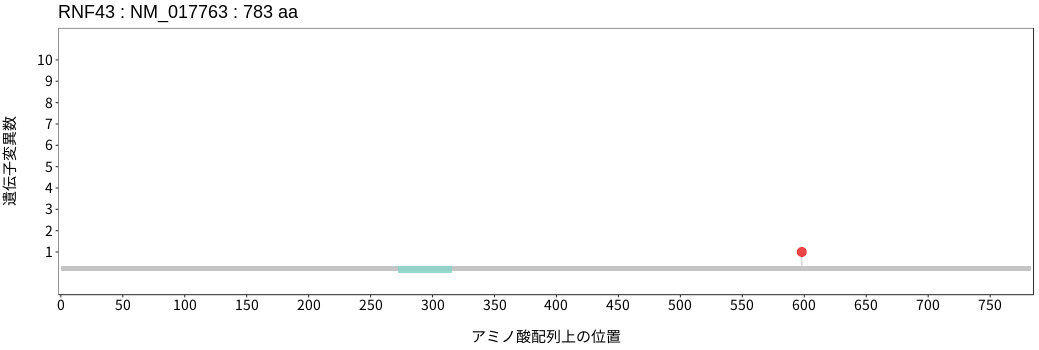
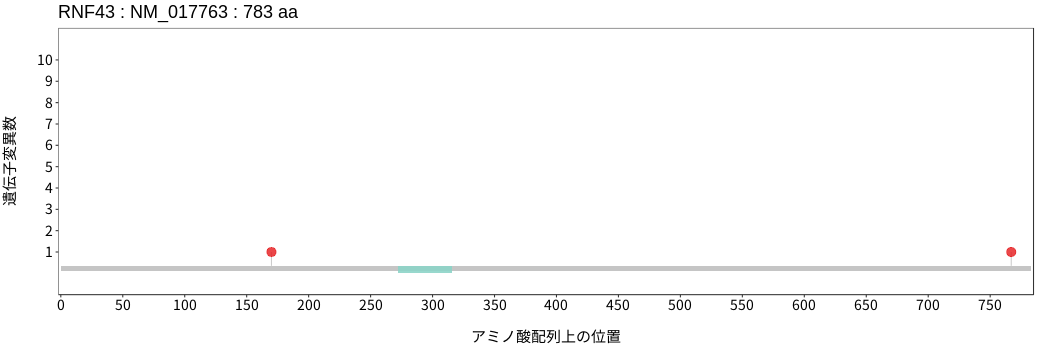
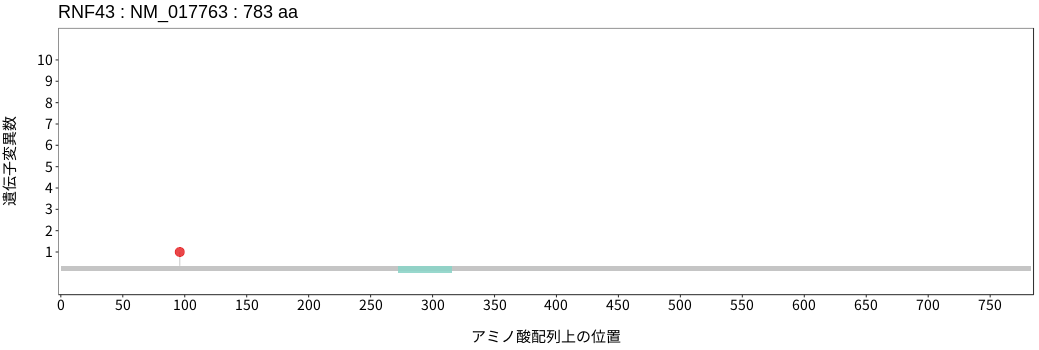
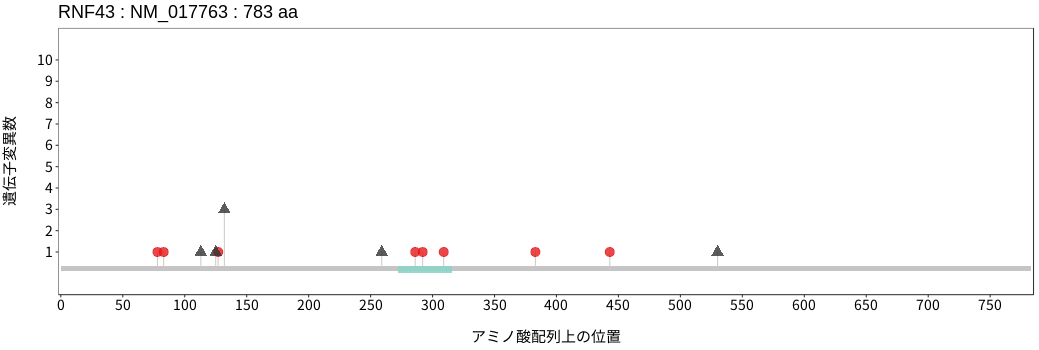
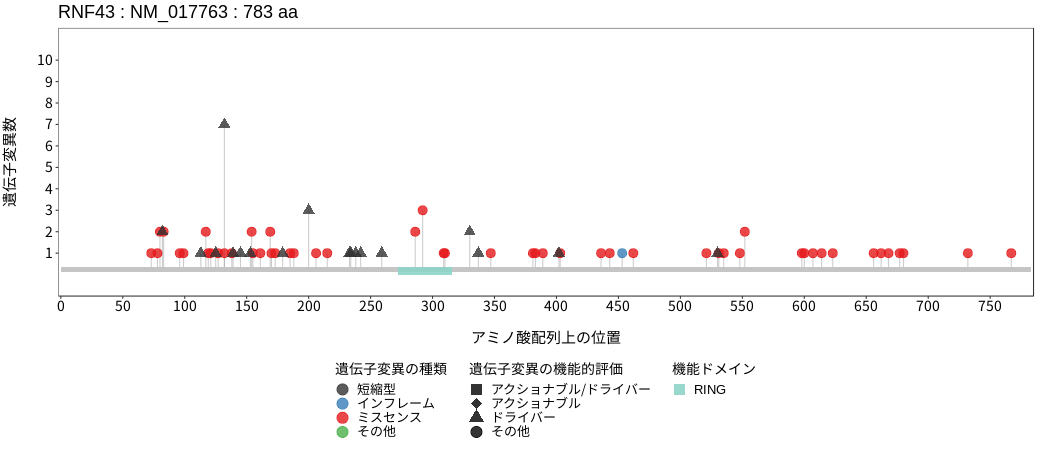
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr17:56492693 | TA | T | 2 / 10 | p.L82fs | c.245delT | Tier2 | 1 / 102 | |
| chr17:56492694 | A | C | 2 / 10 | p.L82* | c.245T>G | COSV60416856 | Tier2 | 1 / 102 |
| chr17:56448274 | T | TGCTA | 3 / 10 | p.K125fs | c.369_372dupTAGC | Tier2 | 1 / 102 | |
| chr17:56448310 | G | A | 3 / 10 | p.R113* | c.337C>T | COSV68457279 | Tier2 | 1 / 102 |
| chr17:56440904 | G | A | 4 / 10 | p.R145* | c.433C>T | COSV68456834 | Tier2 | 1 / 102 |
| chr17:56440919 | CAA | C | 4 / 10 | p.F139fs | c.416_417delTT | Tier2 | 1 / 102 | |
| chr17:56440943 | G | A | 4 / 10 | p.R132* | c.394C>T | COSV68457376 | Tier2 | 7 / 102 |
| chr17:56440676 | TTTTGGTTC | T | 5 / 10 | p.N179fs | c.534_541delGAACCAAA | Tier2 | 1 / 102 | |
| chr17:56440761 | G | A | 5 / 10 | p.Q153* | c.457C>T | COSV68459258 | Tier2 | 1 / 102 |
| chr17:56439992 | C | T | 6 / 10 | p.W200* | c.600G>A | COSV68457592 | Tier2 | 1 / 102 |
| chr17:56439993 | C | T | 6 / 10 | p.W200* | c.599G>A | COSV68459246 | Tier2 | 2 / 102 |
| chr17:56438216 | C | T | 7 / 10 | p.W259* | c.777G>A | COSV68457166 | Tier2 | 1 / 102 |
| chr17:56438269 | G | A | 7 / 10 | p.Q242* | c.724C>T | COSV68459333 | Tier2 | 1 / 102 |
| chr17:56438280 | C | T | 7 / 10 | p.W238* | c.713G>A | COSV104435047 | Tier2 | 1 / 102 |
| chr17:56438293 | G | A | 7 / 10 | p.Q234* | c.700C>T | Tier2 | 1 / 102 | |
| chr17:56438296 | G | A | 7 / 10 | p.Q233* | c.697C>T | COSV68459713 | Tier2 | 1 / 102 |
| chr17:56435536 | TCCAAGGAACGAGG | T | 9 / 10 | p.P530fs | c.1588_1600delCCTCGTTCCTTGG | Tier2 | 1 / 102 | |
| chr17:56435933 | G | A | 9 / 10 | p.Q402* | c.1204C>T | Tier2 | 1 / 102 | |
| chr17:56436128 | G | A | 9 / 10 | p.R337* | c.1009C>T | COSV68456791 | Tier2 | 1 / 102 |
| chr17:56436149 | G | A | 9 / 10 | p.R330* | c.988C>T | COSV68457287 | Tier2 | 2 / 102 |