Side Menu
SETD2
基本情報
- 遺伝子名
-
SET domain containing 2, histone lysine methyltransferase
- 慣用名
-
HBP231, HIF-1, HIP-1, HSPC069, HYPB, KMT3A, LLS, SET2, p231HBP
- 遺伝子分類
-
がん抑制遺伝子
- 遺伝子ID
- 転写産物ID
- 機能分類
-
エピジェネティック制御
- シグナル伝達経路
-
Epigenetic modification
- 染色体上の位置 (GRCh37/hg19)
-
3p21.31 (chr3:47058583..47205414, complement)
- アミノ酸配列の長さ
-
2,564
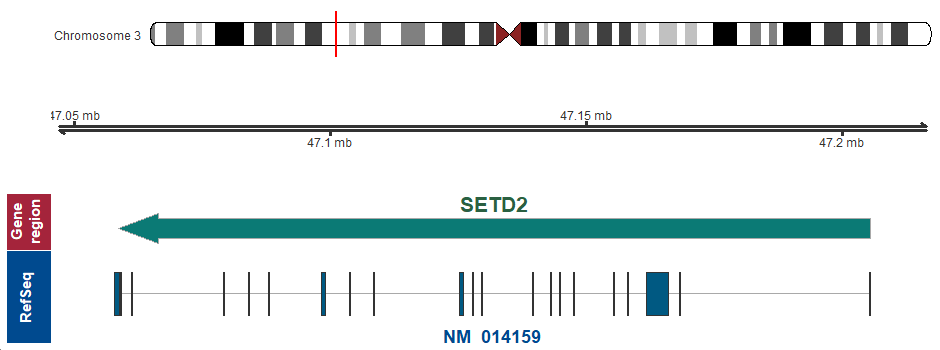
遺伝子マップ
解説
SETD2は、ヒストンH3の36番目のリジン残基 (H3K36)特異的なトリメチル化に働くリジンメチル基転移酵素であり、転写調節に関与する。またSETD2によるH3K36のトリメチル化は、DNA二本鎖切断が起きた際の相同組み換え修復の初期反応に必要なLEDGFをクロマチンへ動員することで、修復反応の促進に関与する。
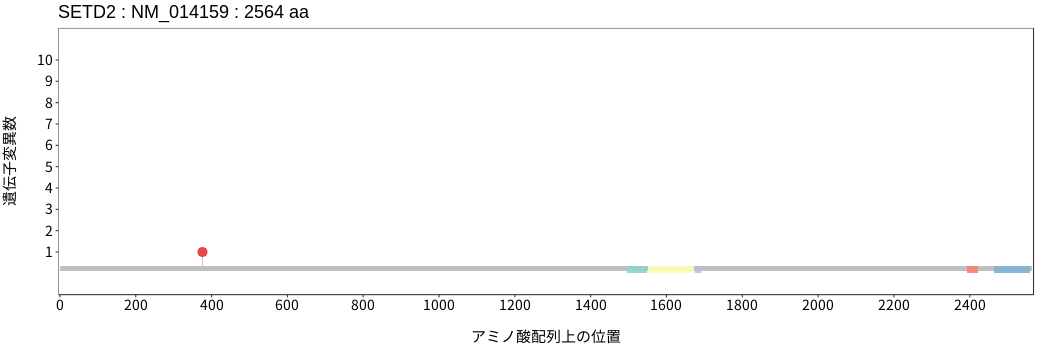
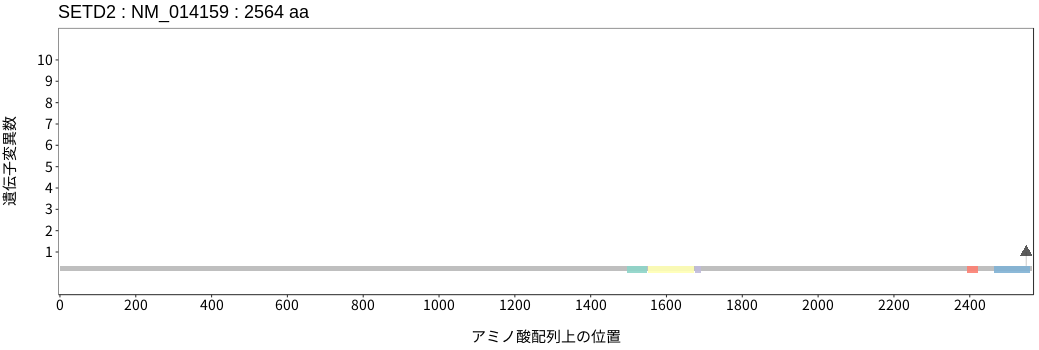
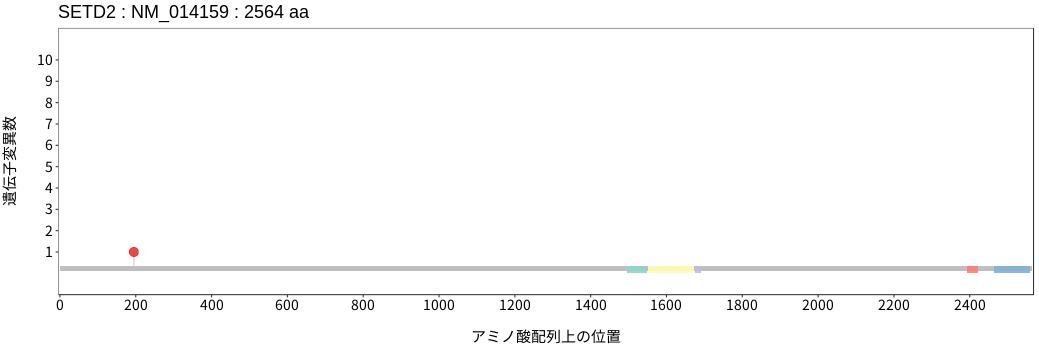
遺伝子変異総量(Tumor Mutation Burden)の分布TMB

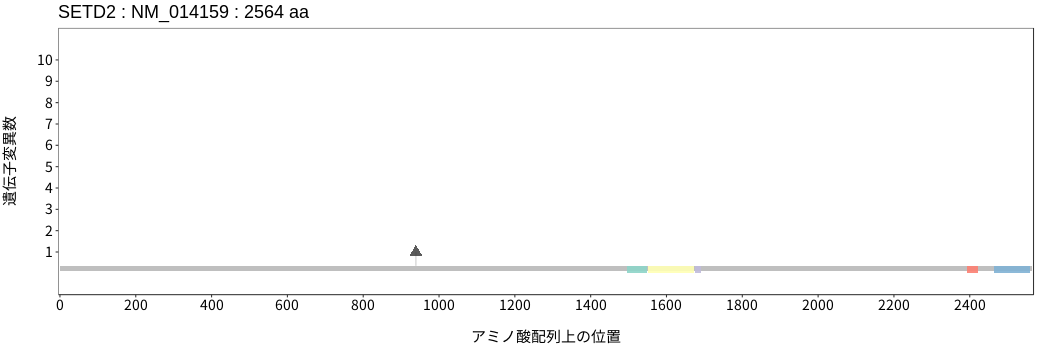
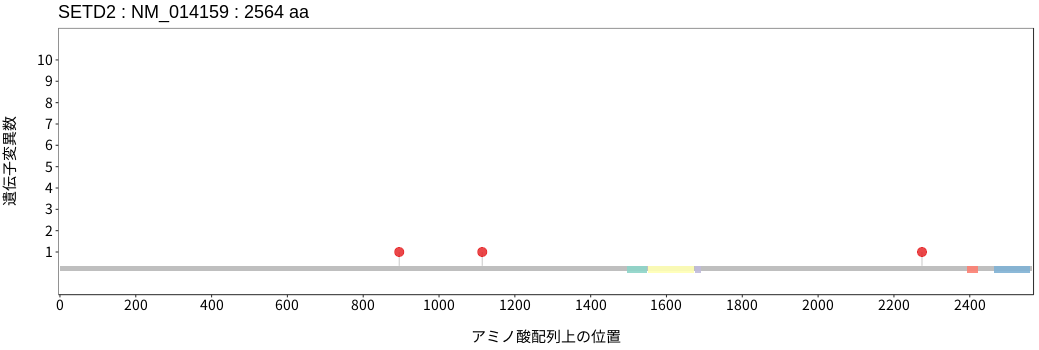
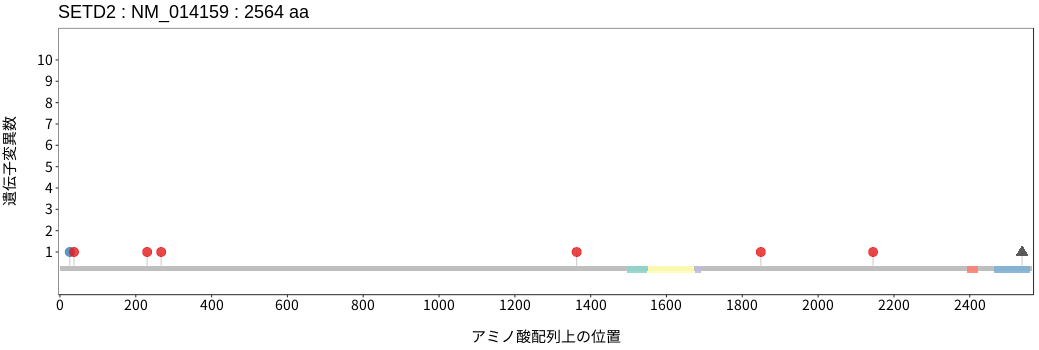
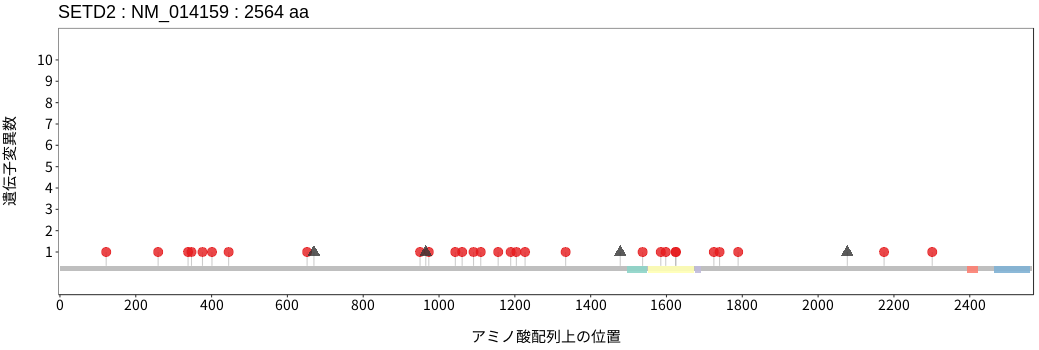
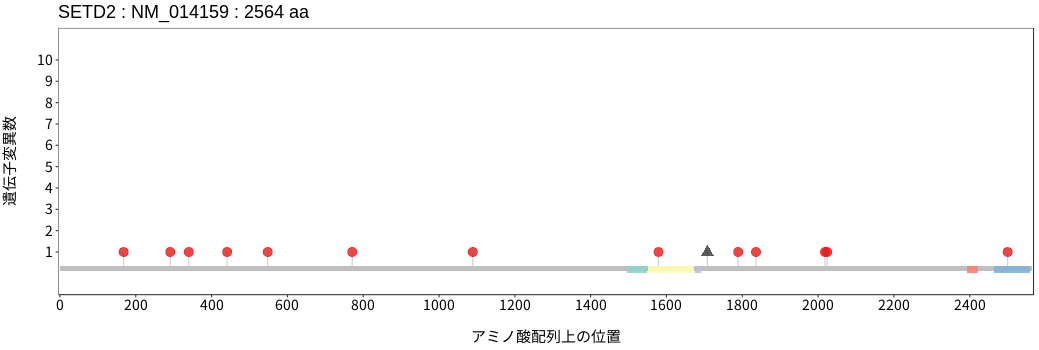
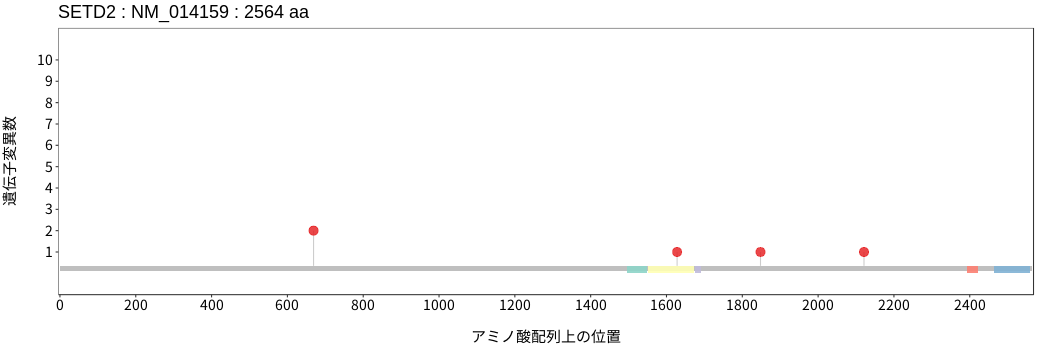
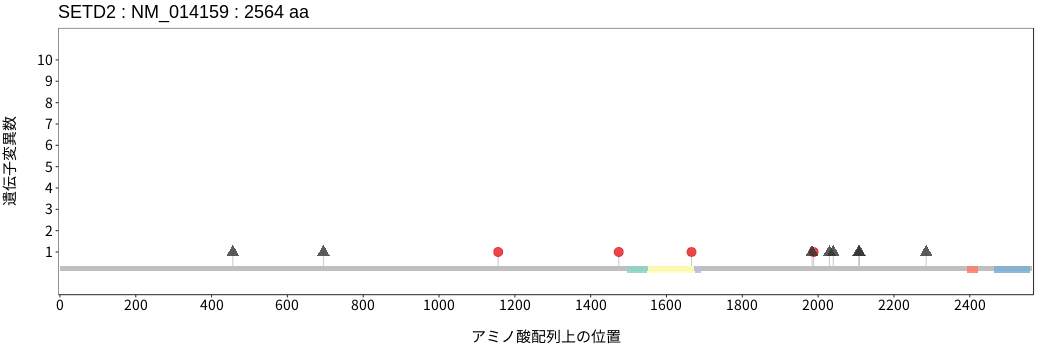
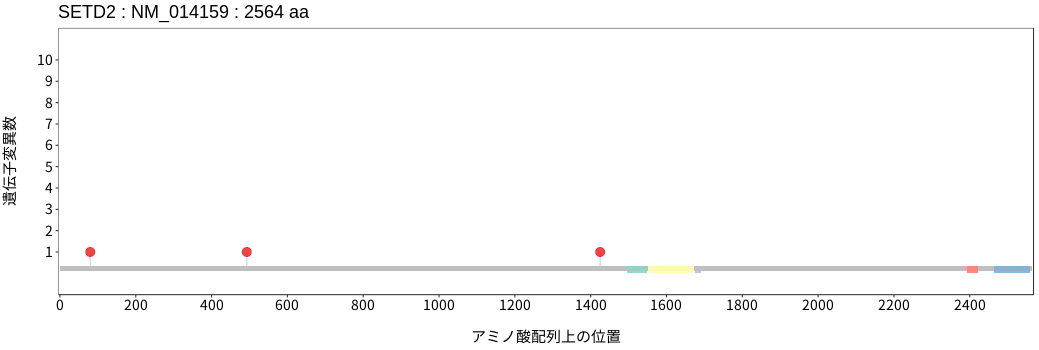
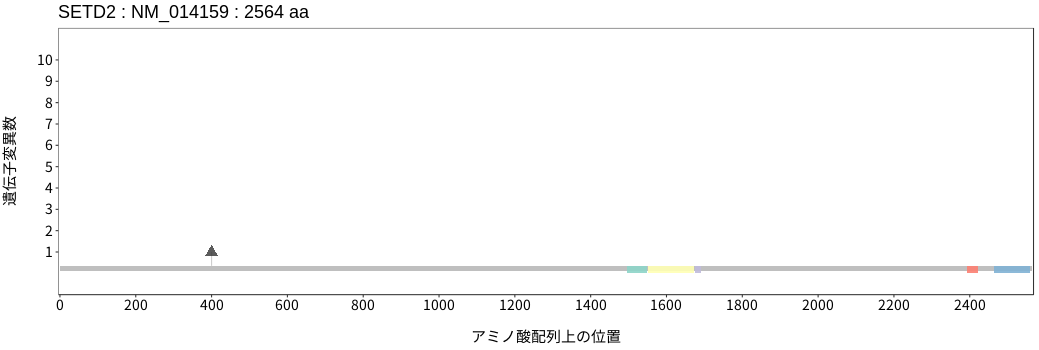
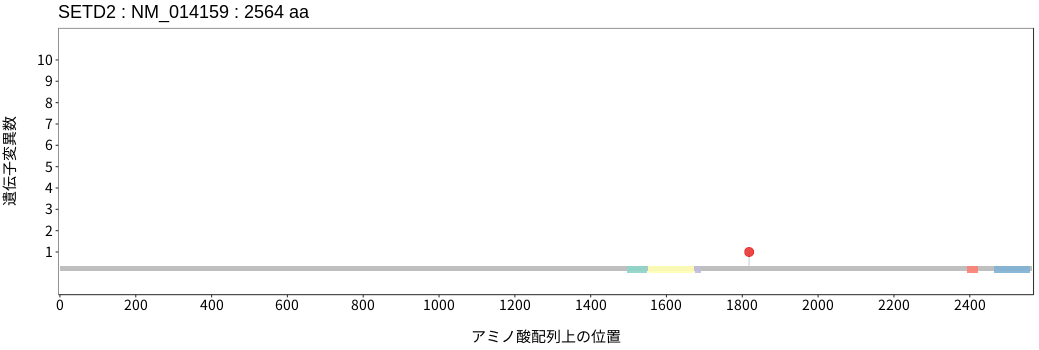
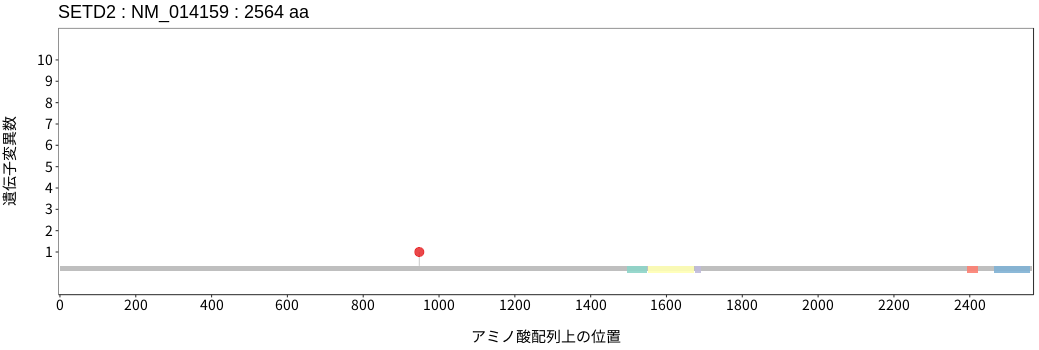
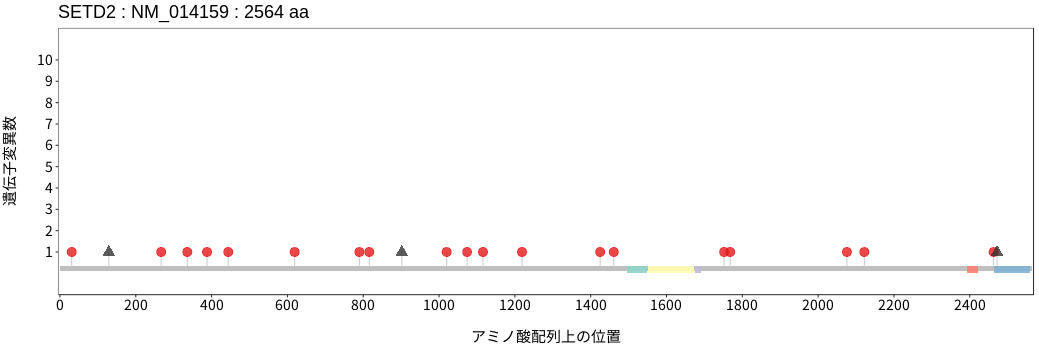
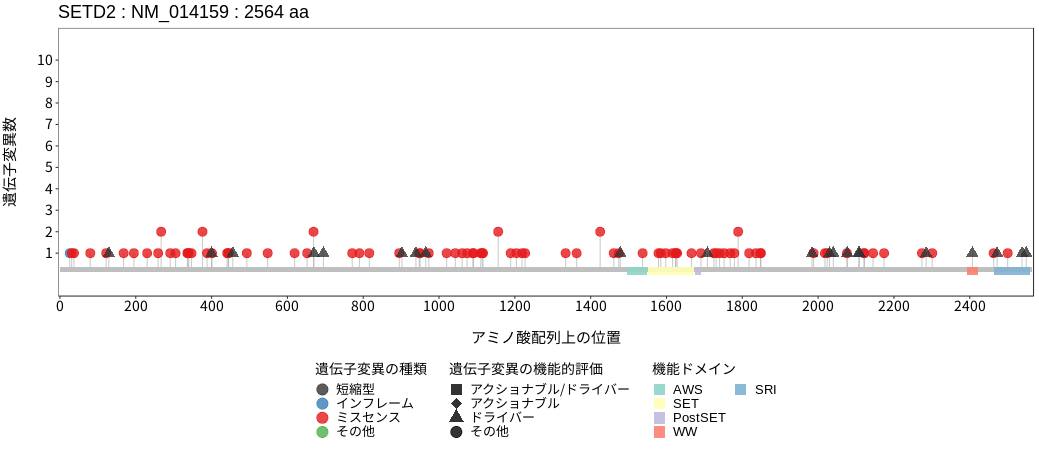
アミノ酸配列上の変異分布
- 低悪性度
神経膠腫 - 多形膠芽腫
- 髄膜腫
- 頭頸部
扁平上皮癌 - 唾液腺癌
- 食道扁平上皮癌
- 胃腺癌
- 小腸癌
- 結腸腺癌
- 直腸腺癌
- 肝細胞癌
- 胆管癌
- 膵腺癌
- 肺腺癌
- 肺扁平上皮癌
- 肺
神経内分泌腫瘍 - 胸膜中皮腫
- 胸腺腫
- 浸潤性乳管癌
- 浸潤性小葉癌
- 化生癌
- 骨肉腫
- 消化管間質腫瘍
- 軟部肉腫
- 子宮頸癌
- 子宮類内膜腺癌
- 上皮性卵巣癌
- 淡明細胞型
腎細胞癌 - 悪性黒色腫
- 有棘細胞癌
ドライバー変異リスト
| 参照配列上の 位置 (GRCh37/hg19) |
参照配列の塩基 | 変異の塩基 | 変異が位置するエクソン番号 | 変異 (アミノ酸 変化) |
変異 (CDS 変化) |
COSMIC登録ID (v92) | がん化との 関連度による 分類 |
変異が検出されたサンプル数 |
|---|---|---|---|---|---|---|---|---|
| chr3:47161694 | C | A | 3 / 21 | p.E1478* | c.4432G>T | COSV57436969 | Tier2 | 1 / 120 |
| chr3:47163233 | C | A | 3 / 21 | p.E965* | c.2893G>T | COSV57457123 | Tier2 | 1 / 120 |
| chr3:47163310 | G | T | 3 / 21 | p.S939* | c.2816C>A | Tier2 | 1 / 120 | |
| chr3:47163422 | C | A | 3 / 21 | p.E902* | c.2704G>T | Tier2 | 1 / 120 | |
| chr3:47164042 | A | C | 3 / 21 | p.L695* | c.2084T>G | Tier2 | 1 / 120 | |
| chr3:47164118 | C | A | 3 / 21 | p.E670* | c.2008G>T | COSV57446526 | Tier2 | 1 / 120 |
| chr3:47164760 | G | A | 3 / 21 | p.R456* | c.1366C>T | Tier2 | 1 / 120 | |
| chr3:47164928 | G | A | 3 / 21 | p.R400* | c.1198C>T | COSV57443300 | Tier2 | 1 / 120 |
| chr3:47165741 | C | A | 3 / 21 | p.E129* | c.385G>T | Tier2 | 1 / 120 | |
| chr3:47139465 | G | A | 9 / 21 | p.R1708* | c.5122C>T | COSV57449337 | Tier2 | 1 / 120 |
| chr3:47125317 | CCT | C | 12 / 21 | p.E1984fs | c.5951_5952delAG | Tier2 | 1 / 120 | |
| chr3:47108581 | G | A | 13 / 21 | p.Q2030* | c.6088C>T | Tier2 | 1 / 120 | |
| chr3:47103717 | G | A | 14 / 21 | p.R2077* | c.6229C>T | COSV57434124 | Tier2 | 1 / 120 |
| chr3:47103828 | G | A | 14 / 21 | p.R2040* | c.6118C>T | COSV57434358 | Tier2 | 1 / 120 |
| chr3:47098421 | G | A | 15 / 21 | p.Q2285* | c.6853C>T | Tier2 | 1 / 120 | |
| chr3:47098949 | G | A | 15 / 21 | p.R2109* | c.6325C>T | COSV57430221 | Tier2 | 1 / 120 |
| chr3:47098955 | T | A | 15 / 21 | p.K2107* | c.6319A>T | Tier2 | 1 / 120 | |
| chr3:47084068 | G | T | 17 / 21 | p.Y2407* | c.7221C>A | Tier2 | 1 / 120 | |
| chr3:47061267 | C | A | 19 / 21 | p.E2472* | c.7414G>T | Tier2 | 1 / 120 | |
| chr3:47058633 | G | A | 21 / 21 | p.Q2549* | c.7645C>T | Tier2 | 1 / 120 | |
| chr3:47058662 | TTG | T | 21 / 21 | p.H2538fs | c.7614_7615delCA | Tier2 | 1 / 120 |